机器学习 | K均值聚类(K-means Clustering)
本文从概念、应用场景、原理、工作流程、优缺点、应用实践、代码、可视化等几方面诠释 K 均值聚类模型
概述
K-Means 是一种无监督的聚类算法,其目的是将 n 个数据点分为 k 个聚类。每个聚类都有一个质心,这些质心最小化了其内部数据点与质心之间的距离。
它能做什么
-
市场细分: 识别具有相似属性的潜在客户群体。
-
图像分析: 图像压缩和图像分割中的像素聚类。
-
异常检测: 通过标识不符合标准集群特征的观测结果来检测异常。
-
基因表达数据分析: 对基因进行聚类以识别具有相似表达模式的基因家族。
原理
初始化: 随机选择 k 个数据点作为初始聚类中心,这些可以是数据集中实际存在的点,也可以是随机生成的点。
-
K-Means++: 为了避免随机初始化可能导致的不良结果,K-Means++ 策略通过特别选择远离其他中心的起始中心来优化初始聚类中心的位置。
分配: 遍历样本数据集,计算每个数据点到每个质心的距离,找出数据点距离最近的质心,将数据点分配给该聚类。
-
公式
设 是第 i 个集群, 是第 i 个集群的中心,目标是最小化所有数据点和其所在集群中心之间的平方距离之和:
更新: 计算每个聚类内的平均值,并将平均值设为新的聚类中心。
假设我们有一个聚类 ,其中包含数据点 ,那么该集群的质心 可以使用以下公式计算:
迭代: 重复分配和更新两步,直到质心不在显著移动或达到预定的迭代次数
优缺点
优点
-
简单且直观。
-
对于大数据集,存在可扩展的 K-Means 变种。
-
常被用作预处理步骤或数据分析。
缺点
-
需要预先指定 K(聚类数量),但 K 的最佳值不总是明确的。
-
对初始聚类中心的选择敏感,可能导致局部最优解。
-
可能不适用于非球形的聚类或大小差异很大的聚类。
-
在大规模数据集上收敛速度慢
-
适用于数值型数据类型
选择正确的 K 值
-
K 的选择是个挑战,因为它是预先设定的,而实际的数据集群数量可能是未知的。一种常用的方法是使用肘部法则(Elbow Method)来确定最优的 K 值。
局部最优解问题
-
K-Means 容易陷入局部最优解,这是因为算法的结果受初始聚类中心的选择影响。解决方案包括多次运行算法,每次用不同的初始聚类中心,或使用全局优化算法。
处理不同大小和密度的集群
-
K-Means 假设所有集群在形状和大小上都是相似的。对于不同大小或密度的集群,算法可能无法有效地划分数据。在这些情况下,可能需要考虑使用更高级的聚类技术,如 DBSCAN 或谱聚类。
Python 实现
加载数据
import pandas as pd
from sklearn.datasets import load_iris
# 加载数据集
data = load_iris()
df = pd.DataFrame(data.data, columns=data.feature_names)
数据探索(EDA)
import seaborn as sns
import matplotlib.pyplot as plt
# 打印数据的头部和描述性统计
print(df.head())
print(df.describe())
# 使用pairplot查看数据的分布和特征间的关系
sns.pairplot(df)
plt.show()
# 检查数据中是否有缺失值
print(df.isnull().sum())
-
数据分布与特征间关系
图中petal length和petal width之间存在明显的线性关系,并且可以看到至少两个明显的群集。具体的数值会因数据集而异,但可以观察到这些聚类的大致范围和关系。
数据预处理
from sklearn.preprocessing import StandardScaler
# 使用标准化器标准化数据(由于KMeans对特征的尺度敏感)
scaler = StandardScaler()
df_scaled = scaler.fit_transform(df)
df_scaled = pd.DataFrame(df_scaled, columns=df.columns)
特征工程
-
用PCA对特征降维
from sklearn.decomposition import PCA
# 使用PCA进行特征降维
pca = PCA(n_components=2)
data_2d = pca.fit_transform(df_scaled)
df_pca = pd.DataFrame(data_2d, columns=['PC1', 'PC2'])
模型训练
from sklearn.cluster import KMeans
# 进行K-Means聚类
kmeans = KMeans(n_clusters=3)
df_pca['cluster'] = kmeans.fit_predict(df_scaled)
可视化结果
# 使用散点图可视化PCA后的数据
plt.figure(figsize=(12, 6))
plt.scatter(df_pca['PC1'], df_pca['PC2'], c=df_pca['cluster'], cmap='viridis', s=50)
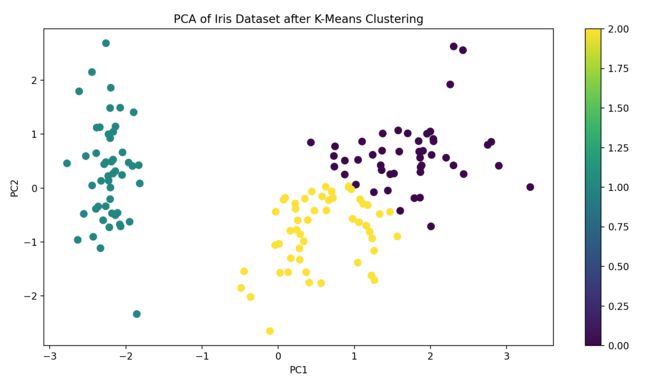
plt.title('PCA of Iris Dataset after K-Means Clustering')
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.colorbar()
plt.show()
在此图中,你会看到三个明显的聚类(如果选择了k=3)。每个聚类的中心点可以通过查看KMeans的cluster_centers_属性来确定。这些中心点在PCA降维的2D空间中给出了聚类的中心位置。
# 使用并行坐标轴进行可视化
plt.figure(figsize=(12, 6))
pd.plotting.parallel_coordinates(df.assign(cluster=df_pca['cluster']), 'cluster', colormap='viridis')
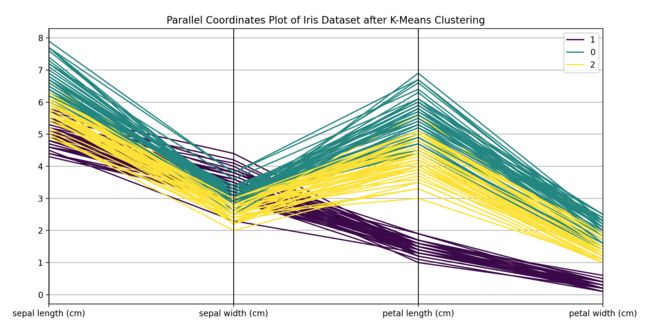
plt.title('Parallel Coordinates Plot of Iris Dataset after K-Means Clustering')
plt.show()
在此图中,你会注意到不同颜色的线表示不同的聚类。如果某个特征对于某个群集有显著的值,你会在该特征上看到这个群集的线与其他线有明显的分离。
模型评估
from sklearn.metrics import silhouette_score
# 计算不同K值的WCSS来选择最佳K值
wcss = []
k_values = range(1, 11)
for k in k_values:
kmeans = KMeans(n_clusters=k, random_state=42)
kmeans.fit(df_scaled)
wcss.append(kmeans.inertia_)
plt.figure(figsize=(12, 6))
plt.plot(k_values, wcss, '-o')
plt.title('The Elbow Method')
plt.xlabel('Number of clusters (K)')
plt.ylabel('WCSS')
plt.show()
WCSS的值会随着K的增加而减少。在"肘点",WCSS的下降速度会突然变慢。例如,如果在k=3时WCSS从400降到250,而在k=4时从250降到240,那么k=3可能是一个好的选择,因为增加更多的群集并没有显著降低WCSS。
# 使用轮廓系数评估聚类效果
k_values = range(2, 10)
silhouette_scores = []
for k in k_values:
kmeans = KMeans(n_clusters=k, random_state=42).fit(df_scaled)
labels = kmeans.labels_
score = silhouette_score(df_scaled, labels)
silhouette_scores.append(score)
plt.figure(figsize=(10, 6))
plt.plot(k_values, silhouette_scores, marker='o')
plt.xlabel('Number of clusters')
plt.ylabel('Silhouette Score')
plt.title('Silhouette Score vs. Number of Clusters')
plt.grid(True)
plt.show()
对于k=2到k=10的每个值,都会有一个轮廓系数。数值越接近1,表示聚类效果越好。你可以找到具有最高轮廓系数的k值。
# 显示每个群集的特征均值
cluster_means = df.assign(cluster=df_pca['cluster']).groupby('cluster').mean()
plt.figure(figsize=(10, 6))
sns.heatmap(cluster_means.T, cmap='coolwarm', annot=True)
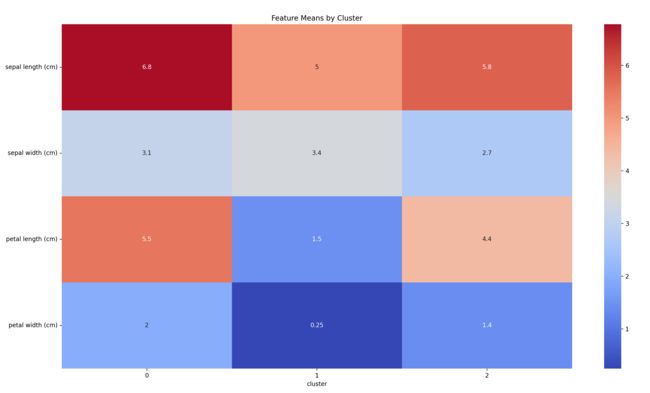
plt.title('Feature Means by Cluster')
plt.show()
图中的数值显示了每个特征在每个群集中的平均值。例如,如果群集1的petal length均值为1.5,而群集2为4.4,那么这说明petal length是一个对于这两个群集有区分性的特征。
关注公众号每日思行回复:KMC代码 获取完整版源码