研究论文 2022-Oncoimmunology:AI+癌RNA-seq数据 识别细胞景观
Wang, Xin, et al. "Deep learning using bulk RNA-seq data expands cell landscape identification in tumor microenvironment." Oncoimmunology 11.1 (2022): 2043662. https://www.tandfonline.com/doi/full/10.1080/2162402X.2022.2043662
被引次数:5
2022年分区:大类医学2区;小类免疫学2区,肿瘤学 3区
IF 7.723 JCR Q1
一、数据集
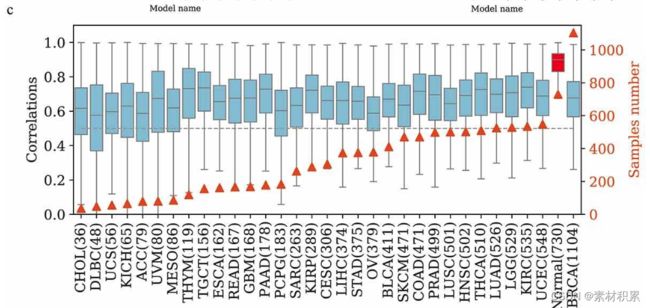
从GDC数据库(https://portal.gdc.cancer.gov/projects)下载32种“癌症样本和正常样本”,共有10906患者的表达谱和临床数据。
非小细胞肺癌数据的两种亚型:肺腺癌(LUAD)和肺鳞状细胞癌(LUSC)
从GEO数据集(https://www.ncbi.nlm.nih.gov/geo/)下载5个数据集:
-
GSE81861(CRC):11例结直肠癌患者的样本,包含7种细胞类型。引用文献32
-
GSE75688(BC):11个乳腺癌细胞和淋巴结转移的单细胞测序数据。引用文献33。包括515个单细胞RNA测序数据,使用Illumina HiSeq 2500进行测序。基因表达水平以TPM(每百万转录本)表示,已经进行了标准化,可以在下一步的分析中进行比较。
-
GSE86146(FGC):2167个个体生殖细胞及其生殖腔细胞,涵盖了从怀孕后4周到26周的女性和男性人类胚胎的发育阶段。引用文献21。
-
GSE72056(Melanoma):31个黑色素瘤样本及其6种细胞类型。引用文献34。
-
GSE78220(PD1):38个黑色素瘤活检样本在抗PD-1治疗之前的转录组样本和相应的临床数据。引用文献35。
-
细胞类型及其对应的标记基因来自CellMarker数据库(http://bio-bigdata.hrbmu.edu.cn/CellMarker/)。
CellMarker数据预处理
从CellMarker数据库中下载了来自158个人类组织中的467种细胞类型的13,605个标记基因。
数据处理如下:由于一些细胞及其标记基因在不同人类组织中重复出现,我们删除了组织特异性的重复,仅保留一个细胞及其标记基因作为重复组的代表。为了DCNet模型的训练目的,删除了在TCGA基因集中未检测到的标记基因[详见TCGA数据预处理],然后排除了33个细胞类型,因为未检测到任何标记基因。最终,保留了434个细胞类型,包括免疫细胞、癌细胞、基质细胞等,以及它们对应的9078个标记基因,用于进一步的分析。通过整合CellMarker数据库(http://biocc.hrbmu.edu.cn/CellMarker/)和Cell Ontology数据库(OBO:http://www.obofoundry.org/ontology/cl.html)中的类别信息,计算了主要细胞类型的细胞丰度。在DCNet识别的434个细胞类型中,有77个细胞类型没有Cell Ontology ID,137个细胞类型没有自己的Cell Ontology ID,但被分配到其父类的术语ID,220个细胞类型在OBO数据库中有自己的Cell Ontology ID。对于没有Cell Ontology ID或仅有其父类Cell Ontology ID的细胞类型,从CellMarker数据库中获取了细胞类型之间的层次关系信息。对于具有自己Cell Ontology ID的细胞类型,从Cell OBO数据库中下载了细胞类型之间的本体结构信息。整合这些信息,创建了附表1,其中包含了父细胞类型和子细胞类型的名称、细胞本体ID的父细胞类型和子细胞类型、数据源数据库(CellMarker或OBO)。
对于CellMarker数据库,父细胞类型的细胞丰度是通过累加其子细胞类型的细胞丰度计算的;而对于OBO数据库,父细胞类型的细胞丰度是通过累加其在细胞本体结构中的叶节点的细胞丰度计算的。
TCGA的表达谱数据预处理
对于TCGA的表达谱数据,删除了在超过1/3的样本中表达水平为0的基因,保留了21,136个基因,并进行了对数归一化。我们将每个样本的基因表达分为输入数据(9078个标记基因的表达水平)和输出数据(21,136个基因的表达水平)。由于癌症样本分布不均匀,我们采用了过采样方法来扩大样本数量,同时平衡样本类别。此外,在实验测量过程中,由于某些基因的低表达强度或实验误差,可能无法检测到一些基因,这将导致模型的输入维度与标记基因维度不匹配。为解决这个问题,输入数据以0.1、0.3、0.5的概率被随机删除(0填充),这不仅可以增加训练样本,还可以降低过拟合的风险。最后,所有样本按80%和20%的比例划分为训练集和测试集。
我们还使用TCGA biolink 软件包从TCGA队列中获取了1487名肿瘤转移患者的临床数据和药物治疗信息。如果接受某种药物治疗的患者数量小于48,则该药物的信息将被截断。
二、DCNet神经网络构建与训练
DCNet model trains a deep neural network, which embeds the relationships between cells and their marker genes, to predict more than 400 cell types proportion within bulk seq dataset.
DCNet 模型训练深度神经网络,该网络嵌入细胞及其标记基因之间的关系,以预测批量 seq 数据集中 400 多种细胞类型的比例。
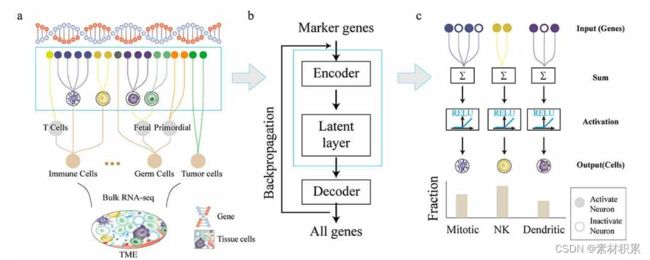
图1. DCNet架构和设计流程。 (a) 组织中不同的细胞类型,如基质细胞、红细胞、巨噬细胞、T细胞、B细胞、神经元细胞等。这些细胞又可进一步分割不同的细胞(b) DCNet模型的基本结构。输入是bulk RNA-seq中标记基因的表达水平,输出是所有基因的表达水平。中间层人工神经网络。(c) 神经灰色DCNet模型的第一级关系是细胞与标记基因的对应关系。DCNet模型的中间层代表细胞的相对内容。 显示全尺寸
三、比较
不同参数量下的NN比较(类似敏感性分析)
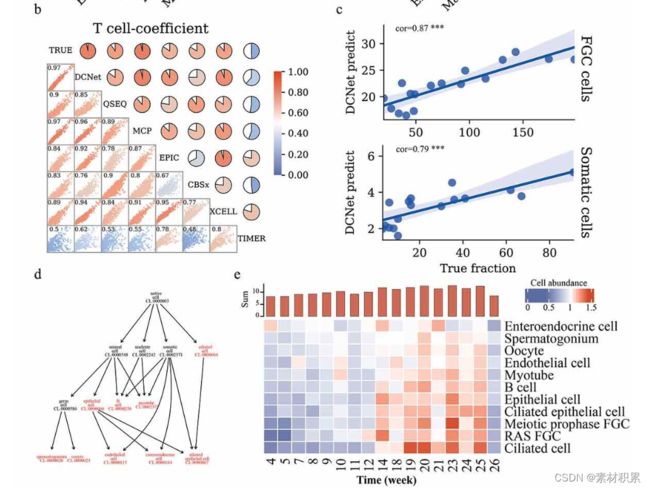
与其他方法比较:CIBERSORTx、EPIC、MCP-Counter、quanTIseq、xCell(默认参数,代码链接见正文,同时R包immunoconv包含了这些方法可用来预测细胞丰度)