【R语言科研绘图】--- 柱状图
R语言科研绘图——柱状图
柱状图
- R语言科研绘图——柱状图
- 前言
- 一、柱状图
- 二、使用工具
-
- 1.所使用的包
- 2.数据特征
- 3.绘图
-
- 3.1 两组均值之间的比较
- 3.2 多组均值之间的比较
- 后记
前言
使用R语言绘制能够发表的图片,ggplot2是一个很好的选择。并且一些基于ggplot2开发的包,能够达到这个目标。graphpad prism做出来的图深得大家喜爱,因此本系列的文章就利用R语言ggprism这个包进绘制
一、柱状图
柱状图在绘图中经常使用到,例如PCR的数据结果,绘制过程如下
二、使用工具
1.所使用的包
为了科研绘图的美观,所以使用了ggprism这个包的绘制,能够绘制出和graphpad prism类似的效果,便于放在论文中发表
2.数据特征
使用的是R自带的ToothGrowth数据,包含了三个数据
len是指牙生长的长度
supp factor指的是给予的干扰因素,其中包括了VC和橘子汁
dose 指的是给予干扰因素的剂量
3.绘图
3.1 两组均值之间的比较
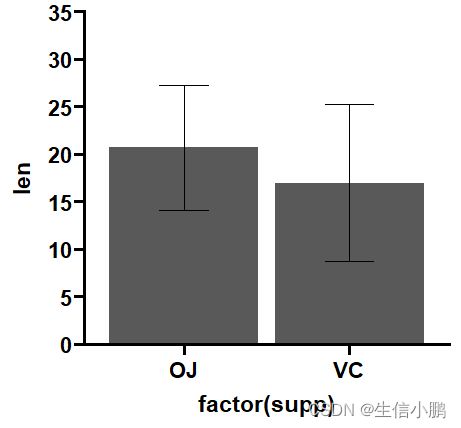
先把柱状图画出来
df_p_val <- rstatix::t_test(ToothGrowth, len ~ supp) %>%
rstatix::add_x_position()
p <- ggplot(ToothGrowth, aes(x = factor(supp), y = len)) +
stat_summary(geom = "col", fun = mean) +
stat_summary(geom = "errorbar",
fun = mean,
fun.min = function(x) mean(x) - sd(x),
fun.max = function(x) mean(x) + sd(x),
width = 0.3) +
theme_prism() +
coord_cartesian(ylim = c(0, 35)) +
scale_y_continuous(breaks = seq(0, 35, 5), expand = c(0, 0))
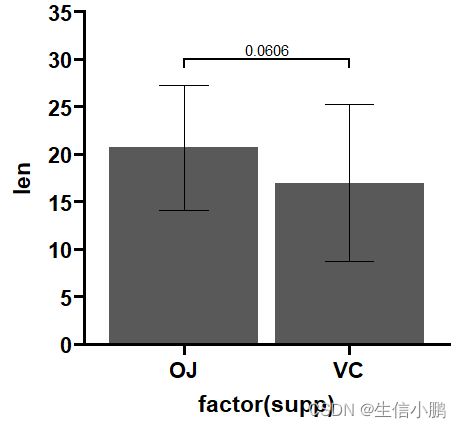
加上统计结果
这里使用rstatix这个包里面的函数进行计算。
rstatix这个包的特点是能够和tidyverse类似,能够使用管道符进行计算,便于和tidyverse进行对接
df_p_val <- rstatix::t_test(ToothGrowth, len ~ supp) %>%
rstatix::add_x_position()
p + add_pvalue(df_p_val, y.position = 30)
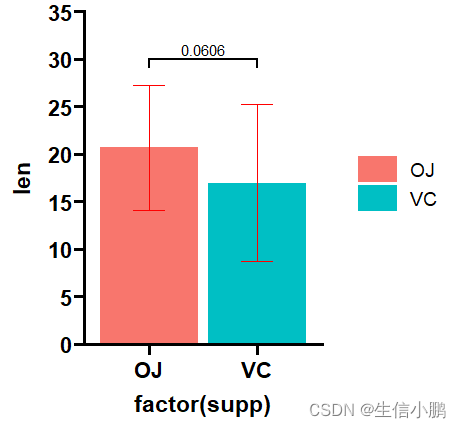
缺少颜色,加上颜色
p <- ggplot(ToothGrowth,
aes(x = factor(supp), y = len)
) +
stat_summary(geom = "col", aes(fill = factor(supp)),fun = mean) +
stat_summary(geom = "errorbar", colour = 'red',
fun = mean,
fun.min = function(x) mean(x) - sd(x),
fun.max = function(x) mean(x) + sd(x),
width = 0.3) +
theme_prism() +
coord_cartesian(ylim = c(0, 35)) +
scale_y_continuous(breaks = seq(0, 35, 5), expand = c(0, 0))
p
p+ add_pvalue(df_p_val, y.position = 30)
需要注意的是,在添加颜色的时候,是在
stat_summary当中添加aes(fill = factor(supp),如果添加到ggplot函数中,后面的统计结果又加不上去。
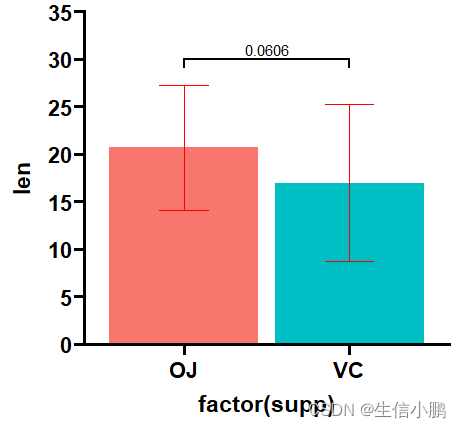
去除图例
因为图片的下方已经有了内容,反而图例显得多余,所以就去除掉图例。使用的是ggplot2绘制,因此按如下方法即可:
p + theme(legend.position = 'none')+
add_pvalue(df_p_val, y.position = 30)
3.2 多组均值之间的比较
依然使用ToothGrowth 这个数据,比较不同的剂量之间生长的差异。总共有三组不同的剂量,这属于多组比较,这就需要确定一个比照的参照系。
p <- ToothGrowth %>%
ggplot(aes(x = factor(dose), y = len)) +
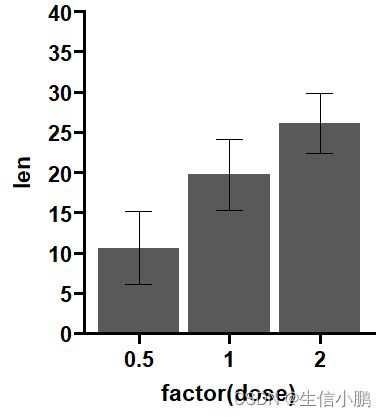
stat_summary(geom = "col", fun = mean) +
stat_summary(geom = "errorbar",
fun = mean,
fun.min = function(x) mean(x) - sd(x),
fun.max = function(x) mean(x) + sd(x),
width = 0.3) +
theme_prism() +
coord_cartesian(ylim = c(0, 40)) +
scale_y_continuous(breaks = seq(0, 40, 5), expand = c(0, 0))
p
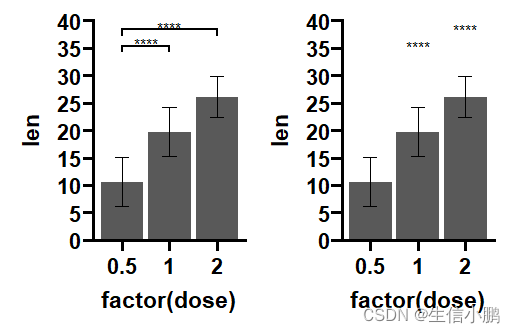
df_p_val <-
rstatix::t_test(ToothGrowth, len ~ dose, ref.group = "0.5") %>%
rstatix::add_xy_position()
p1 <- p + add_pvalue(df_p_val, label = "p.adj.signif")
p2 <- p + add_pvalue(df_p_val, label = "p.adj.signif", remove.bracket = TRUE)
p <- ToothGrowth %>%
ggplot(aes(x = factor(dose), y = len)) +
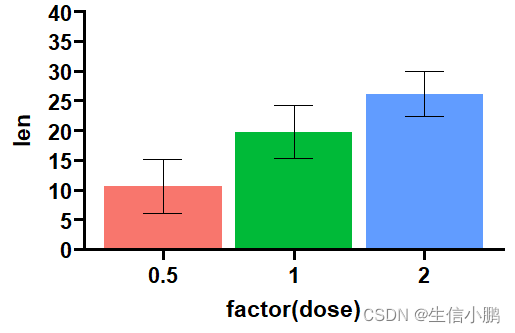
stat_summary(geom = "col", fun = mean,aes(fill = factor(dose))) +
stat_summary(geom = "errorbar",
fun = mean,
fun.min = function(x) mean(x) - sd(x),
fun.max = function(x) mean(x) + sd(x),
width = 0.3) +
theme_prism() +
coord_cartesian(ylim = c(0, 40)) +
scale_y_continuous(breaks = seq(0, 40, 5), expand = c(0, 0))
p + theme(legend.position = 'none')
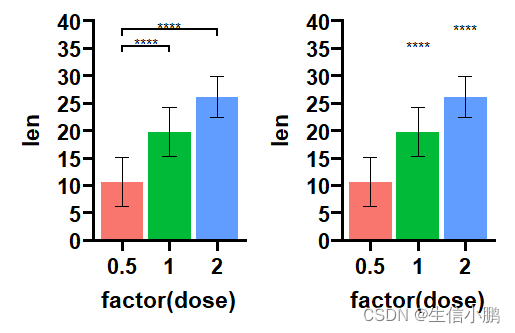
df_p_val <- rstatix::t_test(ToothGrowth, len ~ dose, ref.group = "0.5") %>%
rstatix::add_xy_position()
p <- ToothGrowth %>%
ggplot(aes(x = factor(dose), y = len)) +
stat_summary(geom = "col", fun = mean,aes(fill = factor(dose))) +
stat_summary(geom = "errorbar",
fun = mean,
fun.min = function(x) mean(x) - sd(x),
fun.max = function(x) mean(x) + sd(x),
width = 0.3) +
theme_prism() +
coord_cartesian(ylim = c(0, 40)) +
scale_y_continuous(breaks = seq(0, 40, 5), expand = c(0, 0))
p1 <- p +theme(legend.position = 'none') + add_pvalue(df_p_val, label = "p.adj.signif")
p2 <- p+theme(legend.position = 'none') + add_pvalue(df_p_val, label = "p.adj.signif", remove.bracket = TRUE)
后记
以上是使用R进行绘制柱状图并且加上统计分析的过程,其中一些细节改变图例,更换颜色,导出图片,改变坐标轴 等等没有细致说明,可以进行细微调节。另外,统计分析使用了rstatix这个包,能够和tidyverse很好的结合,后续专门对统计分析进行梳理