生信软件10 - DNA/RNA/蛋白多序列比对图R包ggmsa
R包安装
BiocManager::install("ggplot2")
BiocManager::install("ggmsa")
DNA多序列比对图
# 导入内置核酸序列
nt_sequences <- system.file("extdata", "LeaderRepeat_All.fa", package = "ggmsa")
ggmsa(nt_sequences, color="Chemistry_AA", font = "DroidSansMono", char_width = 0.5, seq_name = TRUE)
RNA多序列比对图
miRNA_sequences <- system.file("extdata", "seedSample.fa", package = "ggmsa")
ggmsa(miRNA_sequences, font = 'DroidSansMono',color = "Chemistry_NT", none_bg = TRUE) +
geom_seed(seed = "GAGGUAG", star = FALSE)ggmsa(miRNA_sequences, font = 'DroidSansMono',color = "Chemistry_NT") +
geom_seed(seed = "GAGGUAG", star = TRUE)
蛋白质多序列比对图
protein_sequences <- system.file("extdata", "sample.fasta", package = "ggmsa")
ggmsa(protein_sequences, 221, 280, seq_name = TRUE, char_width = 0.5) +
geom_seqlogo(color = "Chemistry_AA") + geom_msaBar()
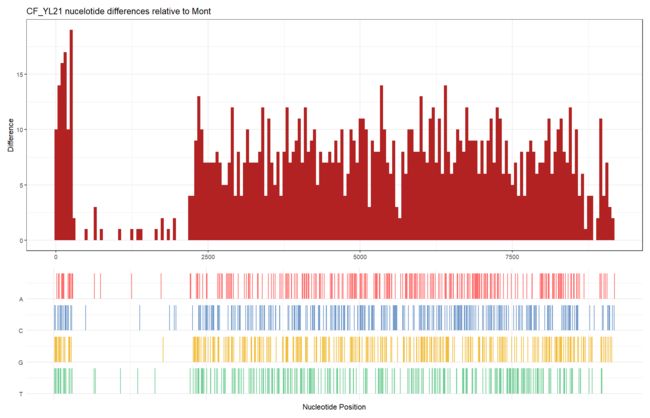
基因组变异图
fas <- list.files(system.file("extdata", "GVariation", package="ggmsa"), pattern="fas", full.names=TRUE)
x <- seqdiff(fas[1], reference=1)
plot(
x,
width = 50,
title = "auto",
xlab = "Nucleotide Position",
by = "bar",
fill = "firebrick",
colors = c(A = "#ff6d6d", C = "#769dcc", G = "#f2be3c", T = "#74ce98"),
xlim = NULL
)
生信软件文章推荐
生信软件1 - 测序下机文件比对结果可视化工具 visNano
生信软件2 - 下游比对数据的统计工具 picard
生信软件3 - mapping比对bam文件质量评估工具 qualimap
生信软件4 - 拷贝数变异CNV分析软件 WisecondorX
生信软件5 - RIdeogram包绘制染色体密度图
生信软件6 - bcftools查找指定区域的变异位点信息
生信软件7 - 多线程并行运行Linux效率工具Parallel
生信软件8 - bedtools进行窗口划分、窗口GC含量、窗口测序深度和窗口SNP统计
生信软件9 - 多公共数据库数据下载软件Kingfisher