WES学习2:外显子测序分析——小男孩的硬骨化病基因突变
首先感谢生信技能树!
书接上回,看完了jimmy B站的WES视频及教程《给学徒的WES数据分析流程》。因为不知道他的原始数据在哪,找了一个乳腺癌WES cohort队列把整个流程跑完了。
然后还想找一些WES/WGS的现成教程跟着学习的。结果找到这个教程:Exome sequencing data analysis for diagnosing a genetic disease 。
是一个在线数据处理平台:galaxy。当然了,先是按教程走完了分析。结果还不错,发现了导致骨硬化病的基因CA2。
在线服务器挺好的,可是如果自己原始的几百G的数据上传给galaxy,国内的这种网络环境下,估计上传完了,地球也毁灭了。
所以,接下来想着在本地电脑也复现一下整个流程。
一个想着安装galaxy,结果又跟着入了一下坑,自己手动在本地安装了一下galaxy。发现要自己一个个的安装这无数个软件,着实麻烦。最后安装好了,还是没用。还是在线服务器好用。
不用galaxy,这个教程的最后call完variant,最后在gemini里面进行一些相关遗传性状分析。折腾了好久,入了第二个坑!
上述两个方面galaxy和Gemini的教程,中文教程都很少。直接看原版的说明书才是正道。
最后软件环境准备好了,用这个教程的原始fastq数据,折腾了两天,建立一个几个单/多样本的全自动WES流程分析。参考:《GATK4全基因组数据分析最佳实践,我以这篇文章为标志,终结当前WGS系列数据分析的流程主体问题 | 完全代码》。
初步了解了外显子测序分析处理的原理和原则。原文贴的图片代码,我一行行代码敲出来,然后进行了优化,合并了单样本和多样本处理流程。
大概介绍一下这个疾病,来自百度:
骨硬化病,又称大理石骨症,或称原发性脆骨硬化症。是一种少见的全身性骨结构发育异常的先天性疾病,颅骨为好发部位之一。骨质极为密,并失去原来的结构,犹如大理石。但骨脆性增加易发生骨折。尚可以伴贫血、眼萎缩及耳聋等情况。一般认为,绝大多数病人在出生前即已开始有病变。根据临床表现分为恶性(幼儿型)及良性(成人型)两种,前者常为死产或出生后死于贫血,预后差。
石骨症的发病原因尚不明确,可能与骨吸收异常有关,致使钙盐过量沉积于骨内,外观呈大理石或象牙样,脆性增加。本病有家族史,多见于近亲结婚的子女中。有人认为属遗传性疾病。本病分轻重二型,轻型为显性遗传,重型为隐性遗传。
再看英文资料:
https://ghr.nlm.nih.gov/condition/osteopetrosis
密切相关的突变基因:
CA2
CLCN7
IKBKG
ITGB3
OSTM1
PLEKHM1
TCIRG1
TNFRSF11A
TNFSF11
下面开始分析这个小男孩的外显子测序数据。
1. 数据获取
作者提供的fastq数据:
https://zenodo.org/record/3243160/files/father_R1.fq.gz
https://zenodo.org/record/3243160/files/father_R2.fq.gz
https://zenodo.org/record/3243160/files/mother_R1.fq.gz
https://zenodo.org/record/3243160/files/mother_R2.fq.gz
https://zenodo.org/record/3243160/files/proband_R1.fq.gz
https://zenodo.org/record/3243160/files/proband_R2.fq.gz2. 搭建好的流程
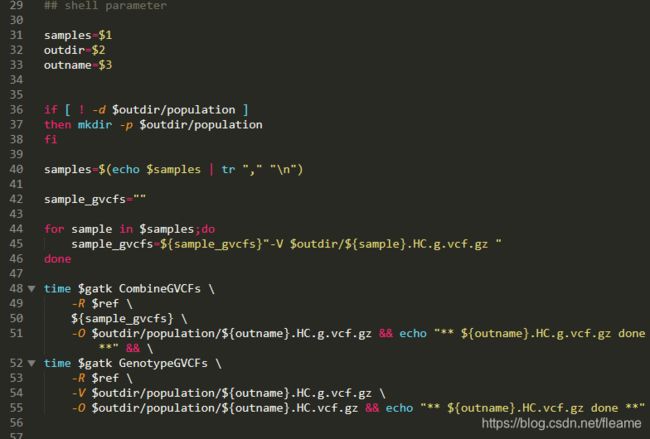
多样本分析分两步走:
第一步先获取每个样本的gvcf,
第二步,所有的gvcf合并,一起call variant,最后再进行一次质量过滤。
2.1 第一步
mkdir wes.boy && cd wes.boy
conda activate rna3 # conda安装好了gatk4
for sample in {father,mother,proband};do
echo $sample;
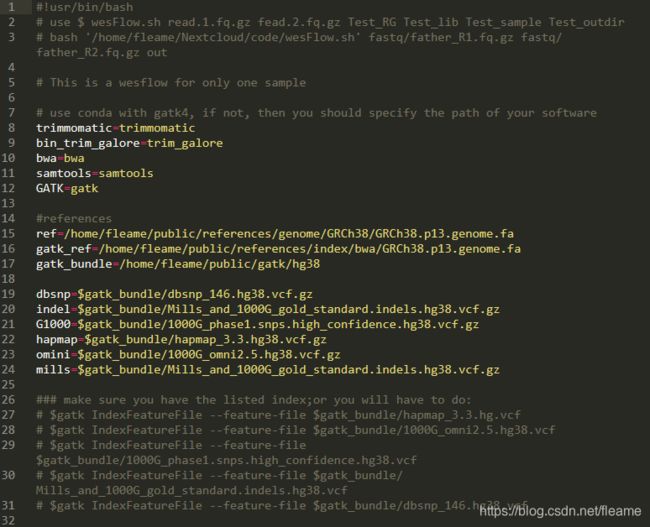
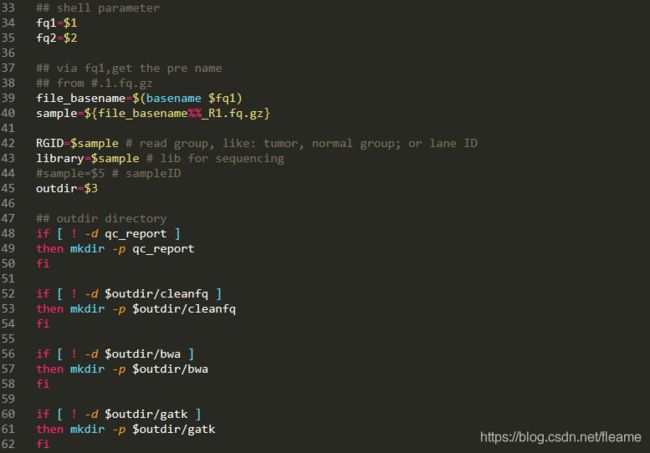
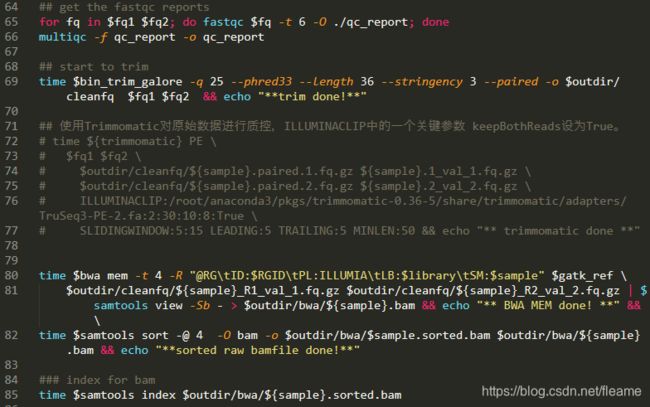
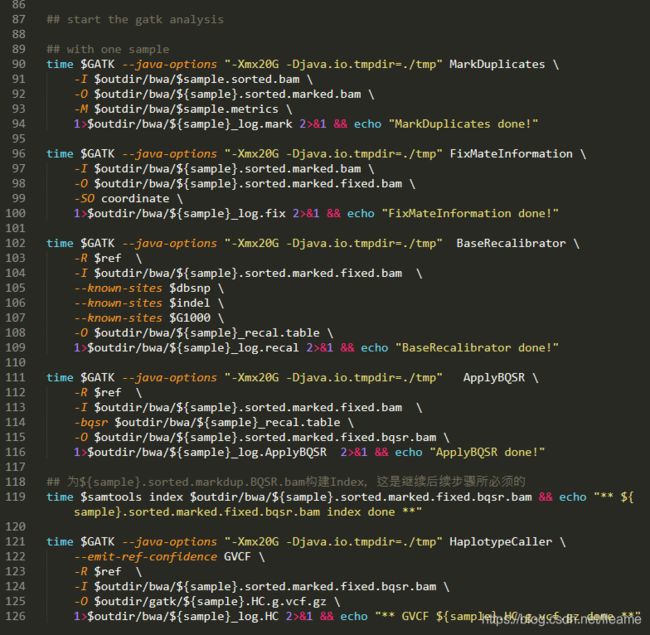
bash /home/fleame/Nextcloud/code/wesFlow/wesFlow_multi_1_to_gvcf.sh fastq/${sample}_R1.fq.gz fastq/${sample}_R2.fq.gz out;
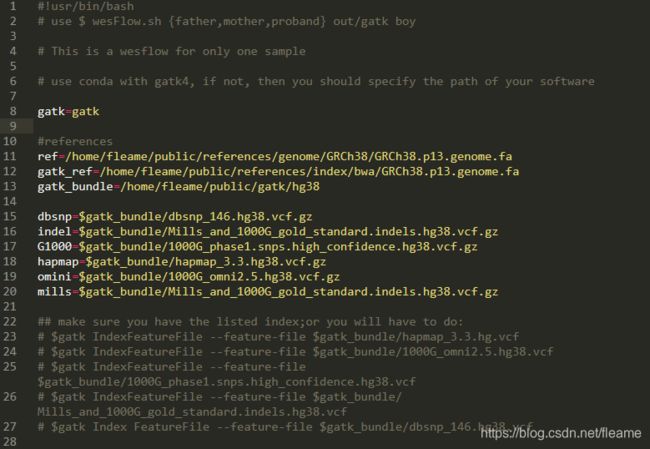
donewesFlow_multi_1_to_gvcf.sh:
2.2 第二步
bash '/home/fleame/Nextcloud/code/wesFlow/wesFlow_multi_2_to_vcf.sh' father,mother,proband out/gatk boyswesFlow_multi_2_to_vcf.sh:
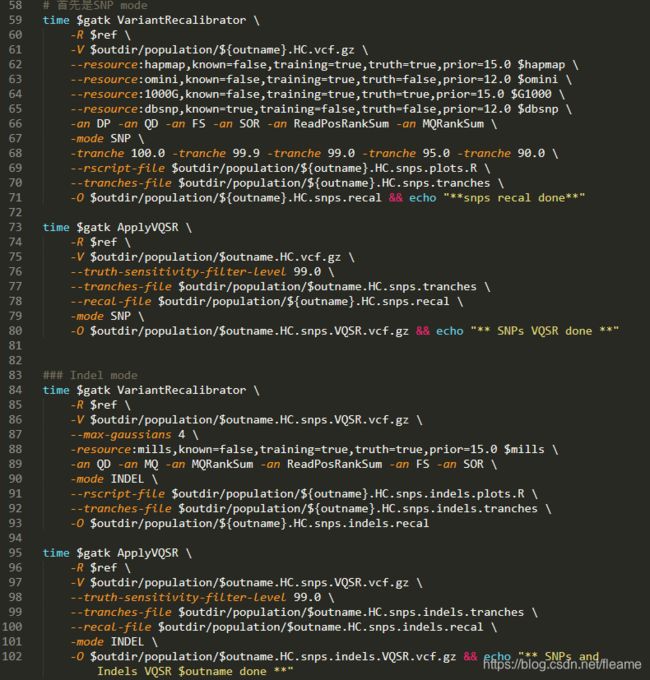
行到这里已经基本拿到了所有样本的vcf了。后续是否进行进一步的过滤,得根据你们的课题考虑。后面的过滤会过滤很多信息,这是需要注意的。你可以用这个帖子的数据,对比试试。
这样所有流程执行完毕。得到了你想要的所有样本的vcf文件。
3 进行vcf注释
使用snpEff 注释,注释很简单,关键是相关的参考文件太大,太难下载!
## 这里注意一下,最好都是所有的参考基因组版本一致的。不过我测试了hg38,hg19,最后出来的结果差不多。
mkdir snpEff && cd snpEff
snpEffpath=/home/fleame/biosoft/snpEff # project path
java -Xmx4g -jar $snpEffpath/snpEff.jar -v -stats boy.html hg19 out/gatk/pupulation/boy.HC.vcf.gz > boy.ann.vcfCreate family tree, ped for gemini load. save as 'ped.tabular'
#family_id name paternal_id maternal_id sex phenotype
FAM father 0 0 1 1
FAM mother 0 0 2 1
FAM proband father mother 1 2gemini load and do autosomal_recessive
gemini load -v boy.ann.vcf -t snpEff -p ped.tabular boy.db
gemini autosomal_recessive boy.db \
--columns "chrom, start,ref, alt, impact, gene, clinvar_sig, clinvar_disease_name, clinvar_gene_phenotype, rs_ids" \
--filter "impact_severity!='LOW'" \
> results.tabular
## the result show:
chrom start ref alt impact gene clinvar_sig clinvar_disease_name clinvar_gene_phenotype rs_ids variant_id family_id family_members family_genotypes samples family_count
chr8 85473750 G A stop_gained CA2 None None carbonic_anhydrase_ii_variant|osteopetrosis_with_renal_tubular_acidosis None 3608 FAM father(father;unaffected;male),mother(mother;unaffected;female),proband(proband;affected;male) G/A,G/A,A/A proband 1 这样,我们就在这个男孩身上找到了一个与osteopetrosis相关的常染色体隐性遗传基因CA2的突变,位于第8号染色体上坐标85473750,G突变成了A,导致了CA2 “stop_gained”。父亲母亲都是等位基因突变一半,性状都没有问题,遗传给这孩子等位基因全突变,1/4的几率得了硬骨化病。可怜的娃!
学习技术不是我们的目的。技术始终是工具,我们应该懂得利用工具解决生物学问题。
###############
请注意,上面的流程代码是图片。两个目的:第一,如果你真的想学,和我一样敲一遍代码是最好的。第二,实在是想要源码,我也无私奉上,但需要关注微信公众号:“颗粒神经园”,并回复关键词“wes.boy”。
###############