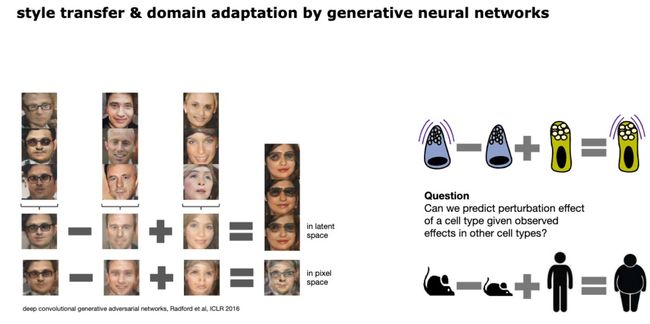
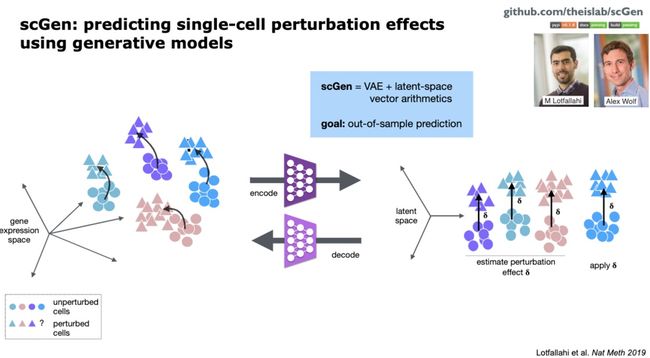
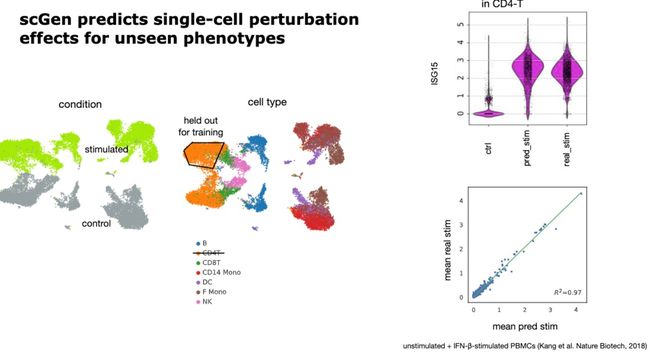
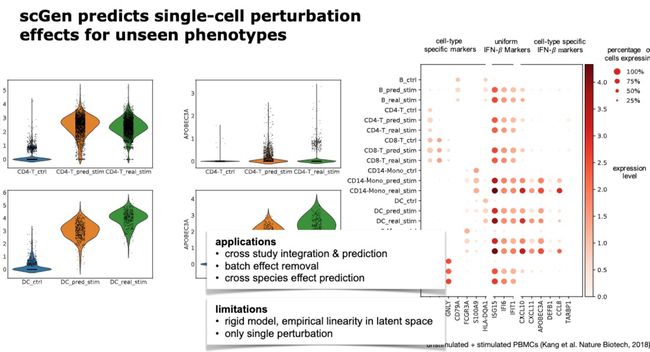
- OpenAI的编程语言和框架,给程序员带来了帮助有哪些
API技术大佬Anzexi58
OpenAI人工智能人工智能深度学习
OpenAI是一个人工智能开发公司,成立于2015年,总部位于美国旧金山。这家公司致力于研究和开发先进的人工智能技术,旨在将这些技术应用到解决全球一些最棘手的问题上。OpenAI以其卓越的技术和实验室出品的groundbreakingAIpapers而闻名。OpenAI的研究涉及深度学习、自然语言处理、视觉感知、强化学习等多个领域,并已在各种应用中取得了令人瞩目的成果。例如,在机器人领域,Open
- DeepSeek:突破传统的AI算法与下载排行分析
smart_ljh
行业搜索人工智能AI
DeepSeek的AI算法突破DeepSeek相较于OpenAI以及其它平台的性能对比DeepSeek的下载排行分析(截止2025/1/28AI人工智能相关DeepSeek甚至一度被推上了搜索)未来发展趋势总结在人工智能技术飞速发展的当下,搜索引擎市场也迎来了新的变革。DeepSeek,作为一款基于深度学习技术和大数据算法的搜索引擎,以其独特的优势在国内外市场上引起了广泛关注。下面介绍一下针对De
- DeepSeek--通向通用人工智能的深度探索者
油泼辣子多加
专业名词解释人工智能
一、词源与全称“DeepSeek"由"Deep”(深度)与"Seek"(探索)组合而成,中文译名为"深度求索"。其全称为"深度求索人工智能基础技术研究有限公司",英文对应"DeepSeekArtificialIntelligenceResearchInstitute"。这一命名体现了企业对深度学习技术与未知领域持续探索的双重追求。二、发展历程初创期(2023)公司成立于中国杭州,创始团队汇聚了来自
- linux git clone出现fatal: unable to access Failed to connect to github.com port 443: Timed out解决方案
herosunly
C/C++/Linux解决方案linuxgitgithubtimeoutport443
大家好,我是herosunly。985院校硕士毕业,现担任算法研究员一职,热衷于机器学习算法研究与应用。曾获得阿里云天池比赛第一名,CCF比赛第二名,科大讯飞比赛第三名。拥有多项发明专利。对机器学习和深度学习拥有自己独到的见解。曾经辅导过若干个非计算机专业的学生进入到算法行业就业。希望和大家一起成长进步。 本文主要介绍了linuxgitclone出现fatal:unabletoaccessF
- 人脸识别的经典深度学习方法
明初啥都能学会
深度学习人工智能
人脸识别的经典深度学习方法引言1.卷积神经网络(CNN)1.1LeNet1.2AlexNet1.3VGGNet1.4ResNet2.人脸检测2.1Viola-Jones算法2.2基于深度学习的人脸检测3.人脸特征提取3.1主成分分析(PCA)3.2人脸对齐3.2.1基于特征点的对齐3.2.2基于深度学习的对齐4.人脸识别模型4.1传统机器学习方法4.2基于深度学习的方法5.公式解读5.1卷积运算5
- 基于深度学习的遥感目标检测系统:UI界面、R-CNN模型与数据集准备
2025年数学建模美赛
R-CNN检测系统人工智能深度学习r语言cnnpythonui目标检测
一、引言遥感图像中的目标检测在很多领域,如环境监测、土地利用、城市规划、农业资源监测等方面有着广泛应用。遥感图像具有高分辨率和丰富的空间信息,但同时也带来了目标检测中的许多挑战,特别是在目标尺度变化、遮挡和复杂背景的情况下。因此,采用深度学习技术,尤其是卷积神经网络(CNN)和区域卷积神经网络(R-CNN),在遥感图像目标检测中取得了显著的成果。本文将详细介绍基于深度学习的遥感目标检测系统,使用R
- Kaggle房价预测
一名小菜鸟的学习之路
深度学习pytorch深度学习机器学习python人工智能神经网络
Kaggle房价预测作为深度学习基础篇章的总结,我们将对本章内容学以致用。下面,让我们动手实战一个Kaggle比赛:房价预测。本节将提供未经调优的数据的预处理、模型的设计和超参数的选择。我们希望读者通过动手操作、仔细观察实验现象、认真分析实验结果并不断调整方法,得到令自己满意的结果。%matplotlibinlineimporttorchimporttorch.nnasnnimportnumpya
- C++ 与机器学习:构建高效推理引擎的秘诀
salsm
C++编程魔法师c++机器学习开发语言
随着深度学习模型逐渐从研究走向生产环境,推理能力成为部署中的关键环节。模型的推理引擎需要以极低的延迟快速处理输入数据,同时最大化地利用硬件资源。虽然Python被广泛用于模型的训练和开发,但C++却在推理领域独占鳌头,其性能优势和硬件控制能力无可替代。在这篇文章中,我们将从为什么选择C++、构建高效推理引擎的细节,以及相似的开源项目三个方面深入探讨如何利用C++打造高效的机器学习推理引擎。目录为什
- 《动手学深度学习》(PyTorch版)
chaser&upper
深度学习pytorch深度学习python
《动手学深度学习》PyTorch版前言简介面向人群食用方法方法一方法二方法三目录原书地址引用阅读指南前言读书啦!!!本项目将《动手学深度学习》原书中MXNet代码实现改为PyTorch实现。原书作者:阿斯顿·张、李沐、扎卡里C.立顿、亚历山大J.斯莫拉以及其他社区贡献者,GitHub地址:https://github.com/d2l-ai/d2l-zh此书的中英版本存在一些不同,针对此书英文版的P
- 从简单到深刻的认知发展
AI架构设计之禅
计算机软件编程原理与应用实践javapythonjavascriptkotlingolang架构人工智能
认知发展,人工智能,深度学习,神经网络,机器学习,自然语言处理,计算机视觉1.背景介绍认知发展是人类从简单到复杂的思维方式演进的过程,它涉及感知、记忆、语言、推理和决策等多个方面。随着人工智能技术的飞速发展,我们开始尝试用计算机模拟人类的认知能力,构建能够学习、理解和解决复杂问题的智能系统。从早期的符号逻辑到如今的深度学习,人工智能的发展经历了多个阶段。早期的人工智能研究主要集中在规则和逻辑推理上
- 使用onnxruntime-web 运行yolov8-nano推理
CHEN_RUI_2200
机器学习YOLO
ONNX(OpenNeuralNetworkExchange)模型具有以下两个特点促成了我们可以使用onnxruntime-web直接在web端上运行推理模型,为了让这个推理更直观,我选择了试验下yolov8识别预览图片:1.跨平台兼容性ONNX是一种开放的格式,可以在不同的深度学习框架之间共享模型,如PyTorch、TensorFlow、MXNet和Caffe2。这使得用户可以在一个框架中训练模
- 人工智能学习框架:深入解析与实战指南
一ge科研小菜鸡
人工智能人工智能
个人主页:一ge科研小菜鸡-CSDN博客期待您的关注引言随着人工智能(AI)技术的飞速发展,深度学习、强化学习和自然语言处理等领域的应用愈加广泛。掌握人工智能学习框架(如TensorFlow、PyTorch、Keras等)已成为开发智能系统、研究前沿技术的必备技能。本指南将全面介绍人工智能主流学习框架的特点、安装方法、核心功能,以及通过实践案例展示如何使用这些框架进行AI模型开发、训练与优化。1.
- AI人工智能深度学习算法:搭建可拓展的深度学习模型架构
AI天才研究院
大数据AI人工智能AI大模型企业级应用开发实战大厂Offer收割机面试题简历程序员读书硅基计算碳基计算认知计算生物计算深度学习神经网络大数据AIGCAGILLMJavaPython架构设计Agent程序员实现财富自由
AI人工智能深度学习算法:搭建可拓展的深度学习模型架构作者:禅与计算机程序设计艺术/ZenandtheArtofComputerProgramming1.背景介绍1.1问题的由来随着人工智能技术的飞速发展,深度学习作为其主要驱动力之一,已经在各个领域取得了显著的成果。然而,随着模型规模的不断扩大,如何高效地搭建、训练和部署深度学习模型,成为一个亟待解决的问题。传统的单机训练方式在计算资源有限的情况
- # 第一章:认识chatgpt
出门喝奶茶
chatgptchatgpt
chatgpt发展背景详细介绍一、基础理论背景人工智能和自然语言处理的兴起早期理论:20世纪中期,人工智能(AI)初见端倪,目标是模拟人类智能。自然语言处理作为AI的重要分支,致力于让机器理解和生成人类语言。关键里程碑:1980年代的统计方法和2000年代的神经网络技术,使NLP实现了从规则驱动到数据驱动的转变。神经网络与深度学习2010年代,深度学习的兴起极大推动了NLP的发展。基于大规模语料库
- 机器学习&深度学习目录
UQI-LIUWJ
各专栏目录深度学习人工智能1024程序员节
机器学习模型机器学习笔记:Transformer_刘文巾的博客-CSDN博客attention相关机器学习笔记:attention_UQI-LIUWJ的博客-CSDN博客机器学习笔记:ELMOBERT_UQI-LIUWJ的博客-CSDN博客机器学习笔记:ViT(论文AnImageIsWorth16X16Words:TransformersforImageRecognitionatScale)_UQ
- 遥感深度学习过程中图像分割的尺寸对模型训练结果的影响
司南锤
深度学习遥感深度学习人工智能
1.计算资源与显存占用大尺寸图像:需要更高的显存和计算资源,可能限制训练时的批大小(batchsize),甚至导致无法训练。解决方案:通常将大图裁剪为小尺寸的补丁(patches),例如256x256或512x512。小尺寸图像:显存占用低,但可能丢失全局上下文信息(如大面积地物分布),影响模型对复杂场景的理解。2.模型感受野与上下文信息小尺寸输入:模型感受野受限,可能无法捕捉大范围地物(如河流、
- #深度学习:从基础到实践
single_ffish
深度学习gpt神经网络生成对抗网络1024程序员节
深度学习是人工智能领域近年来最为火热的技术之一。它通过构建由多个隐藏层组成的神经网络模型,能够从海量数据中自动学习特征和表征,在图像识别、自然语言处理、语音识别等领域取得了突破性进展。本文将全面介绍深度学习的基础知识、主要算法和实践应用,帮助您快速掌握这一前沿技术。1.深度学习的基础1.1人工神经网络深度学习是基于人工神经网络(ArtificialNeuralNetwork,ANN)的一种机器学习
- 【深度学习|地学应用】遥感与深度学习:揭示梦柯冰川奥秘的前沿应用与实践解析
985小水博一枚呀
深度学习地学应用深度学习人工智能
【深度学习|地学应用】遥感与深度学习:揭示梦柯冰川奥秘的前沿应用与实践解析【深度学习|地学应用】遥感与深度学习:揭示梦柯冰川奥秘的前沿应用与实践解析文章目录【深度学习|地学应用】遥感与深度学习:揭示梦柯冰川奥秘的前沿应用与实践解析前言一、使用高分6号遥感影像结合深度学习对梦柯冰川的研究方向1.冰川边界自动化提取2.冰川变化动态监测3.冰川分类与分布特征分析二、梦柯冰川(MunkuGlacier)的
- 基于R-CNN深度学习的无人机目标检测系统:数据集、模型和UI界面的完整实现
2025年数学建模美赛
R-CNN检测系统深度学习cnn无人机计算机视觉目标检测人工智能
摘要随着无人机技术的迅猛发展,无人机在军事、农业、环境监测等多个领域的应用日益广泛。无人机目标检测系统的建设成为提升无人机自主飞行和环境感知能力的重要环节。本文将详细介绍如何构建一个基于深度学习的无人机目标检测系统,采用R-CNN(区域卷积神经网络)算法,通过用户界面设计和数据集处理,实现高效的目标检测功能。通过本项目,旨在为无人机目标检测提供一种可行的解决方案,并提高其在复杂环境下的工作效率。目
- 【Python】已解决:error: subprocess-exited-with-error
屿小夏
python开发语言linux
个人简介:某不知名博主,致力于全栈领域的优质博客分享|用最优质的内容带来最舒适的阅读体验!文末获取免费IT学习资料!文末获取更多信息精彩专栏推荐订阅收藏专栏系列直达链接相关介绍书籍分享点我跳转书籍作为获取知识的重要途径,对于IT从业者来说更是不可或缺的资源。不定期更新IT图书,并在评论区抽取随机粉丝,书籍免费包邮到家AI前沿点我跳转探讨人工智能技术领域的最新发展和创新,涵盖机器学习、深度学习、自然
- AI在电商平台商品描述生成中的应用
AI天才研究院
计算AI大模型企业级应用开发实战大数据AI人工智能javapythonjavascriptkotlingolang架构人工智能大厂程序员硅基计算碳基计算认知计算生物计算深度学习神经网络大数据AIGCAGILLM系统架构设计软件哲学Agent程序员实现财富自由
AI在电商平台商品描述生成中的应用关键词:人工智能、电商平台、商品描述、自然语言处理、机器学习、深度学习摘要:本文深入探讨了人工智能在电商平台商品描述生成中的应用。首先,我们回顾了人工智能的概述和电商平台的发展背景。随后,分析了商品描述在电商平台中的重要性以及存在的问题。接下来,我们重点介绍了AI在商品描述生成中的应用技术,包括自然语言处理、机器学习和深度学习等。文章还通过实战案例展示了AI商品描
- 【Python】已解决:(cmd进入Python环境报错)No Python at ‘C:\Users…\Python\Python39\python.exe’
屿小夏
pythonlinux开发语言
个人简介:某不知名博主,致力于全栈领域的优质博客分享|用最优质的内容带来最舒适的阅读体验!文末获取免费IT学习资料!文末获取更多信息精彩专栏推荐订阅收藏专栏系列直达链接相关介绍书籍分享点我跳转书籍作为获取知识的重要途径,对于IT从业者来说更是不可或缺的资源。不定期更新IT图书,并在评论区抽取随机粉丝,书籍免费包邮到家AI前沿点我跳转探讨人工智能技术领域的最新发展和创新,涵盖机器学习、深度学习、自然
- 深度学习之基于Django+YOLOv5商标识别
Q1744828575
pythonplotlypython
欢迎大家点赞、收藏、关注、评论啦,由于篇幅有限,只展示了部分核心代码。文章目录一项目简介二、功能三、系统四.总结一项目简介 一、项目背景在数字化时代,商标作为企业的重要资产,其保护和管理显得尤为重要。然而,传统的商标识别方法往往依赖于人工审查,效率低下且容易出错。随着深度学习技术的不断发展,尤其是目标检测领域的进步,自动化、高精度的商标识别成为可能。本项目旨在利用DjangoWeb框架和YOLO
- 动手学深度学习-卷积神经网络-3填充和步幅
像污秽一样
动手学深度学习深度学习cnn人工智能神经网络
目录填充步幅小结在上一节的例子(下图)中,输入的高度和宽度都为3,卷积核的高度和宽度都为2,生成的输出表征的维数为2×2。正如我们在上一节中所概括的那样,假设输入形状为nh×nw,卷积核形状为kh×kw,那么输出形状将是(nh−kh+1)×(nw−kw+1)。因此,卷积的输出形状取决于输入形状和卷积核的形状。还有什么因素会影响输出的大小呢?本节我们将介绍填充(padding)和步幅(stride)
- RTX 4090D和A10和T4显卡差别、价格对比
张3蜂
软件安装部署开源神经网络人工智能深度学习机器学习
RTX4090D、NVIDIAA10和NVIDIAT4三款显卡在设计用途、性能、功耗、价格等方面都有显著差异,以下是它们的主要区别:1.设计用途:RTX4090D:面向高端消费者市场,主要用于游戏、内容创作和3D渲染,适合需要高图形性能的用户,如游戏玩家和内容创作者。NVIDIAA10:专为数据中心和企业应用设计,优化了AI推理、深度学习、虚拟桌面基础设施(VDI)以及多实例GPU(多用户共享显卡
- 【TVM教程】为 Mobile GPU 自动调优卷积网络
ApacheTVM是一个深度的深度学习编译框架,适用于CPU、GPU和各种机器学习加速芯片。更多TVM中文文档可访问→https://tvm.hyper.ai/作者:LianminZheng,EddieYan针对特定设备的自动调优对于获得最佳性能至关重要。本文介绍如何调优整个卷积网络。TVM中MobileGPU的算子实现是以template形式编写的。该template有许多可调参数(tile因子
- 非凸科技招聘来啦!技术岗及非技术岗由你选!欢迎大家加入!
招聘
公司介绍:非凸科技成立于2018年,是国内领先的智能算法和交易系统服务公司,专注于智能算法交易领域的研究和开发。公司特点:投研团队来自华尔街顶级资管公司BlackRock等,以及多位来自腾讯、字节跳动的顶尖工程师;在职员工100+,投研和技术团队占总人数比例75%,多位成员是ACM/ICPCWorldFinal选手;公司司正基于Rust生态,结合机器学习、深度学习等新兴技术,打造高效率、低延迟、高
- 深度学习-70-大语言模型LLM之基于大模型LLM与检索增强技术RAG的智能知识库
皮皮冰燃
深度学习人工智能深度学习语言模型
文章目录1RAG出现的背景2搭建过程2.1数据收集2.2数据处理2.2.1数据清洗与预处理2.2.2文本分块2.2.3微调数据格式统一2.3建立向量索引2.4大模型选择与微调3开源知识库项目3.1FastGPT3.2AnythingLLM3.3LangChain-Chatchat4商业化解决方案4.1百度智能云+千帆大模型知识库4.2阿里云PAI+向量检索5参考附录1RAG出现的背景自从2022年
- transformer.js(一):这个前端大模型运行框架的可运行环境、使用方式、代码示例以及适合与不适合的场景
余生H
前端的AI工具书前端transformerjavascripthugginfacewebmlweb大模型
随着大模型的广泛应用,越来越多的开发者希望在前端直接运行机器学习模型,从而减少对后端的依赖,并提升用户体验。Transformer.js是一个专为前端环境设计的框架,它支持运行基于Transformer架构的深度学习模型,尤其是像BERT、GPT等广泛应用于自然语言处理(NLP)的模型。本文将全面解析Transformer.js的运行环境、使用方式、代码示例,以及其能够完成的功能与目前的限制,帮助
- Python magenta库:一款人工智能生成音乐与艺术的创新工具
程序员喵哥
python人工智能开发语言
更多Python学习内容:ipengtao.com随着人工智能在创意领域的不断进步,音乐和艺术生成正成为一种新的可能性。Magenta是由Google推出的一个开源项目,它结合了深度学习与艺术创作,为开发者提供了一系列强大的工具,帮助他们创作音乐、绘画等艺术作品。基于TensorFlow,Magenta不仅适用于研究人员,也适合开发者和艺术家,提供了易于上手的API和丰富的模型。安装在使用Mage
- Spring的注解积累
yijiesuifeng
spring注解
用注解来向Spring容器注册Bean。
需要在applicationContext.xml中注册:
<context:component-scan base-package=”pagkage1[,pagkage2,…,pagkageN]”/>。
如:在base-package指明一个包
<context:component-sc
- 传感器
百合不是茶
android传感器
android传感器的作用主要就是来获取数据,根据得到的数据来触发某种事件
下面就以重力传感器为例;
1,在onCreate中获得传感器服务
private SensorManager sm;// 获得系统的服务
private Sensor sensor;// 创建传感器实例
@Override
protected void
- [光磁与探测]金吕玉衣的意义
comsci
这是一个古代人的秘密:现在告诉大家
信不信由你们:
穿上金律玉衣的人,如果处于灵魂出窍的状态,可以飞到宇宙中去看星星
这就是为什么古代
- 精简的反序打印某个数
沐刃青蛟
打印
以前看到一些让求反序打印某个数的程序。
比如:输入123,输出321。
记得以前是告诉你是几位数的,当时就抓耳挠腮,完全没有思路。
似乎最后是用到%和/方法解决的。
而今突然想到一个简短的方法,就可以实现任意位数的反序打印(但是如果是首位数或者尾位数为0时就没有打印出来了)
代码如下:
long num, num1=0;
- PHP:6种方法获取文件的扩展名
IT独行者
PHP扩展名
PHP:6种方法获取文件的扩展名
1、字符串查找和截取的方法
1
$extension
=
substr
(
strrchr
(
$file
,
'.'
), 1);
2、字符串查找和截取的方法二
1
$extension
=
substr
- 面试111
文强chu
面试
1事务隔离级别有那些 ,事务特性是什么(问到一次)
2 spring aop 如何管理事务的,如何实现的。动态代理如何实现,jdk怎么实现动态代理的,ioc是怎么实现的,spring是单例还是多例,有那些初始化bean的方式,各有什么区别(经常问)
3 struts默认提供了那些拦截器 (一次)
4 过滤器和拦截器的区别 (频率也挺高)
5 final,finally final
- XML的四种解析方式
小桔子
domjdomdom4jsax
在平时工作中,难免会遇到把 XML 作为数据存储格式。面对目前种类繁多的解决方案,哪个最适合我们呢?在这篇文章中,我对这四种主流方案做一个不完全评测,仅仅针对遍历 XML 这块来测试,因为遍历 XML 是工作中使用最多的(至少我认为)。 预 备 测试环境: AMD 毒龙1.4G OC 1.5G、256M DDR333、Windows2000 Server
- wordpress中常见的操作
aichenglong
中文注册wordpress移除菜单
1 wordpress中使用中文名注册解决办法
1)使用插件
2)修改wp源代码
进入到wp-include/formatting.php文件中找到
function sanitize_user( $username, $strict = false
- 小飞飞学管理-1
alafqq
管理
项目管理的下午题,其实就在提出问题(挑刺),分析问题,解决问题。
今天我随意看下10年上半年的第一题。主要就是项目经理的提拨和培养。
结合我自己经历写下心得
对于公司选拔和培养项目经理的制度有什么毛病呢?
1,公司考察,选拔项目经理,只关注技术能力,而很少或没有关注管理方面的经验,能力。
2,公司对项目经理缺乏必要的项目管理知识和技能方面的培训。
3,公司对项目经理的工作缺乏进行指
- IO输入输出部分探讨
百合不是茶
IO
//文件处理 在处理文件输入输出时要引入java.IO这个包;
/*
1,运用File类对文件目录和属性进行操作
2,理解流,理解输入输出流的概念
3,使用字节/符流对文件进行读/写操作
4,了解标准的I/O
5,了解对象序列化
*/
//1,运用File类对文件目录和属性进行操作
//在工程中线创建一个text.txt
- getElementById的用法
bijian1013
element
getElementById是通过Id来设置/返回HTML标签的属性及调用其事件与方法。用这个方法基本上可以控制页面所有标签,条件很简单,就是给每个标签分配一个ID号。
返回具有指定ID属性值的第一个对象的一个引用。
语法:
&n
- 励志经典语录
bijian1013
励志人生
经典语录1:
哈佛有一个著名的理论:人的差别在于业余时间,而一个人的命运决定于晚上8点到10点之间。每晚抽出2个小时的时间用来阅读、进修、思考或参加有意的演讲、讨论,你会发现,你的人生正在发生改变,坚持数年之后,成功会向你招手。不要每天抱着QQ/MSN/游戏/电影/肥皂剧……奋斗到12点都舍不得休息,看就看一些励志的影视或者文章,不要当作消遣;学会思考人生,学会感悟人生
- [MongoDB学习笔记三]MongoDB分片
bit1129
mongodb
MongoDB的副本集(Replica Set)一方面解决了数据的备份和数据的可靠性问题,另一方面也提升了数据的读写性能。MongoDB分片(Sharding)则解决了数据的扩容问题,MongoDB作为云计算时代的分布式数据库,大容量数据存储,高效并发的数据存取,自动容错等是MongoDB的关键指标。
本篇介绍MongoDB的切片(Sharding)
1.何时需要分片
&nbs
- 【Spark八十三】BlockManager在Spark中的使用场景
bit1129
manager
1. Broadcast变量的存储,在HttpBroadcast类中可以知道
2. RDD通过CacheManager存储RDD中的数据,CacheManager也是通过BlockManager进行存储的
3. ShuffleMapTask得到的结果数据,是通过FileShuffleBlockManager进行管理的,而FileShuffleBlockManager最终也是使用BlockMan
- yum方式部署zabbix
ronin47
yum方式部署zabbix
安装网络yum库#rpm -ivh http://repo.zabbix.com/zabbix/2.4/rhel/6/x86_64/zabbix-release-2.4-1.el6.noarch.rpm 通过yum装mysql和zabbix调用的插件还有agent代理#yum install zabbix-server-mysql zabbix-web-mysql mysql-
- Hibernate4和MySQL5.5自动创建表失败问题解决方法
byalias
J2EEHibernate4
今天初学Hibernate4,了解了使用Hibernate的过程。大体分为4个步骤:
①创建hibernate.cfg.xml文件
②创建持久化对象
③创建*.hbm.xml映射文件
④编写hibernate相应代码
在第四步中,进行了单元测试,测试预期结果是hibernate自动帮助在数据库中创建数据表,结果JUnit单元测试没有问题,在控制台打印了创建数据表的SQL语句,但在数据库中
- Netty源码学习-FrameDecoder
bylijinnan
javanetty
Netty 3.x的user guide里FrameDecoder的例子,有几个疑问:
1.文档说:FrameDecoder calls decode method with an internally maintained cumulative buffer whenever new data is received.
为什么每次有新数据到达时,都会调用decode方法?
2.Dec
- SQL行列转换方法
chicony
行列转换
create table tb(终端名称 varchar(10) , CEI分值 varchar(10) , 终端数量 int)
insert into tb values('三星' , '0-5' , 74)
insert into tb values('三星' , '10-15' , 83)
insert into tb values('苹果' , '0-5' , 93)
- 中文编码测试
ctrain
编码
循环打印转换编码
String[] codes = {
"iso-8859-1",
"utf-8",
"gbk",
"unicode"
};
for (int i = 0; i < codes.length; i++) {
for (int j
- hive 客户端查询报堆内存溢出解决方法
daizj
hive堆内存溢出
hive> select * from t_test where ds=20150323 limit 2;
OK
Exception in thread "main" java.lang.OutOfMemoryError: Java heap space
问题原因: hive堆内存默认为256M
这个问题的解决方法为:
修改/us
- 人有多大懒,才有多大闲 (评论『卓有成效的程序员』)
dcj3sjt126com
程序员
卓有成效的程序员给我的震撼很大,程序员作为特殊的群体,有的人可以这么懒, 懒到事情都交给机器去做 ,而有的人又可以那么勤奋,每天都孜孜不倦得做着重复单调的工作。
在看这本书之前,我属于勤奋的人,而看完这本书以后,我要努力变成懒惰的人。
不要在去庞大的开始菜单里面一项一项搜索自己的应用程序,也不要在自己的桌面上放置眼花缭乱的快捷图标
- Eclipse简单有用的配置
dcj3sjt126com
eclipse
1、显示行号 Window -- Prefences -- General -- Editors -- Text Editors -- show line numbers
2、代码提示字符 Window ->Perferences,并依次展开 Java -> Editor -> Content Assist,最下面一栏 auto-Activation
- 在tomcat上面安装solr4.8.0全过程
eksliang
Solrsolr4.0后的版本安装solr4.8.0安装
转载请出自出处:
http://eksliang.iteye.com/blog/2096478
首先solr是一个基于java的web的应用,所以安装solr之前必须先安装JDK和tomcat,我这里就先省略安装tomcat和jdk了
第一步:当然是下载去官网上下载最新的solr版本,下载地址
- Android APP通用型拒绝服务、漏洞分析报告
gg163
漏洞androidAPP分析
点评:记得曾经有段时间很多SRC平台被刷了大量APP本地拒绝服务漏洞,移动安全团队爱内测(ineice.com)发现了一个安卓客户端的通用型拒绝服务漏洞,来看看他们的详细分析吧。
0xr0ot和Xbalien交流所有可能导致应用拒绝服务的异常类型时,发现了一处通用的本地拒绝服务漏洞。该通用型本地拒绝服务可以造成大面积的app拒绝服务。
针对序列化对象而出现的拒绝服务主要
- HoverTree项目已经实现分层
hvt
编程.netWebC#ASP.ENT
HoverTree项目已经初步实现分层,源代码已经上传到 http://hovertree.codeplex.com请到SOURCE CODE查看。在本地用SQL Server 2008 数据库测试成功。数据库和表请参考:http://keleyi.com/a/bjae/ue6stb42.htmHoverTree是一个ASP.NET 开源项目,希望对你学习ASP.NET或者C#语言有帮助,如果你对
- Google Maps API v3: Remove Markers 移除标记
天梯梦
google maps api
Simply do the following:
I. Declare a global variable:
var markersArray = [];
II. Define a function:
function clearOverlays() {
for (var i = 0; i < markersArray.length; i++ )
- jQuery选择器总结
lq38366
jquery选择器
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40
- 基础数据结构和算法六:Quick sort
sunwinner
AlgorithmQuicksort
Quick sort is probably used more widely than any other. It is popular because it is not difficult to implement, works well for a variety of different kinds of input data, and is substantially faster t
- 如何让Flash不遮挡HTML div元素的技巧_HTML/Xhtml_网页制作
刘星宇
htmlWeb
今天在写一个flash广告代码的时候,因为flash自带的链接,容易被当成弹出广告,所以做了一个div层放到flash上面,这样链接都是a触发的不会被拦截,但发现flash一直处于div层上面,原来flash需要加个参数才可以。
让flash置于DIV层之下的方法,让flash不挡住飘浮层或下拉菜单,让Flash不档住浮动对象或层的关键参数:wmode=opaque。
方法如下:
- Mybatis实用Mapper SQL汇总示例
wdmcygah
sqlmysqlmybatis实用
Mybatis作为一个非常好用的持久层框架,相关资料真的是少得可怜,所幸的是官方文档还算详细。本博文主要列举一些个人感觉比较常用的场景及相应的Mapper SQL写法,希望能够对大家有所帮助。
不少持久层框架对动态SQL的支持不足,在SQL需要动态拼接时非常苦恼,而Mybatis很好地解决了这个问题,算是框架的一大亮点。对于常见的场景,例如:批量插入/更新/删除,模糊查询,多条件查询,联表查询,