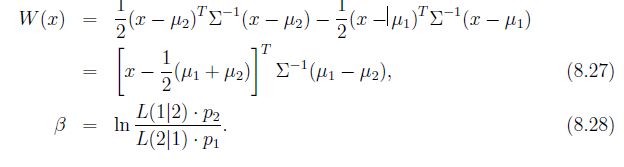R语言 分类算法
一、线性分类法
通俗点理解线性分类法就是画条线,尽量的使同样类别的点,落在线的同一边。如果是三维的,那么画一个面。当不同类样本的协方差矩阵相同时,我们可以在R中使用MASS包的lda函数实现线性判别。lda函数以Bayes判别思想为基础。当分类只有 两种且总体服从多元正态分布条件下,Bayes判别与Fisher判别、距离判别是等价的。
本例使用iris数据集来对花的品种进行分类。首先载入 MASS包,建立判别模型,其中的prior参数表示先验概率。然后利用table函数建立混淆矩阵,比对真实类别和预测类别。
当样本的协方差矩阵相同时用lda函数判别
#用线性判别法,来对鸢尾花数据集进行判别。 > library(MASS) > model <- lda(Species ~ ., data=iris,prior=c(1,1,1)/3) #prior是先验概率。 > predict(model,iris)$class #利用模型预测 [1] setosa setosa setosa setosa setosa setosa setosa [8] setosa setosa setosa setosa setosa setosa setosa [15] setosa setosa setosa setosa setosa setosa setosa [22] setosa setosa setosa setosa setosa setosa setosa [29] setosa setosa setosa setosa setosa setosa setosa [36] setosa setosa setosa setosa setosa setosa setosa [43] setosa setosa setosa setosa setosa setosa setosa [50] setosa versicolor versicolor versicolor versicolor versicolor versicolor [57] versicolor versicolor versicolor versicolor versicolor versicolor versicolor [64] versicolor versicolor versicolor versicolor versicolor versicolor versicolor [71] virginica versicolor versicolor versicolor versicolor versicolor versicolor [78] versicolor versicolor versicolor versicolor versicolor versicolor virginica [85] versicolor versicolor versicolor versicolor versicolor versicolor versicolor [92] versicolor versicolor versicolor versicolor versicolor versicolor versicolor [99] versicolor versicolor virginica virginica virginica virginica virginica [106] virginica virginica virginica virginica virginica virginica virginica [113] virginica virginica virginica virginica virginica virginica virginica [120] virginica virginica virginica virginica virginica virginica virginica [127] virginica virginica virginica virginica virginica virginica virginica [134] versicolor virginica virginica virginica virginica virginica virginica [141] virginica virginica virginica virginica virginica virginica virginica [148] virginica virginica virginica Levels: setosa versicolor virginica
当样本的协方差矩阵不同时,使用qda()函数判别
>model2=qda(Species~.,data=iris,cv=T) #cv使用留一交叉检验(leave-one-out cross-validation),并自动生成预测值 >predict(model)$posterior #查看后验概率
使用这两种线性判别法,需要总体服从多元正态分布。
二、距离判别法
算距离,某个样本点离哪个类比较近,就判断为哪个类别。但这里计算的不是欧氏距离,而是马氏距离。
distinguish.distance <- function(TrnX, TrnG, TstX = NULL, var.equal = FALSE){
if(is.factor(TrnG) == FALSE){ #如果传入的trnG 不是因子,就当成第二个类别看待
mx <- nrow(TrnX); mg <- nrow(TrnG) #把他们放到一起,总共两个分类
TrnX <- rbind(TrnX, TrnG)
TrnG <- factor(rep(1:2, c(mx, mg)))
}
if(is.null(TstX)){
TstX <- TrnX
}
if(is.vector(TstX)){ #如果传入的测试集是向量,变成矩阵并且竖起来
TstX <- t(as.matrix(TstX))
}else if(is.matrix(TstX) != TRUE){ #不是矩阵的变成矩阵
TstX <- as.matrix(TstX)
}
if(is.matrix(TrnX) != TRUE){ #把训练集当矩阵对待
TrnX <- as.matrix(TrnX)
}
nx <- nrow(TstX) #测试集总共有行
#生成一个只有一行的矩阵,长度为要预测的数据的个数,这个用来存测试集的分类
blong <- matrix(rep(0, nx), nrow=1, dimnames=list("blong", 1:nx))
g <- length(levels(TrnG)) #有多少分类
mu <- matrix(0, nrow=g, ncol=ncol(TrnX)) #生成列方向上的平均值矩阵,行数为类别数量
for (i in 1:g){
mu[i,] <- colMeans(TrnX[TrnG==i,]) #求每一列数据,每一个水平的平均值
}
D <- matrix(0, nrow=g, ncol=nx) #新建一个矩阵用来存 待测样本点到个类别的距离。行数是类别数,
if(var.equal == TRUE || var.equal == T){
for(i in 1:g){
D[i,] <- mahalanobis(TstX, mu[i,], var(TrnX))
}
}else{
for (i in 1:g){
D[i,] <- mahalanobis(TstX, mu[i,], var(TrnX[TrnG==i,]))
}
}
for (j in 1:nx){
dmin <- Inf
for (i in 1:g)
if (D[i,j] < dmin){ #循环着跑一下,比较各个类别见哪个距离是最短的。就属于哪个类别
dmin <- D[i,j]; blong[j] <- i
}
}
blong #输出
}
#上面程序中mahalanobis(向量或矩阵,样本中心,协方差矩阵)是用来求马氏距离的。
> source("E:hutao\\learning\\rscript\\myR.R")
> x <- iris[,1:4]
> g <- gl(3,50)
> distinguish.distance(x,g)
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30
blong 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57
blong 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2
58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84
blong 2 2 2 2 2 2 2 2 2 2 2 2 2 3 2 3 2 2 2 2 2 2 2 2 2 2 3
85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108
blong 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 3 3 3 3 3 3 3 3
109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128
blong 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148
blong 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
149 150
blong 3 3
三、贝叶斯分类器
前提假设,总体是正态分布的。
记 统计量ECM(R1, R2) = L(2|1)P(2|1)p1 + L(1|2)P(1|2)p2, 前面是明明是1类,却被判定为2类的概率乘以损失L(2|1)
,后面是另一种损失。 我们要划定分类,使得这个ECM最小。数学上可以证明,在上面这个值达到最小的情况下划分出来的R1、R2,f1(x)/f2(x) 和 L(1|2)p2/L(2|1)p1的大小关系是这2个类的分界。前者大于后者的时候是R1类,另一种情况是R2类。其中f1(x)是R1类的概率密度函数。p是先验概率,L是损失。对上面两个值同时取对数,然后进行约简,得到左边的w 和右边的 β。在两个类别总体协方差相同和不同的情况下,分别求的下面的值

现在变成了 W(x)>=β是 R1类,另一个是R2类。神奇的事情发生了,w统计量和我们在距离判别法里算的距离的差值是一样的。所以贝叶斯可以看成是距离判别法的推广。当β值 = 0 的时候,贝叶斯就是距离判别。
上面是只有两种类型的情况。如果有多种类型,我们先假设,误判的损失是相同的。然后计算d统计量 ,即
dj(x) =(1/2)(x − μj)T Σ^(−1) (x − μj) − ln pj (协方差不同时还要− (1/2)ln(|Σj |)), 计算每个样本到各个分类的d统计量,谁最小就把样本判到谁的类里。根据这个办法写R函数
distinguish.bayes <- function(TrnX, TrnG, p = rep(1, length(levels(TrnG))),
TstX = NULL, var.equal = FALSE){ #p是先验概率,默认都是1
if(is.factor(TrnG) == FALSE){
mx <- nrow(TrnX); mg <- nrow(TrnG)
TrnX <- rbind(TrnX, TrnG)
TrnG <- factor(rep(1:2, c(mx, mg)))
} #上面的代码还是老意思,如果分别传入两个类别的数据,就合成一个。并给因子
if(is.null(TstX) == TRUE){
TstX <- TrnX #没给测试集,就用训练集代替
}
if(is.vector(TstX) == TRUE){
TstX <- t(as.matrix(TstX)) #变成竖着的一维矩阵
}else if(is.matrix(TstX) != TRUE){
TstX <- as.matrix(TstX) #给了测试集 就变成矩阵
}
if(is.matrix(TrnX) != TRUE){
TrnX <- as.matrix(TrnX) #变成矩阵
}
nx <- nrow(TstX) #测试集样本数
blong <- matrix(rep(0, nx), nrow=1,
dimnames=list("blong", 1:nx))
g <- length(levels(TrnG))
mu <- matrix(0, nrow=g, ncol=ncol(TrnX))
for(i in 1:g){
mu[i,] <- colMeans(TrnX[TrnG==i,])
}
D <- matrix(0, nrow=g, ncol=nx)
if(var.equal == TRUE || var.equal == T){
for(i in 1:g){
d2 <- mahalanobis(TstX, mu[i,], var(TrnX))
D[i,] <- d2 - 2*log(p[i])
}
}else{
for(i in 1:g){
S <- var(TrnX[TrnG==i,])
d2 <- mahalanobis(TstX, mu[i,], S)
D[i,] <- d2 - 2*log(p[i])-log(det(S))
}
}
for(j in 1:nx){
dmin <- Inf
for(i in 1:g){
if(D[i,j] < dmin){
dmin <- D[i,j]
blong[j] <- i
}
}
}
blong
} #跟上面的距离判别法比较雷同
> source("E:\\hutao\\learning\\rscript\\myR.R")
> distinguish.bayes(iris[1:4],gl(3,50))
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27
blong 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51
blong 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2
52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75
blong 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 3 2 3 2 3 2 2
76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99
blong 2 2 3 2 2 2 2 2 3 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117
blong 2 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135
blong 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
136 137 138 139 140 141 142 143 144 145 146 147 148 149 150
blong 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
四、Fisher判别
discriminiant.fisher <- function(TrnX1, TrnX2, TstX = NULL){
if(is.null(TstX) == TRUE){
TstX <- rbind(TrnX1,TrnX2)
}
if(is.vector(TstX) == TRUE){
TstX <- t(as.matrix(TstX))
}else if(is.matrix(TstX) != TRUE){
TstX <- as.matrix(TstX)
}
if(is.matrix(TrnX1) != TRUE){
TrnX1 <- as.matrix(TrnX1)
}
if(is.matrix(TrnX2) != TRUE){
TrnX2 <- as.matrix(TrnX2)
}
nx <- nrow(TstX)
blong <- matrix(rep(0, nx), nrow=1, byrow=TRUE,
dimnames=list("blong", 1:nx))
n1 <- nrow(TrnX1); n2 <- nrow(TrnX2)
mu1 <- colMeans(TrnX1); mu2 <- colMeans(TrnX2)
S <- (n1-1)*var(TrnX1) + (n2-1)*var(TrnX2)
mu <- n1/(n1+n2)*mu1 + n2/(n1+n2)*mu2
w <- (TstX-rep(1,nx) %o% mu) %*% solve(S, mu2-mu1);
for (i in 1:nx){
if(w[i] <= 0){
blong[i] <- 1
}else{
blong[i] <- 2
}
}
blong
}
五、决策树
http://www.statmethods.net/advstats/cart.html
照着敲一把
> library(rpart) > fit <- rpart(Kyphosis ~ Age + Number + Start, method="class", data=kyphosis) > fit > plot(fit,uniform=T,main="myTree") > text(fit,use.n=T,all=T,cex=0.8)