DICOM医学图像处理:深入剖析Orthanc的SQLite,了解WADO & RESTful API
背景:
上一篇博文简单翻译了Orthanc官网给出的CodeProject上“利用Orthanc Plugin SDK开发WADO插件”的博文,其中提到了Orthanc从0.8.0版本之后支持快速查询,而原本的WADO请求需要是直接借助于Orthanc内部的REST API逐级定位。那么为什么之前的Orthanc必须要逐级来定位WADO请求的Instance呢?新版本中又是如何进行改进的呢?此篇博文通过分析Orthanc内嵌的SQLite数据库,来剖析Orthanc的RESTful API机制,以及WADO服务的实现。
Orthanc UUID与DICOM UID:
1)Orthanc Plugin SDK模拟实现WADO Server
上一篇博文中提到的LocateStudy、LocateSeries、LocateInstanc函数都不是直接查询WADO请求传入的各级UID(StudyUID、SeriesUID、InstanceUID),而是通过内部构建出等同的RESTful API来实现。举个例子,测试DCM文件名为test1.dcm,其对应的三级UID分别是:
StudyUID=1.3.6.1.4.1.30071.6.176694098609799.4240639413125000,
SeriesUID=1.3.6.1.4.1.30071.6.176694098609799.4240639413125000.1,
InstanceUID(即SOP Instance UID)=2.16.840.114421.81623.9430067258.9493139258,正常的WADO协议规定的请求连接为:
http://localhost:8042/wado?requestType=WADO&studyUID=1.3.6.1.4.1.30071.6.176694098609799.4240639413125000&
seriesUID=1.3.6.1.4.1.30071.6.176694098609799.4240639413125000.1&
objectUID=2.16.840.114421.81623.9430067258.9493139258
按照常规方式来实现的话,应该是直接利用SQL语句在指定的数据库中直接搜索WADO Request中的三级UID,而在Orthanc Plugin SDK实现的WADO插件中,却是分级进行,详细流程如下:
Study级别:第一,LocateStudy函数中构建http://localhost:8042/studies请求,利用内置的REST API服务获得当前数据中所有的studies的UUID(后面会讲到该UUID与DICOM UID之间的转换关系);第二,LocateStudy中的每一个studyUUID,构造http://localhost:8042/studies/XXXX-XXXX-XXXX-XXXX,通过对比返回JSON数据中study["MainDicomTags"]["StudyInstanceUID"]标签值与WADO中的studyUID,实现定位Study的功能;
Series级别:与Study相同,先构造http://localhost:8042/series获取全部seriesUUID,然后针对每个seriesUUID构造http://localhost:8042/series/XXXX-XXXX-XXXX-XXXX,对比返回值中的series["MainDicomTags"]["SeriesInstanceUID"]与seriesUID,实现定位Series的功能;
Instance级别:先构造http://localhost:8042/instances获取全部instanceUUID,然后对每个instanceUUID构造http://localhost:8042/instances/XXXX-XXXX-XXXX-XXXX对比返回值中的instance["MainDicomTags"]["SOPInstanceUID"]与WADO请求中的objectUID,实现最终定位图像的目的。
2)Orthanc UUID与DICOM UID
上面的实现是不是很繁琐啊,哈哈。好在官方Plugin SDK说明博文中给出了最新版的定位方式,具体的实现可参见我上一篇博文(http://blog.csdn.net/zssureqh/article/details/41836885)。那么为何Orthanc起初需要如此繁琐的定位图像呢?这里我们先简单的分析一下Orthanc内部是如何来标记文件的唯一性的,后续章节再详细分析之前Orthanc模拟WADO服务为何如此繁琐。
在Orthanc源码中有这样一个类DicomInstanceHasher(定义在DicomInstanceHasher.h,实现在DicomInstanceHasher.cpp),其注释中如此描述:
/** * This class implements the hashing mechanism that is used to * convert DICOM unique identifiers to Orthanc identifiers. Any * Orthanc identifier for a DICOM resource corresponds to the SHA-1 * hash of the DICOM identifiers. * \note SHA-1 hash is used because it is less sensitive to * collision attacks than MD5. <a * href="http://en.wikipedia.org/wiki/SHA-256#Comparison_of_SHA_functions">[Reference]</a> **/
从描述中我们可以知道Orthanc内部时利用SHA1(百度百科:维基百科:)算法来计算出DCM文件的唯一标识的,具体计算过程为:
PatientID对应的UUID:即向SHA1计算函数中直接输入【PatientID】,获得SHA1值
StudyUID对应的UUID:向SHA1计算函数中输入【PatientID+”|"+StudyUID】,获得SHA1值
SeriesUID对应的UUID:向SHA1计算函数中输入【PatientID+”|"+StudyUID+”|"+SeriesUID】,获得SHA1值
InstanceUID对应的UUID:向SHA1计算函数中输入【PatientID+”|"+StudyUID+”|"+SeriesUID+”|"+InstanceUID】,获得SHA1值
这就是OrthancUUID与DICOM UID之间的转换关系,下一节讲解数据库时再给出真实的示例。
Orthanc SQLite介绍:
1)Orthanc SQLite数据库列表介绍:
Orthanc采用了SQLite嵌入式数据库,对数据库的操作在工程代码中集成,因此在使用过程中并未能感觉到数据库的管理,这也支撑了Orthanc主打的轻型、便捷、网络化优点。下面简单介绍一下Orthanc SQLite数据表的逻辑:
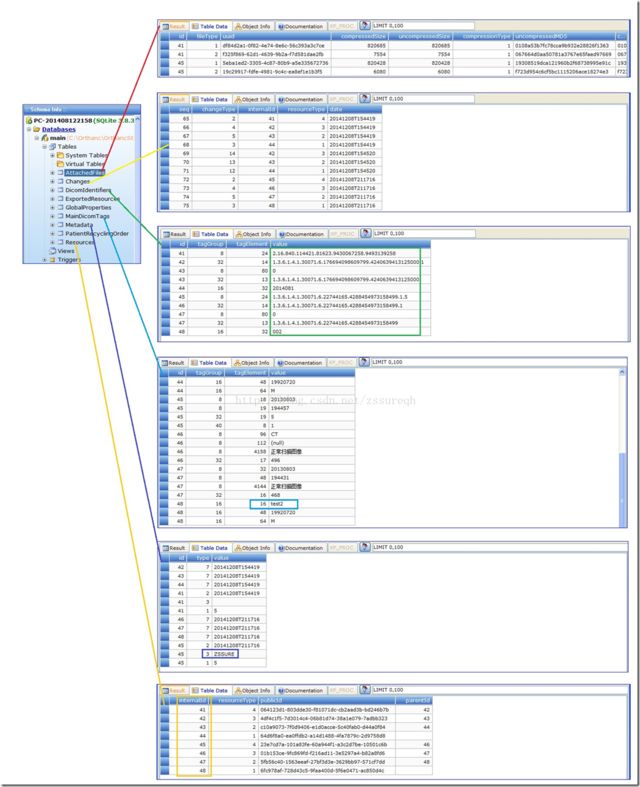
SQLite的数据库文件默认存储位置为:C:\Orthanc\OrthancStoragef\index(其真实后缀为db3)。用SQLite可视化工具打开index文件,可以看到如下几张表:
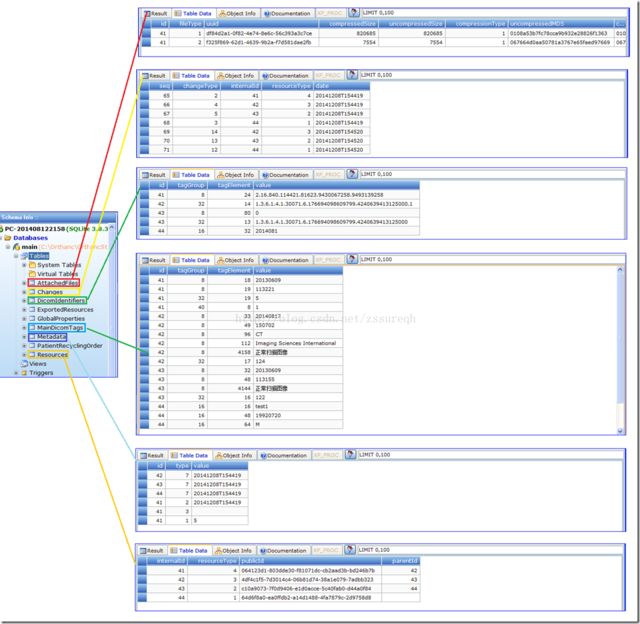
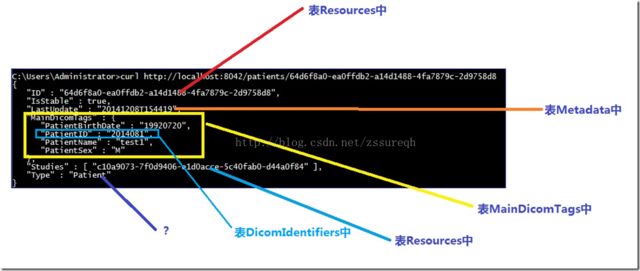
从表名称中可以推断出各表大致的用途:例如AttachedFiles是添加文件的记录、Changes可能为修改操作(删除、匿名化等)、DicomIdentifiers为DICOM文件标示符(各级UID)、ExportedResources可能为导出或上传操作、GlobalProperties应该是全局属性、MainDicomTags应该是Orthanc返回给REST API操作的JSON格式数据、Metadata是数据体、Resources应该是文件体标记(PatientRecyclingOrder暂时不清楚,请看下文分析)。
2)Orthanc主要数据操作类介绍:
Orthanc源码中有DatabaseWrapper类,其中有如下注释:
/** * This class manages an instance of the Orthanc SQLite database. It * translates low-level requests into SQL statements. Mutual * exclusion MUST be implemented at a higher level. **/
说明该类是Orthanc操作SQLite数据库的封装类,具体的涉及到SQLite数据库底层的操作都由DatabaseWrapper来完成。与上节看到的index中的表对比,将DatabaseWrapper类主要函数分类:
| 数据表 | DatabaseWrapper操作函数 |
| AttachedFiles | AddAttachment DeleteAttachment LookupAttachment ListAvailableAttachments |
| Resources | CreateResource DeleteResource GetResourceType GetResourceCount LookupResource |
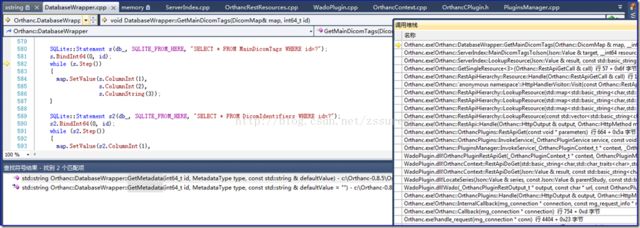
| Metadata | DeleteMetadata GetAllMetadata GetMetadata GetMetadataAsInteger LookupMetadata SetMetadata |
另外还会看到众多获取各表字段的函数,例如GetPublicId、GetChildrenPublicId等等。
Orthanc中SQLite实例测试:
在大致了解了Orthanc中SQLite数据库的基本结构后,进行一下实例测试。如博文(http://blog.csdn.net/zssureqh/article/details/41836885)所述,向Orthanc中添加数据有多种方式,命令行工具,REST API,以及网页。下面我们对Orthanc自带的Explorer和DCMTK工具包storescu.exe进行真实数据上传测试。
SQLite数据写入逻辑实例测试
1)Explorer中Drag & Drop测试:
先打开Orthanc的浏览界面:http://localhost:8042/app/explorer.html#upload
拖拽任意图像到浏览器内,单击【Start the upload】,直到出现绿色'【Done】,表明上传成功。
数据库变化如下:
2)storescu.exe测试:
上述利用Orthanc内嵌的Explorer成功上传并写入数据库。此次使用storescu.exe,把Orthanc当做Dicom Server查看数据写入情况,写入指令如下:
storescu.exe -d localhost 4242 -aet ZSSURE -aec ORTHANC c:\test2.dcm
完成后数据库变化如下:
SQLite查询逻辑测试:
上面利用两种方式来完成了添加数据到Orthanc内嵌SQLite数据库(还有REST API第三种方式,参见之前博文:,由于原理与Explorer中类同就不单独介绍了),并且观察到了数据库的真实变化,但是具体的字段含义此刻可能还不是很清楚,让我们利用REST API来读取数据库并尝试分析下其中的含义。
1)Patients:
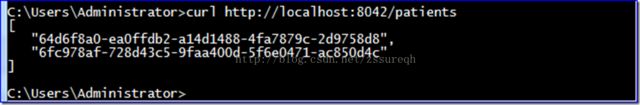
curl http://localhost:8042/patients
返回结果如上图所示,通过对比上一节中观察到的数据库变化发现:返回的两个Patient UUID分别记录在Resources表中PublicId列的第4与8行,其对应的internalId分别为44和48。因此我们可以推断出Resources中应该是我们上传文件的记录,下面来验证一下我们的猜想。
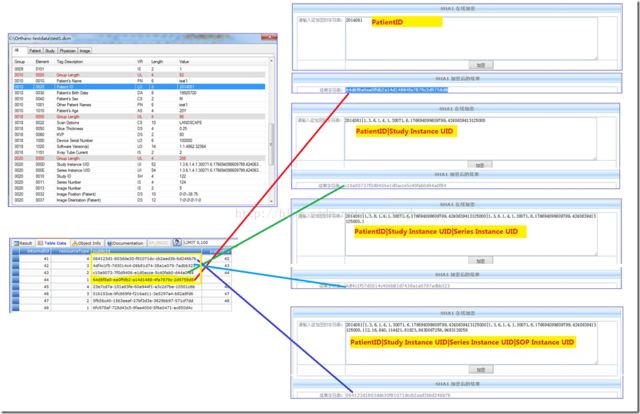
根据上一节分析指导此处的publicId应该是DICOM UID对应的UUID,即SHA1计算值。打开在线计算SHA1网站:http://www.seacha.com/tools/sha1.html。按照上一节分析输入test1.dcm的各级UID,计算结果如下所示:
从图中我们可以看出在Resources表中的前四条记录按照级别深度分别存储的是InstanceUUID、SeriesUUID、StudyUUID、PatientUUID,这些UUID是由DICOM 各级UID进行SHA1计算所得。有兴趣的话可以验证一下后四条记录,自然也是相同的含义。至此我们搞清楚了Resources表的意义,是用于存储DICOM图像的UUID
2)Studies:
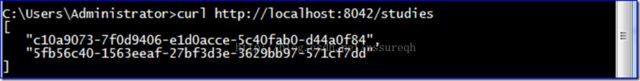
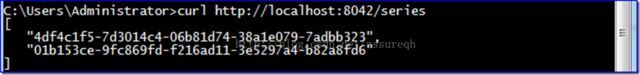
curl http://localhost:8042/studies
返回结果为,
即上述分析的Resources表中的每组的第三条记录,也就是表中的43和47行。
3)Series:
curl http://localhost:8042/series
返回结果为,
Resources表中每组记录的第二条,表中的42和46行。
4)Instances:
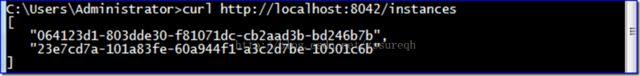
curl http://localhost:8042/instances
返回结果为,
Resources表中每组记录的第一条,表中的41和45行。
5)查看每个Patient内容:
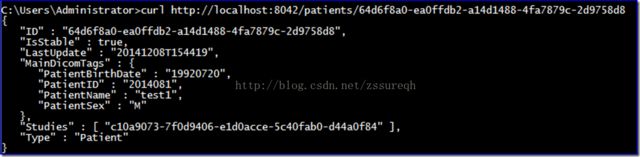
curl http://localhost:8042/patients/64d6f8a0-ea0ffdb2-a14d1488-4fa7879c-2d9758d8
对比前面数据库的分析,发现大多数字段都可以直接在数据库中看到对应的值,如下图所示:
6)查看具体Instance内容
因为查看Study和Series级别的内容与查看Patient级别类似,就不啰嗦了,直接看一下具体Instance(即DICOM文件)的查询结果,输入指令:
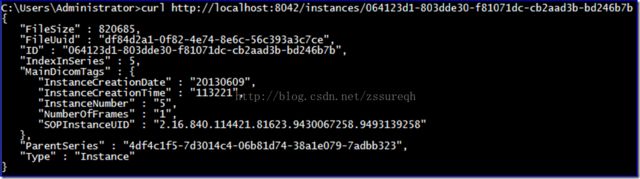
curl http://localhost:8042/instances/064123d1-803dde30-f81071dc-cb2aad3b-bd246b7b
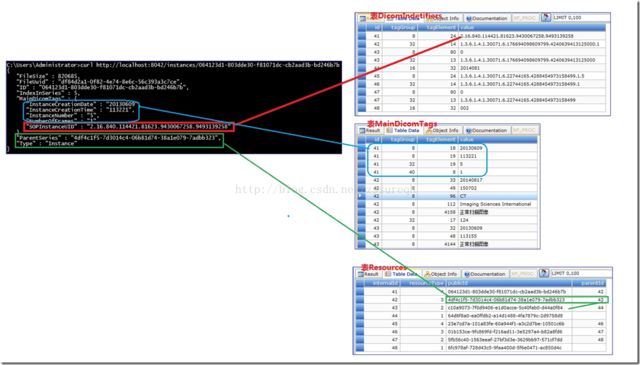
上述结果在数据库中都可以直接找到,如下图所示:
至此我们看到了熟悉的【SOP Instance UID】,原来存储在DicomIdentifiers表中。
从上述的多次实例测试我们也大致猜出来Orthanc SQLite数据库中各表的作用,Resources表中是利用SHA1来计算出UUID唯一标识我们的DCM文件;DicomIdentifiers表记录的是对应DCM文件的各级DICOM UID,想必这也是WADO协议中需要定位文件的必要参数;MainDicomTags表存储的是对应DCM文件的主要几种Tag,包括Group号、Element号,以及值域数据。各个表之间的关联是通过Resources表中的internalId来完成的,internalId是大多数表的主键(PK)。
到这里本文就可以结束了,已经达到了剖析Orthanc SQLite的目的,但是还并未清晰的看出REST API与WADO的区别。为此,也为了更好的了解Orthanc的操作流程,再补充一节,通过单步调试来深入分析一下Orthanc的实现机制,达到深入剖析的境界。
Orthanc SQLite总结:
前一篇博文中对Orthanc官方给出的Plugin SDK开发文档进行了简短的翻译,文档中指出在0.8.0版本之前,Orthanc是利用内建的RESTful API来模拟是实现WADO服务的,并非是直接响应浏览器发送过来的WADO请求。前文中已经介绍了如何具体编译和安装官方WadoPlugin.dll,这里在剖析SQLite的基础上采用单步调试的方式查看一下早期Orthanc是如何利用RESTful API来模拟实现WADO服务的。
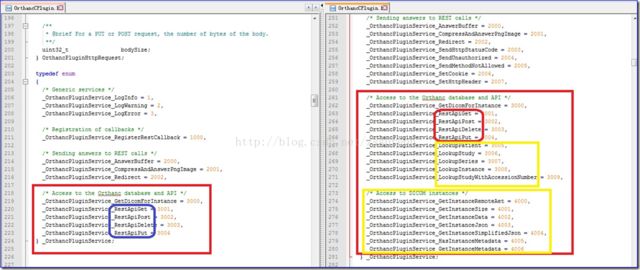
RESTful API模拟WADO
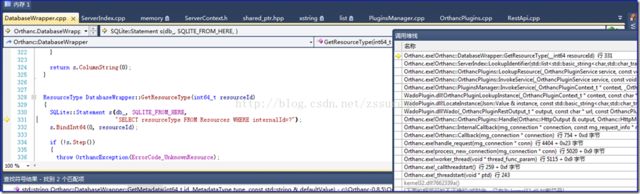
官网给出的利用内建RESTful API仿真WADO的代码在WadoPlugin.cpp中的Wado函数内,其中最主要的是LocateStudy、LocateSeries和LocateInstance三个定位函数。下图是LocateStudy级别的单步调试结果:
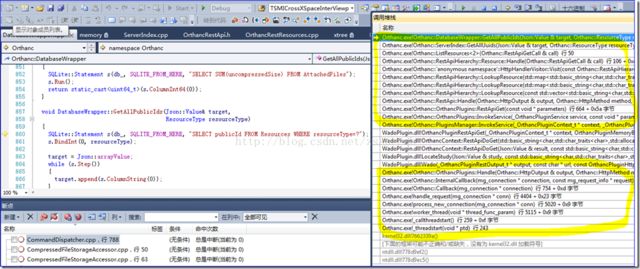
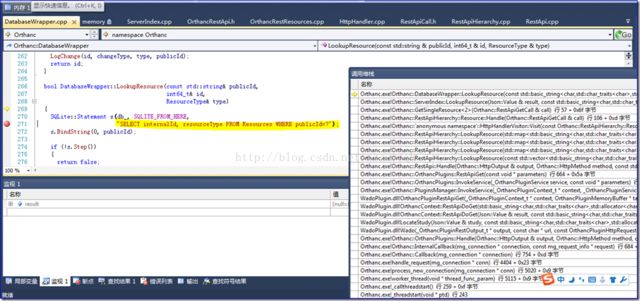
从上图可以看出在LocateStudy函数内部,首先是利用DatabaseWrapper.cpp中的GetAllPublicId函数从SQLite数据库的Resources表中提取出全部的publicId,如我们上面分析,每一个上传的文件都有唯一对应的UUID格式的publicId。
随后,在LocateStudy函数内部,对前面返回的所有publicId进行循环遍历,针对每一个/studies/{publicId}进行资源定位,用到的函数是LookupResource(同样在DatabaseWrapper.cpp中)。通过下图中可以看出该函数从Resources表中根据publicId查询出internalId和resourceType两个字段。查看LookupResource函数参数type的类型ResourceType定义可知:Resources表中第二列字段存储的是publicId对应的资源级别,该级别按照DICOM3.0标准划分为Patient(=1)、Study(=2)、Series(=3)、Instance(=4)四级,如Enumeration.h中定义所示:
enum ResourceType
{
ResourceType_Patient = 1,
ResourceType_Study = 2,
ResourceType_Series = 3,
ResourceType_Instance = 4
};
下面直接贴出调试的截图:
从截图中可以看出Orthanc中响应WADO请求的大致数据库检索流程,首先是在Resources表中查询所有的publicId(因为初次查询无法利用WADO请求中的studyID/seriesID/objectID计算出任何有效UUID);然后构造/studies/{id}形式的uri,利用RESTful API机制查询组合出各个级别的publicId,其各级之间的关系由表Resources中的parentId字段标明,而唯一性由主键internalId来决定。这也就是上述多次发起RESTful API查询数据库的主要原因;待获得了各级publicId和internalId后,就是从DicomIdentifiers表、MainDicomTags表和Metadata表中提取DICOM文件关键信息操作;最后自然就是将查询到的结果图像返回到浏览器端(可以DICOM格式或JPEG缩略图形式返回)。
【注】:在表Metadata中记录的type由Enumerations.h文件给出定义,如下:
enum MetadataType
{
MetadataType_Instance_IndexInSeries = 1,
MetadataType_Instance_ReceptionDate = 2,
MetadataType_Instance_RemoteAet = 3,
MetadataType_Series_ExpectedNumberOfInstances = 4,
MetadataType_ModifiedFrom = 5,
MetadataType_AnonymizedFrom = 6,
MetadataType_LastUpdate = 7,
// Make sure that the value "65535" can be stored into this enumeration
MetadataType_StartUser = 1024,
MetadataType_EndUser = 65535
};
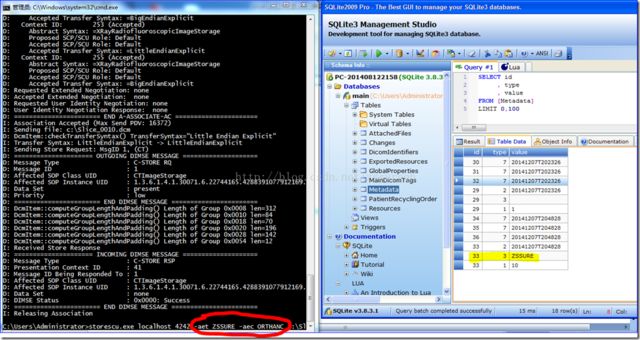
可以发现其中有RemoteAet类型,因此猜测可能跟DICOM 协议有关,用于记录上传端的AE Title,通过输入指令验证如下:
指令:storescu.exe -d localhost 4242 -aet ZSSURE -aec ORTHANC c:\Slice_0010.dcm
测试结果:
直接实现WADO
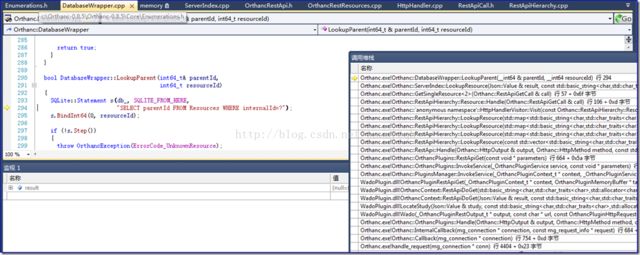
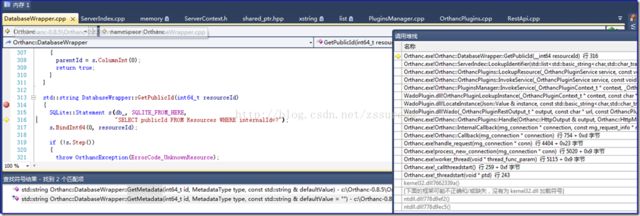
在分析了原有的效率较低的WadoPlugin查询方式后,我们按照同样的方式单步调试,查看新的Orthanc PluginSDK的查询过程。具体截图如下:
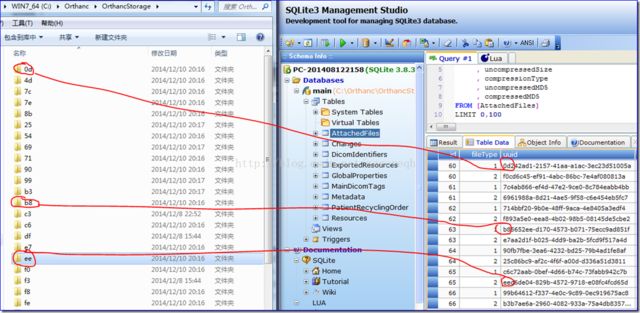
上述系列截图可以看出新的Orthanc Plugin SDK通过三步可以轻松从SQLite数据库中读取指定Instance的publicId(即上文说的UUID);获得了InstanceUUID后构造/instances/{id}类型的RESTful API uri来直接获取Orthanc数据库中的文件信息。如是减少了循环查询数据库的次数,提升了效率。仔细分析下来可以发现之所以原本的PluginSDK需要查询多次数据库是因为Orthanc中将DICOM文件及相关信息按照不同级别将信息分类存储,因此提取时需要分别定位然后将查询结果组合。另外打开Orthanc的Storage目录可以发现对于每个DCM文件Orthanc采用了publicId的两级目录方式来存储:第一级目录是文件的MD5值中的第一部分的前2个字节;第二级是后两个字节。如下图所示:
至此可以清楚地了解了Orthanc底层SQLite数据库的结构及相关操作,为了兼容RESTful API和DICOM3.0标准,数据库的逻辑设计是很精妙的,后续可深入研究一下。
后续专栏博文介绍
fo-dicom搭建简单的DICOM Server服务器
时间:2014-12-10