3行程序搞定SVM分类-用JAVA程序调用LibSVM API 最简单的示例
最近用LibSVM做分类实验,发现网上的资料大都是关于SVM基本原理和怎么用终端命令行使用svm-train,svm-predict等几个程序,这些其实在README文件中已经讲得很清楚了,Readme里面几乎包含了所有可以帮助你灵活使用LibSVM的信息,可以很多人都不怎么看。这里给出用JAVA调用LibSVM API最简单的示例,用JAVA进行SVM分类只需要几行程序就搞定了,前提是你已经准备好了符合LibSVM处理数据格式的训练样本和测试样本。下面一一道来(注:有网友向我要eclipse工程,下载地址见点击打开链接)
1、 准备训练样本和测试样本
直接上LibSVM官网就可以下载,我下载的UCI的UCI-breast-cancer数据集,训练样本和测试样本的基本格式是这样的
<label> <index1>:<value1> <index2>:<value2> ...
分别代表 类别 feature1索引:feature1值 feature2索引:feature2值
如果想自己定义核函数,那么属于precompute kernel values and input them as training and testing files.
此时训练样本和测试样本变成如下格式:
New training instance for xi: <label> 0:i 1:K(xi,x1) ... L:K(xi,xL)
New testing instance for any x: <label> 0:? 1:K(x,x1) ... L:K(x,xL)
That is, in the training file the first column must be the "ID" of xi. In testing, ? can be any value.
很好理解,有篇EMNLP2009做观点和关系抽取就是自己定义核函数借助SVM分类器实现的。
2、建立JAVA工程,导入LibSVM 的JAR包,要注意还需要导入java文件下的svm_train.java和svm_predict.java这两个文件,这两个类其实主要在LibSVM基础上做了进一步封装,把命令行参数转化成了String []类型的函数参数,方便API调用。至于另外两个svm_tony.java和svm_scale可以不导入,它们分别是图形界面和数据压缩用的,不是必要文件。
3、把训练样本和测试样本放在工程文件夹下,当然,你也可以自定义data目录。
4 、修改svm_train.java和svm_predict.java这两个文件,前者主要是把model_file_name返回,因为在svm_predict的main函数中需要使用,后者主要是把分类的Accuracy返回。
5、编写JAVA调用LibSVM API分类代码如下,非常简单,代码中给出了注释
- import java.io.IOException;
- import libsvm.*;
- /**JAVA test code for LibSVM
- * @author yangliu
- * @blog http://blog.csdn.net/yangliuy
- * @mail [email protected]
- */
- public class LibSVMTest {
- public static void main(String[] args) throws IOException {
- // TODO Auto-generated method stub
- //Test for svm_train and svm_predict
- //svm_train:
- // param: String[], parse result of command line parameter of svm-train
- // return: String, the directory of modelFile
- //svm_predect:
- // param: String[], parse result of command line parameter of svm-predict, including the modelfile
- // return: Double, the accuracy of SVM classification
- String[] trainArgs = {"UCI-breast-cancer-tra"};//directory of training file
- String modelFile = svm_train.main(trainArgs);
- String[] testArgs = {"UCI-breast-cancer-test", modelFile, "UCI-breast-cancer-result"};//directory of test file, model file, result file
- Double accuracy = svm_predict.main(testArgs);
- System.out.println("SVM Classification is done! The accuracy is " + accuracy);
- //Test for cross validation
- //String[] crossValidationTrainArgs = {"-v", "10", "UCI-breast-cancer-tra"};// 10 fold cross validation
- //modelFile = svm_train.main(crossValidationTrainArgs);
- //System.out.print("Cross validation is done! The modelFile is " + modelFile);
- }
- }
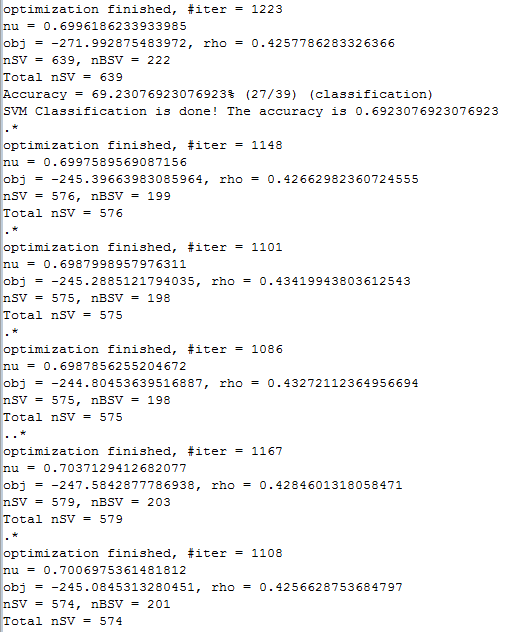
6 运行工程,观察分类准确率,我的代码主要进行了两次实验,第一次是基于训练样本训练分类model(分类超平面),然后对测试样本进行分类;第二次直接对训练样本采用10折交叉验证法测试SVM分类性能,其他参数采用默认值,运行结果如下
是不是很容易,其实仔细看看LibSVM的Readme,很多问题都不是问题。如果需要完整工程文件可以在下面评论栏留下邮箱。
附:svm 主调用程序经常使用的几个方法,注意这是LibSVM中SVM类下的函数,与svm_train.java等文件中的相关函数要区分:
svm.svm_train(svm_problem,svm_parameter) 该方法返回一个训练好的svm_model
svm.svm_load_model(文件名); 该方法返回一个训练好的svm_model
svm.svm_save_model(文件名,svm_model); 该方法将svm_model保存到文件中
svm.svm_predict_values(svm_model,svm_node,double); 该方法返回doule类值,svm_node对svm_model测试,返回值确定了svm_node在模型中的定位
原文:http://blog.csdn.net/yangliuy/article/details/8041343