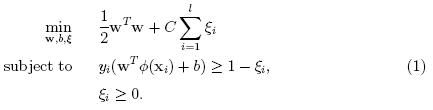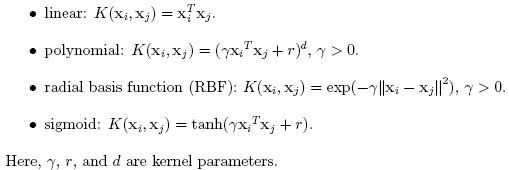LibSVM分类的实用指南
译者注:简单翻译了台湾林智仁教授的文章《A Practical Guide to Support Vector Classification》,未经作者同意,没有版权;同时翻译仅供自己学习之用,不严谨,有错误,还请大家指出。
原文地址:http://www.csie.ntu.edu.tw/~cjlin/。译者博客:http://blog.sina.com.cn/netreview。
摘要
SVM(support vector machine)是一项流行的分类技术。然而,初学者由于不熟悉SVM,常常得不到满意的结果,原因在于丢失了一些简单但是非常必要的步骤。在这篇文档中,我们给出了一个简单的操作流程,得到合理的结果。(译者注:本文中大部分SVM实际指的是LibSVM)
1 入门知识
SVM是一项非常实用的数据分类技术。虽然SVM比起神经网络(Neural Networks)要相对容易一些,但对于不熟悉该方法的用户而言,开始阶段通常很难得到满意的结果。这里,我们给出了一份指南,根据它可以得到合理结果。
需要注意,此指南不适用SVM的研究者,并且也不保证一定能够获得最高精度结果。同时,我们也没有打算要解决有挑战性的或者非常复杂的问题。我们的目的,仅在于给初学者提供快速获得可接受结果的秘诀。
虽然用户不是一定要深入理解SVM背后的理论,但为了后文解释操作过程,我们还是先给出必要的基础的介绍。一项分类任务通常将数据划分成训练集和测试集。训练集的每个实例,包含一个“目标值(target value)”(例如,分类标注)和一些“属性(attribute)”(例如,特征或者观测变量)。SVM的目标是基于训练数据产出一个模型(model),用来预测只给出属性的测试数据的目标值。
给定一个训练集,“实例-标注”对,![]() ,支持向量机需要解决如下的优化问题:
,支持向量机需要解决如下的优化问题:
在这里,训练向量xi通过函数Φ被映射到一个更高维(甚至有可能无穷维)空间。SVM在这个高维空间里寻找一个线性的最大间隔的超平面。C>0是分错项的惩罚因子(penalty parameter of the error term)。被称之为核函数(kernel function)。新的核函数还在研究中,初学者可以在SVM书中找到如下四个最基本的核函数:(线性、多项式、径向基函数、S型)
1.1 实例
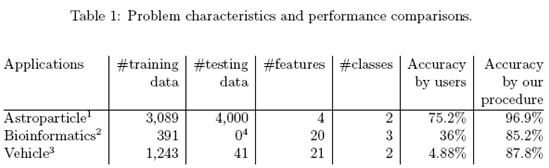
表1是一些现实生活中的实例。这些数据集是由我们的用户提供的,其开始时无法获得理想精度的结果。使用了本指南描述的过程后,我们帮助他们获得了更好的性能。
这些数据集都在:http://www.csie.ntu.edu.tw/~cjlin/papers/guide/data/
1.2 建议流程
许多初学者使用如下的步骤:
· 将数据转换成SVM程序包的格式
· 随机的尝试一些核函数和参数
· 测试
而我们建议初学者先尝试如下的步骤:
· 将数据转换成SVM格式包的格式
· 对数据进行简单的缩放处理(scaling)
· 考虑RBF核:![]()
· 使用交叉验证(cross-validation)寻找最佳参数C和Υ
· 使用最佳参数C和Υ来训练整个训练集
· 测试
值得一提的是,最佳参数是受数据集的大小影响的,但在实践中,从交叉验证中获得的最佳参数已经适用于整个训练集。
后面章节,我们将具体探讨这些步骤。
2 数据预处理
2.1 类别特征
SVM需要每个实例的特征集,是用实数向量表示。因此,如果存在类别属性,我们首先将它们转变成数值。推荐使用m个数值特征来表示m类的属性。每个数值表示其中一个类别为1,其它类为0。例如,一个三类属性{red, green, blue},可以表示成{0,0,1},{0,1,0},{1,0,0}。我们的经验表明,如果属性的数值不是太大,这样分解成多特征比直接将类别属性当做数值使用的效果要稳定。
2.2 缩放
应用SVM之前,缩放是非常重要的。Sarle的神经网络FAQ的第二部分(1997)阐述了缩放的重要性,大多数注意事项也适用于SVM。缩放的最主要优点是能够避免大数值区间的属性过分支配了小数值区间的属性。另一个优点能避免计算过程中数值复杂度。因为关键值通常依赖特征向量的内积(inner products),例如,线性核和多项式核力,属性的大数值可能会导致数值问题。我们推荐将每个属性线性缩放到区间[-1,+1]或者[0, 1]。
当然,我们必须使用同样的方法缩放训练数据和测试数据。例如,假设我们把训练数据的第一个属性从[-10, +10]缩放到[-1, +1],那么如果测试数据的第一个属性属于区间[-11, +8],我们必须将测试数据转变成[-1.1, +0.8]。附录B中有一个实例可参考。
3 模型选择
虽然章节一中只有四个常用核函数,但我们必须决定哪一个是首选。然后是惩罚因子C和核参数的选择。
3.1 RBF核
通常而言,RBF核是合理的首选。这个核函数将样本非线性地映射到一个更高维的空间,与线性核不同,它能够处理分类标注和属性的非线性关系。并且,线性核是RBF的一个特例(Keerthi and Lin 2003),因此,使用一个惩罚因子C的线性核与某些参数(C,γ)的RBF核具有相同的性能。同时,Sigmoid核的表现很像一定参数的RBF核(Lin and Link 2003)。
第二个原因,超参数(hyperparameter)的数量会影响到模型选择的复杂度(因为参数只能靠试验呀!)。多项式核比RBF核有更多的超参数。
最后,RBF核有更少的数值复杂度(numerical difficulties)。一个关键点0<Kij<=1对比多项式核,后者关键值需要 infinity(rxiTxj+r>1)或者zero(rxiTxj+r<1),这是高阶运算。此外,我们必须指出sigmoid核在某些参数下不是合法的(例如,不是两个向量的内积)。(Vapnik 1995)
当然,也存在一些情形RBF核是不适用的。特别地,当特征维数非常大的时候,很可能只能适用线性核。在附录C中有详细探讨。
3.2 交叉验证和网格搜索
RBF核有两个参数:C和γ。对于给定的问题,我们无法事先知道哪个C和γ是最佳的;因此,一些模型选择(参数搜索)是必不可少的。目标是确定good(C,γ),使得分类器能够精确地预测未知数据(例如,测试数据)。然而这不一定对获得高准确率训练有好处。如上讨论,通常的做法是将数据集合划分成两部分,其中一部分假设是未知分类的。从“未知”数据集上获得的预测准确率可以更精确地反映出分类器在独立数据集合上的性能/效果。这种做法的一个改进版本就是交叉验证(cross-validation)。
在v折交叉验证(v-fold cross-validation)中,我们首先将训练集合划分成相同大小的v个子集。然后将其中一个子集作为测试集,其他v-1个子集作为训练集训练分类器。如此,整个训练集中的每个实例都会被预测一次,因此,交叉验证的准确率等于能够被正确分类的数量百分比。
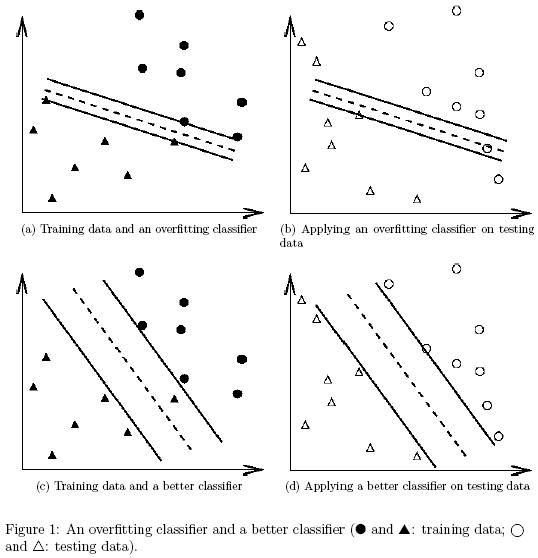
交叉验证的方法能够避免过拟合(overfitting)问题。图1通过一个二分类问题来说明这个问题。实心圆和三角是训练数据,而空心圆和三角是测试数据。在图1a和1b中的分类器的测试准确率不好就是因为它对训练数据过拟合了。如果我们考虑将图1a和1b的训练和测试数据分别作为交叉验证的训练和校验集合,显然这个准确率是不好的。另一方面,在图1c和1d的分类器则没有对训练数据过拟合,从而能够得到更好的测试准确率和交叉验证的准确率。
在使用交叉验证的方法确定参数C和γ时,我们推荐一种“网格搜索”(grid-search)。不同的参数值对(C,γ)被试验着,其中一个能够得到最高的交叉验证准确率。我们发现使用一些指数增长序列的C和γ是一个确定好参数的很实用技巧(例如,C=2-5,2-3,...,215, γ=2-15,2-13,...,23)。
网格搜索是一个简单且朴素的方法。事实上,有其他更高级的方法可以节省计算成本,例如近似逼近交叉验证率(approximating the cross-validation rate)。然而,有两个原因让我们更倾向于这个简单的网格搜索方法。
一方面,在心理上,我们会觉得通过近似法或者启发式而不没有详细的参数搜索的方法不安全。另一方面,网格搜索寻找最佳参数的计算时间并不会比其他高级方法多很多,主要是因为只有两个参数需要确定。进一步而言,网格搜索能够很容易并行化(因为每个(C,γ)是独立的)。许多高级方法是一个迭代过程(例如walking along a path),很难并行化。
由于做一次完整的网格搜索还是挺费时的,所以我们推荐首先使用一个粗糙的网格。在确定网格中一个“更好”区域后,可以在这个区域中执行更好的网格搜索。为了更好说明,我们做了一个实验,数据集来自german from the Statlog collection(Michie et al., 1994)。在对数据集缩放之后,我们先使用一个粗糙的网格(如图2),找到最好的(C,γ)是(23,2-5),交叉验证的正确率能够达到77.5%。然后,我们在(23,2-5)临近区域进行了一次更好的网格搜索(如图3),获得了一个更好的交叉验证的正确率77.6%,参数为(23.25,2-5.25)。在最好的(C,γ)找到以后,可以在整个训练集上再次训练产出最终的分类器。
上述的方法对于上千或更多数据规模的问题上能够工作的很好。而对于超大数据集合而言,一个可行的方法是随机抽取一个子集,运行网格搜索,找到最佳区域,然后在该区域上针对数据全集进行网格搜索。
4 讨论
在某些情况下,上述方法不一定足够好,因此其他技术如特征选取可能就不可少了。而这些议题超出本指南的范畴了。我们的实验表明,这个方法在特征不是很多的情况下能够工作的很好。如果有几千维的属性,那么在使用SVM时可能就需要先选取属性的子集了。
5 附录A:推荐过程的实例
在这个附录中我们使用了一般初学者通常使用的推荐过程来比较了准确率。实验使用LIBSVM软件,处理表1中提到的三个问题。对于每个问题,我们首先给出直接训练和测试的准确率。其次,我们展示一下在是否有缩放之后的准确率差异。从2.2节中讨论的,训练数据的属性的区间必须保存,如此我们才能够在缩放了测试数据之后还能够还原。第三,给出了使用了推荐步骤(包括缩放和模型选择)后的准确率。最后,我们示范了一下使用LIBSVM的工具自动处理整个过程。注意,一个类似的参数选择工具grid.py在R-LIBSVM中也是可利用的。
5.1 天文粒子物理 (Astroparticle Physics)
原始数据集,使用缺省参数:
$ ./svm-train svmguide1
$ ./svm-predict svmguide1.t svmguide1.model svmguide1.t.predict
àAccuracy = 66.925%
缩放数据集,只使用缺省参数:
$ ./svm-scale -l -1 -u 1 -s range1 svmguide1 > svmguide1.scale
$ ./svm-scale -r range1 svmguide1.t > svmguide1.t.scale
$ ./svm-train svmguide1.scale
$ ./svm-predict svmguide1.t.scale svmguide1.scale.model svmguide1.t.predict
àAccuracy = 96.15%
缩放数据集并且进行参数选择(tools目录下有grid.py):
$ python grid.py svmguide1.scale
…
2.0 2.0 96.8922
(Best: C=2.0, γ=2.0 with five-fold cross-validation rate=96.8922%)
$ ./svm-train -c 2 -g 2 svmguide1.scale
$ ./svm-predict svmguide1.t.scale svmguide1.scale.model svmguide1.t.predict
àAccuracy = 96.875%
使用自动脚本:
$ python easy.py svmguide1 svmguide1.t
Scaling training data...
Cross validation...
Best c=2.0, g=2.0
Training...
Scaling testing data...
Testing...
Accuracy = 96.875% (3875/4000) (classification)
5.2 生物信息学(Bioinformatics)
原始数据集使用缺省参数:
$ ./svm-train -v 5 svmguide2
àCross Validation Accuracy = 56.5217%
缩放数据集只使用缺省参数:
$ ./svm-scale -l -1 -u 1 svmguide2 > svmguide2.scale
$ ./svm-train -v 5 svmguide2.scale
àCross Validation Accuracy = 78.5166%
缩放数据集且使用参数选择:
$ python grid.py svmguide2.scale
…
2.0 0.5 85.1662
àCross Validation Accuracy = 85.1662%
(Best C=2.0, γ=0.5 with five fold cross-validation rate=85.1662%)
使用自动脚本:
$ python easy.py svmguide2
Scaling training data...
Cross validation...
Best c=2.0, g=0.5
Training...
5.3 交通工具(Vehicle)
原始数据集,使用缺省参数:
$ ./svm-train svmguide3
$ ./svm-predict svmguide3.t svmguide3.model svmguide3.t.predict
àAccuracy = 2.43902%
缩放数据集,只使用缺省参数:
$ ./svm-scale -l -1 -u 1 -s range3 svmguide3 > svmguide3.scale
$ ./svm-scale -r range3 svmguide3.t > svmguide3.t.scale
$ ./svm-train svmguide3.scale
$ ./svm-predict svmguide3.t.scale svmguide3.scale.model svmguide3.t.predict
àAccuracy = 12.1951%
缩放数据集,并使用参数选择:
$ python grid.py svmguide3.scale
…
128.0 0.125 84.8753
(Best C=128.0, γ=0.125 with five-fold cross-validation rate=84.8753%)
$ ./svm-train -c 128 -g 0.125 svmguide3.scale
$ ./svm-predict svmguide3.t.scale svmguide3.scale.model svmguide3.t.predict
! Accuracy = 87.8049%
使用自动脚本:
$ python easy.py svmguide3 svmguide3.t
Scaling training data...
Cross validation...
Best c=128.0, g=0.125
Training...
Scaling testing data...
Testing...
Accuracy = 87.8049% (36/41) (classification)
6 附录B:缩放训练和测试数据时的常见错误
第2.2节中已经强调了对训练数据和测试数据要使用相同缩放比例因子的重要性。这里我们给出一个实际例子:交通信号灯的分类问题。数据可以在LIBSVM中获取。
如果训练集合测试集被分别缩放到[0,1]区间,则结果的准确率低于70%。
$ ../svm-scale -l 0 svmguide4 > svmguide4.scale
$ ../svm-scale -l 0 svmguide4.t > svmguide4.t.scale
$ python easy.py svmguide4.scale svmguide4.t.scale
Accuracy = 69.2308% (216/312) (classification)
而对训练集和测试集使用相同的缩放比例因子,我们能够获得高的多的准确率。
$ ../svm-scale -l 0 -s range4 svmguide4 > svmguide4.scale
$ ../svm-scale -r range4 svmguide4.t > svmguide4.t.scale
$ python easy.py svmguide4.scale svmguide4.t.scale
Accuracy = 89.4231% (279/312) (classification)
使用正确的设置,svmguide4.t.scale中的10个特征的最大值如下:
0.7402, 0.4421, 0.6291, 0.8583, 0.5385, 0.7407, 0.3982, 1.0000, 0.8218, 0.9874
显然,前一个方法对测试集的缩放到[0,1]生成的是一个错误数据集。
7 附录C:何时使用线性核而不是RBF核
如果特征的数量很大,一种方法是没有必要将数据集映射到更高维空间。也就是说,非线性映射并不能够提升性能。使用线性核(linear kernel)已经足够好了,并且只需要搜索试验出一个参数C即可。3.1节中描述的RBF核能够至少和线性核一样好的观点是建立在已经搜索到合适的参数(C, γ)空间。
接下来,我们分成3部分进行探讨。
7.1 实例数 << 特征数
生物信息学中的许多微阵列数据都属于这类型。我们以一份数据为例:白血病数据( http://www.csie.ntu.edu.tw/~cjlin/libsvmtools/datasets )。其中训练集和测试集分别有38、34个实例,特征有7129个,远大于实例数。我们合并了两个文件,分别使用RBF核和线性核,来比较了交叉校验的准确率。
RBF核,带有参数选择过程:
$ cat leu leu.t > leu.combined
$ python grid.py leu.combined
…
8.0 3.0517578125e-05 97.2222
(Best C=8.0, γ= 0:000030518 with five-fold cross-validation rate=97.2222%)
线性核,带有参数选择过程:
$ python grid.py -log2c -1,2,1 -log2g 1,1,1 -t 0 leu.combined
…
0.5 2.0 98.6111
(Best C=0.5 with five-fold cross-validation rate=98.61111%)
虽然grid.py是为RBF核设计的,但上面的方法也可以对线性核进行不同C的检测(-log2g 1,1,1实际上是设置了一个虚拟的γ)。
可以看到,使用线性核和RBF核其交叉验证的准确率是相当的。显然,当特征数非常大时,其中一种方式就是无需再映射数据了。
除了LIBSVM,在这个实例中,下文提到的LIBLINEAR也是非常有效的工具。
7.2 实例数和特征数都很大
这类数据通常出现在文本分类中。LIBSVM对这类问题不是非常好用。幸运的是,我们有另一款工具LIBLINEAR,非常适合这类数据。我们举例说明在文本分类中LIBSVM和LIBLINEAR的差异,数据集来自LIBSVM中的rcv1_train.binary。实例数有20242,特征数有47236个。
$ time libsvm-2.85/svm-train -c 4 -t 0 -e 0.1 -m 800 -v 5 rcv1_train.binary
Cross Validation Accuracy = 96.8136%
345.569s
$ time liblinear-1.21/train -c 4 -e 0.1 -v 5 rcv1_train.binary
Cross Validation Accuracy = 97.0161%
2.944s
使用5折交叉验证,LIBSVM花费了350秒,而LIBLINEAR只用了3秒。而且,LIBSVM花费了更多的内存,因为我们分配了一些空间用于存储最近使用的核元素(参照-m 800)。显而易见,在生成可比的准确率的模型时,LIBLINEAR比LIBSVM快的多。
LIBLINEAR对于大规模文本分类问题非常高效。让我们实验一个有677399个实例的大数据集合rcv1_test.binary。
$ time liblinear-1.21/train -c 0.25 -v 5 rcv1_test.binary
Cross Validation Accuracy = 97.8538%
68.84s
注意读取数据就需要花费很多时间。而划分后的训练/校验时间是小于4秒的。
7.3 实例数 >> 特征数
当特征数非常少,一种方式是将数据映射到更高维的空间上(例如使用非线性核)。然而,如果你实在想要使用线性核,那么你可以使用LIBLINEAR,选项“-s 2”。当特征数很少时,这个选项一般要比缺省(default)项“-s 1”快的多。数据实例:http://www.csie.ntu.edu.tw/~cjlin/libsvmtools/datasets/binary/covtype.libsvm.binary.scale.bz2 。实例数有581012个,远大于特征数54个。我们分别使用两个选项运行LIBLINEAR:
$ time liblinear-1.21/train -c 4 -v 5 -s 2 covtype.libsvm.binary.scale
Cross Validation Accuracy = 75.67%
67.224s
$ time liblinear-1.21/train -c 4 -v 5 -s 1 covtype.libsvm.binary.scale
Cross Validation Accuracy = 75.6711%
452.736s
显然使用“-s 2”选项训练时间可以短很多。
转载请注明出处:互联网旁观者~黄言之 http://blog.sina.com.cn/netreview/