机器学习算法笔记:谱聚类方法
这方法是昨天听同学提起的,大致翻看了几篇博客跟论文,这里写下自己的理解
从样本相似性到图
根据我们一般的理解,聚类是将相似的样本归为一类,或者说使得同类样本相似度尽量高,异类样本相似性尽量低。无论如何,我们需要一个方式度量样本间的相似性。常用的方式就是引入各种度量,如欧氏距离、余弦相似度、高斯度量等等。
度量的选择提现了你对样本或者业务的理解。比如说如果你要比较两个用户对音乐选择的品味,考虑到有些用户习惯打高分,有些用户习惯打低分,那么选择余弦相似度可能会比欧式距离更合理。
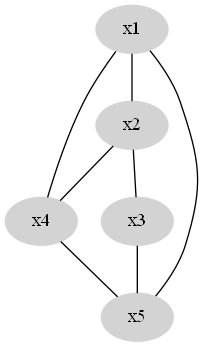
现在我们假设已有的样本为 X={x1,x2,…,xn} , 我们选择的样本相似性度量函数为 (xi,xj)→s(xi,xj) ,其中 s≥0 , s 越大表示相似性越高。一般我们选择的是距离的倒数。据此我们可以构造相似性图,节点为样本,节点之间的连边权重为样本间的相似性,如图所示。
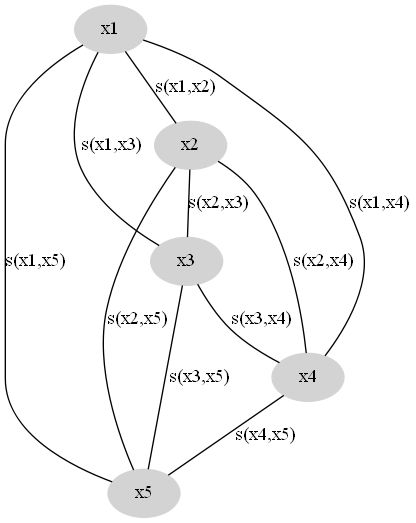
这是一个完全图,我们的目的是去掉一些边,使得这个图变成 K 个连通子图。同一个子图内的节点归为一类。因此有两方面考虑:
- 子图内的连边权重尽量大,即同类样本间尽量相似
- 去掉的边权重尽量小,即异类样本间尽量不同
一个初步的优化方法是去掉部分权重小的边,常用的有两种方式:
- ϵ 准则,即去掉权重小于 ϵ 的所有边
- k 邻近准则,即每个节点之保留权最大的几条边
图与图的Laplacian矩阵
为了下一步的算法推导,首先介绍图的Laplacian矩阵,我们记节点 i,j 连边的权重为 wi,j ,如果两个节点之间没有连边, wi,j=0 ,另外 wii=0 ,那么图对应的Laplacian矩阵为:
容易看到,矩阵 L 行和为零,且对于任意的向量 f 有
优化目标
现在我们来推导我们要优化的目标函数。前面说过,我们的目的是去掉一些边,使得这个图变成 K 个连通子图,我们还希望去掉的边权重尽量小。为此,假设我们已经把图分割成立K个连通子图 A1,⋯,AK ,那么我们去掉的边集合为
为了方便,引入记号
那么
因此去掉的边的权重和为
现在的问题就转换为:找到 X 的划分 A1,⋯,AK ,使得上式最小。不幸的是,存在两个问题:
- 这是个NP难问题,没有有效算法
- 实际实验得到的结果常常将单独的一个样本分为一类
先来解决第二个问题:
我们实际希望的是,每个类别中的样本数要充分大,有两种调整目标函数的方法:
- RatioCut,将目标函数改成
12∑k=1nW(Ak,A¯k)|Ak|
- Ncut, 将目标函数改成
12∑k=1nW(Ak,A¯k)vol(Ak)
其中 vol(A)=∑i∈Adi
两种方法都使得某个类样本量少的时候,对应的目标函数项变大。这里我们以第一种方法为例,第二种是类似的。
现在来解决第二个问题:
我们碰到NP难问题的时候,通常是考虑近似解,谱聚类也不例外。首先,我们要引入列向量 hk=(h1k,⋯,hn,k)′ ,其中
那么,
令 H=(h1,…,hK) ,则 H′H=I ,且
现在的问题是找到 H 使得 tr(H′LH) 尽量小。这当然还是NP问题。原因在于 H 的列向量元素只能选择 0 或者 |Ak|−−−−√
这里用到的一个trick是放宽 H 的限制,在更大的变量空间内寻找目标函数的最小值。在这里,我们让 H 可以取任意实数,只要满足 H′H=I .那么就是求解优化问题:
令 L=Q′ΛQ,Y=QH 则 Y′Y=I ,
由于 Y′Y=I ,有
可以分析得到 tr(H′LH) 取最小值时,必有 (YY′)ii=0 or 1 ,
因此
且当 Yk=ek 时等号成立,对应的 hk=(Q′Y)k=Q′ek=Q′k 为 L 的第 k 小特征值对应的特征向量。
最后一步
现在我们得到了放宽限制条件下的优化问题的最优解解 h1,⋯hK ,如何得到原问题的一个解?
我们知道,如果 H 满足原来的限制条件,那么 hij≠0 表示第 i 个样本归为第 j 类,但是我们放宽限制条件后得到的 H 可能一个零都没有!
谱聚类有意思的地方是选择了对 H 的行向量做K-means聚类,我个人认为是基于如下考虑:
- 对满足原始限制条件的 H ,行向量一致等价于类别一致
- 在原始限制条件下得到的 H 跟放宽限制条件下得到的 H 应该比较相近
如此可以推断在放宽条件下得到的 H 如果行向量相似,则应该类别相似。因此直接对行向量做k-means聚类即可。
总结
至此,谱聚类的大致步骤就完成了,归纳下主要步骤
- 计算样本相似性得到样本为节点的完全图
- 基于 ϵ 准则或者 m 邻近准则将完全图稀疏化
- 计算稀疏化后的图的laplacian矩阵,计算其前 K 小特征值对应的特征向量作为矩阵 H 的列
- 对矩阵 H 的行向量作k-means聚类
- 若 H 的第 i 行与第 j 行被聚为一类,则表示 xi 与 xj 聚为一类。
代码例子
import numpy as np
import networkx as nx
import scipy.linalg as llg
from Queue import PriorityQueue
import matplotlib.pylab as plt
import heapq as hp
from sklearn.cluster import k_means
# fake data from multivariate normal distribution
S = np.random.multivariate_normal([1,1], [[0.5,0],[0,0.7]],100)
T = np.random.multivariate_normal([-1,-1], [[0.3,0],[0,0.8]],100)
R = np.random.multivariate_normal([-1,0], [[0.4,0],[0,0.5]],100)
Data = np.vstack([S,T,R])
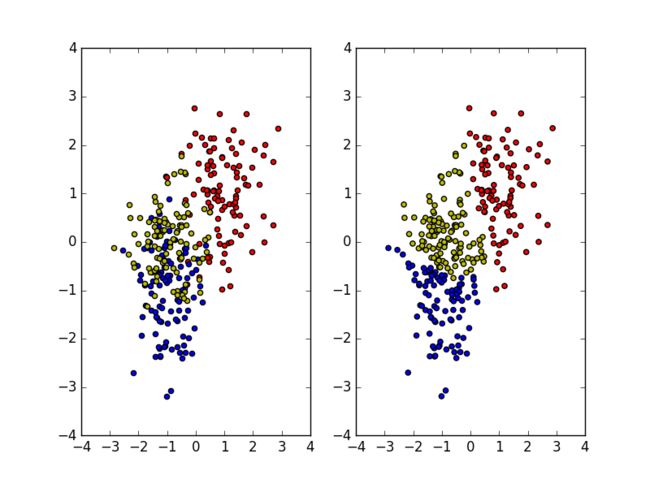
plt.subplot(1,2,1)
plt.scatter(S.T[0],S.T[1],c='r')
plt.scatter(T.T[0],T.T[1],c='b')
plt.scatter(R.T[0],R.T[1],c='y')
# calc k-nearest neighbors
def min_k(i,k):
pq = []
for j in range(len(Data)):
if i == j:
continue
if len(pq) < k:
hp.heappush( pq,(1/np.linalg.norm(Data[i]-Data[j]), j) )
else:
hp.heappushpop( pq, (1/np.linalg.norm(Data[i]-Data[j]), j) )
return pq
# calc laplacian
L = np.zeros((len(Data),len(Data)))
for i in range(len(Data)):
for (v,j) in min_k(i, 3):
L[i,j] = -v
L[j,i] = -v
L = L + np.diag(-np.sum(L,0))
# kmean
(lam, vec) = llg.eigh(L)
A = vec[:,0:3]
kmean = k_means(A,3)
plt.subplot(1,2,2)
plt.scatter(Data.T[0],Data.T[1],c=['r' if v==0 else 'b' if v==1 else 'y' for v in kmean[1]])
plt.show()