Sklearn__支持向量机
支持向量机的基本思想是找到一条"线",使得分类间距最大。
一、线性分类器(线性核)
很多时候由于数据不可能完全分为两类,所以需要设置一定范围,允许分类错误。即设置软间隔,在sklearn 中用超参数 C (惩罚系数)来控制这种平衡:C 比较小,即脾气较小,度量大容忍度高,其对于的软间隔就大;反之则小。
1. Iris数据集试验一个线性核SVM
一下为十分简单的试验
# 加载包
from sklearn import datasets
from sklearn.pipline import Pipeline
from sklearn.preprocessing import StandarScaler
from sklearn.svm import LinearSVC
def get_iris():
# 仅仅需要花瓣长宽
iris = datasets.load_iris()
return iris.data[:, (2, 3)], (iris.target == 2) * 1.0 # Iris-Virginica
def clf_correct(y_train, y_prd):
return sum((y_train - y_prd) == 0) / len(y_train)
if __name__ == '__main__':
x, y = get_iris()
svm_clf = Pipeline([
("scaler", StandardScaler()), # 实际上在算'距离',所以需要标准化
("linearsvc", LinearSVC(C=1, loss='hinge')),
])
svm_clf.fit(x, y)
print(svm_clf.predict([[5.5, 1.7]]))
# 预测
y_prd = svm_clf.predict(x)
print("当前模型的准确率为: {}".format(clf_correct(y, y_prd)))
""" result
[1.]
当前模型的准确率为: 0.9533333333333334
"""
需要注意的是,不同于Logistic回归分类,SVM 分类不会输出每个分类的概率。
当然还可以用SVC类,使用SVC(C = 1, kernel = ‘linear’) , 但是比较慢,尤其是在比较大训练,所以一般不被推荐,另外选择是使用SGDCLassifier 类,即SGDCLassifier(loss = 'hinge', alpha = 1/(m * c))
它应用了随机梯度下降来训练一个线性SVM分类器。尽管它不会和LinearSVC一样快速收敛,但是对于处理那些不适合放在内存的大数据集非常有用,或者在线分类任务同样有用。
二、非线性核
1. 多项式特征变化
一个简单例子
from sklearn.preprocessing import PolynomialFeatures # 多项式特征变换
import matplotlib.pyplot as plt
plt.style.use('ggplot')
from sklearn.metrics import confusion_matrix
def plot_moom(m_x, m_y):
x_1 = [ m_x[i][0] for i in range(len(m_x))]
x_2 = [ m_x[i][1] for i in range(len(m_x))]
plt.scatter(x_1, x_2, c = m_y)
if __name__ == '__main__' :
make_moons = datasets.make_moons()
m_x = make_moons[0]
m_y = make_moons[1]
poly_svm_clf = Pipeline([
('poly_features', PolynomialFeatures(degree=3)),
('scaler', StandardScaler()),
('svm_clf', LinearSVC(C=10, loss='hinge'))
])
poly_svm_clf.fit(m_x, m_y)
# 预测
y_prd = poly_svm_clf.predict(m_x)
print("当前模型的准确率为: {}".format(clf_correct(m_y, y_prd)))
## 评测
print(confusion_matrix(m_y, y_prd))
plot_moom(m_x, m_y)
plot_moom(m_x, y_prd)
plt.show()
""" result
当前模型的准确率为: 1.0
[[50 0]
[ 0 50]]
"""
2. 核方法
from sklearn.svm import SVC
if __name__ == '__main__' :
poly_kernel_svm_clf = Pipeline([
('scaler', StandardScaler()),
('svm_clf', SVC(kernel='poly', degree=3, C=5, coef0=1)) ##coef0控制了高阶多项式与低阶多项式对模型的影响
])
poly_kernel_svm_clf.fit(m_x, m_y)
y_prd = poly_kernel_svm_clf.predict(m_x)
print("当前模型的准确率为: {}".format(clf_correct(m_y, y_prd)))
## 评测
print(confusion_matrix(m_y, y_prd))
""" result
当前模型的准确率为: 1.0
[[50 0]
[ 0 50]]
"""
3. 高斯核
if __name__ == '__main__' :
rbf_kernel_svm_clf = Pipeline([
("scaler", StandardScaler()),
('svm_clf', SVC(kernel='rbf', gamma=5, C=0.001))
])
rbf_kernel_svm_clf.fit(m_x, m_y)
y_prd = rbf_kernel_svm_clf.predict(m_x)
print("当前模型的准确率为: {}".format(clf_correct(m_y, y_prd)))
## 评测
print(confusion_matrix(m_y, y_prd))
""" result
当前模型的准确率为: 1.0
[[50 0]
[ 0 50]]
"""
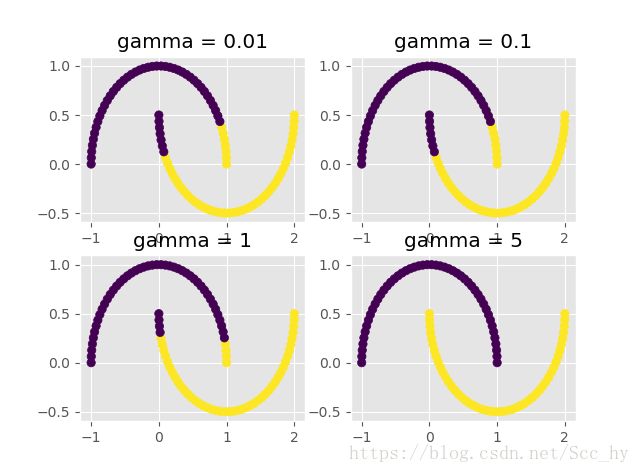
绘图查看gamma 值对分类结果的影响
def plot_gamma(gamma):
rbf_kernel_svm_clf = Pipeline([
("scaler", StandardScaler()),
('svm_clf', SVC(kernel='rbf', gamma=gamma, C=0.001))
])
rbf_kernel_svm_clf.fit(m_x, m_y)
y_prd = rbf_kernel_svm_clf.predict(m_x)
print("当前模型的准确率为: {}".format(clf_correct(m_y, y_prd)))
plot_moom(m_x, y_prd)
plt.title("gamma = {}".format(gamma))
if __name__ == '__main__' :
plt.subplot(2,2,1)
a = plot_gamma(0.01)
plt.subplot(2,2,2)
b = plot_gamma(0.1)
plt.subplot(2,2,3)
c = plot_gamma(1)
plt.subplot(2,2,4)
d = plot_gamma(5)
plt.show()
"""
当前模型的准确率为: 0.86
当前模型的准确率为: 0.86
当前模型的准确率为: 0.92
当前模型的准确率为: 1.0
"""
由上图可以看出 gamma 值影响分类结果:增加gamma 值会使得分类更加精确,即判断边界最终会变的不规则,围绕单个样本周围环绕。 反之,钟型曲线会更加的宽, 样本具有更宽的影响范围,判定边界更加的平滑。
所以gamma 是可调参数: 如果模型过拟合,需要减小 gamma 值; 若欠拟合,则增加 gamma 值
三、核的选择
1、计算复杂度
LinearSVC类基于liblinear库,它实现了线性SVM的优化算法。它并不支持核技巧,但是它样本和特征的数量几乎是线性的:训练时间复杂度大约为O(M*N)
当需要非常高的精度,那么算法就回更加耗时。这是由于 容差值超参数E (在Sklearn 称为 tol)控制的。大多分类任务中,使用默认容差值的效果是已经可以满足一般要求。
SVC类基于libsvm库,它实现了支持核技巧的算法。训练时间复杂度通常介于 O ( m 2 ∗ n ) O(m^2*n) O(m2∗n) 和 O ( m 3 ∗ n ) O(m^3*n) O(m3∗n) 之间。这个算法对于复杂但小型或者中等数量的数据集表现是完美的。
2. 核的一般选择
一般来说先用线性核 LinearSVC ,尤其是当训练集十分大的时候或者有大量的特征的情况下。如果训练集不大,可以尝试高斯径向核,它在大多数情况下都很有效。
其他的核函数较少使用,例如,一些核函数是专门用于特定对数据结构的。在对文本文档或者DNA序列进行分类时,有时候会使用字符串核(例如,使用 SSK核(string subsequence kernel) 或者
基于编辑距离的核函数)。
四、SVM回归
SVM算法应用广泛: 不仅仅支持线性核非线性的分类任务,还支持线性和非线性的回归任务。
技巧在于【逆转目标】:限制间隔违规的情况下,不是试图在两个类别之间找到尽可能大的间隔。
SVM回归任务是限制间隔违规情况下,尽可能防止更多的样本在街道上。
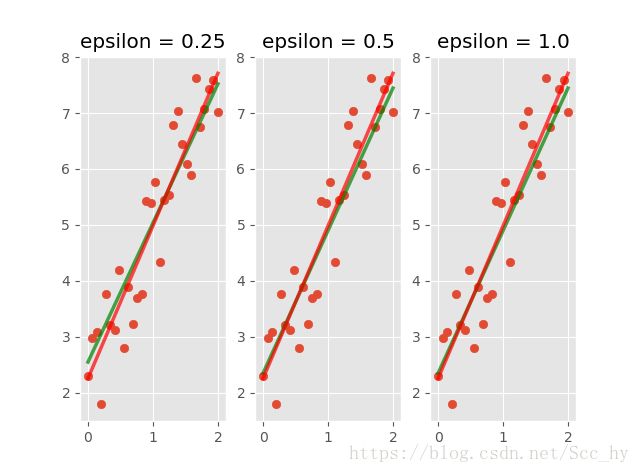
"街道"的宽度由超参数E (epsilon)控制,
添加更多的数据样本在间隔之内并不会影响模型的预测,因此,这个模型认为是不敏感的 (ϵ-insensitive)。
在sklearn 中用 LinearSVR 实现SVM回归
1. 线性回归
from sklearn.svm import LinearSVR
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import cross_val_predict
def get_line_data():
np.random.seed(1314)
x = np.linspace(0, 2, 30)
y = 1 + 3 * x + 2 * np.random.rand(len(x))
plt.scatter(x, y)
plt.show()
x_in_t = x.reshape((-1, 1))
return np.c_[np.ones((30, 1)), x_in_t], y.reshape((-1, 1))
def svm_reg_prd(x_in, y_in, epsilon):
svm_reg = LinearSVR(epsilon = epsilon) # 缩小街区的大小可以增加回归的准确性
svm_reg.fit(x_in, y_in)
return svm_reg.predict(x_in)
def plt_two_prd(x, y_prd, y_prd_l):
plt.scatter(np.array(x).reshape((-1, 1)), y_in)
plt.plot(x, y_prd, c='green', lw = 2.5, alpha=0.7)
plt.plot(x, y_prd_l, c='red', lw = 2.5, alpha=0.7)
if __name__ == '__main__' :
# SVR
x_in , y_in =get_line_data()
x = [x_in[i][1] for i in range(len(x_in))]
y_prd1 = svm_reg_prd(x_in, y_in, 0.25)
y_prd2 = svm_reg_prd(x_in, y_in, 0.5)
y_prd3 = svm_reg_prd(x_in, y_in, 1.0)
# linearregression
l_reg = LinearRegression()
l_reg.fit(x_in, y_in)
y_prd_l = l_reg.predict(x_in)
## 图示两个回归,及其他epsilon的表现
plt.subplot(131)
plt_two_prd(x, y_prd1, y_prd_l), plt.title("epsilon = 0.25")
plt.subplot(132)
plt_two_prd(x, y_prd2, y_prd_l), plt.title("epsilon = 0.5")
plt.subplot(133)
plt_two_prd(x, y_prd2, y_prd_l), plt.title("epsilon = 1.0")
plt.show()
## 评估
mse_svr1 = np.var(y_prd1.reshape((-1, 1)) - y_in)
mse_svr2 = np.var(y_prd2.reshape((-1, 1)) - y_in)
mse_svr3 = np.var(y_prd3.reshape((-1, 1)) - y_in)
mse_lreg = np.var(y_prd_l.reshape((-1, 1)) - y_in)
print("SVM回归(epsilon=0.25)MSE为:%.3f; SVM回归(epsilon=0.5)MSE为:%.3f;\nSVM回归(epsilon=1.0)MSE为:%.3f; 线性回归MSE为:%.3f"%(mse_svr1,mse_svr2,mse_svr3, mse_lreg))
"""
SVM回归(epsilon=0.25)MSE为:0.394; SVM回归(epsilon=0.5)MSE为:0.385;
SVM回归(epsilon=1.0)MSE为:0.376; 线性回归MSE为:0.374
"""
从均方误差和图,我们可以看出当 epsilon 增大的时候回归会更加精确,即 epsilon 控制着街区的大小,当epsilon 街区限制会变高,因而街区会变小。
(在
LinearSVR中)本质是 :不敏感损失函数中的Epsilon参数:添加更多的数据样本在间隔之内并不会影响模型的预测
2. 非线性回归(SVR)
简单实现
from sklearn.svm import SVR
from sklearn.linear_model import LinearRegression
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import PolynomialFeatures
def get_poly_data():
np.random.seed(1212)
x = np.linspace(-2, 2, 60)
y_2 = 3 * x ** 2 + 2 * np.random.rand(len(x)) + 0.3
x_in_t1 = x.reshape((-1, 1))
return np.c_[np.ones((len(x), 1)), x_in_t1], y_2.reshape((-1, 1))
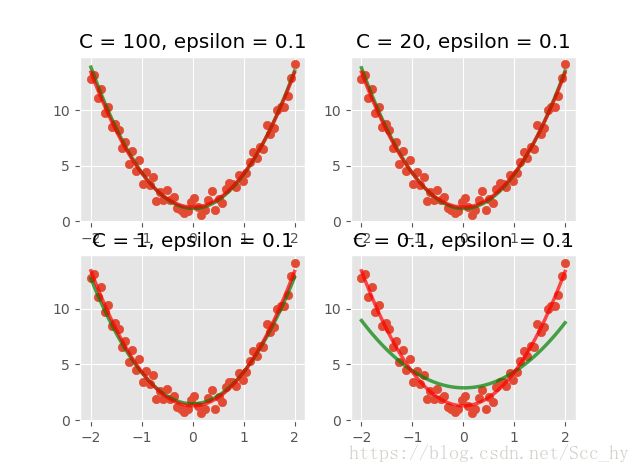
def svm_poly_prd(x_in, y_in, C = 100.0 , epsilon = 0.1):
svm_ploy_reg = SVR(kernel='poly', degree = 2, C = C, epsilon = epsilon)
svm_ploy_reg.fit(x_in, y_in)
return svm_ploy_reg.predict(x_in)
if __name__ == '__main__' :
## SVR
x_in, y_in = get_poly_data()
x = [x_in[i][1] for i in range(len(x_in))]
y_prd1, y_prd2= svm_poly_prd(x_in, y_in, C = 100), svm_poly_prd(x_in, y_in, C = 20)
y_prd3, y_prd4 = svm_poly_prd(x_in, y_in, C = 1), svm_poly_prd(x_in, y_in, C = 0.1)
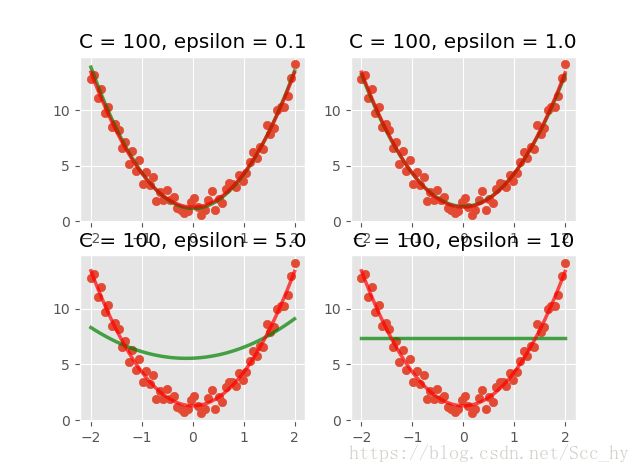
y_prd5, y_prd6= svm_poly_prd(x_in, y_in, epsilon = 0.1), svm_poly_prd(x_in, y_in, epsilon = 1.0)
y_prd7, y_prd8= svm_poly_prd(x_in, y_in, epsilon = 5.0), svm_poly_prd(x_in, y_in, epsilon = 10)
## l_reg
l_poly_reg = Pipeline([
('poly_x', PolynomialFeatures()),
('l_reg', LinearRegression())
])
l_poly_reg.fit(x_in, y_in)
y_prd = l_poly_reg.predict(x_in)
y_prd_plt = [i[0] for i in y_prd]
#绘图
plt.subplot(2,2,1)
plt_two_prd(x, y_prd1, y_prd), plt.title('C = 100, epsilon = 0.1')
plt.subplot(2,2,2)
plt_two_prd(x, y_prd2, y_prd), plt.title('C = 20, epsilon = 0.1')
plt.subplot(2,2,3)
plt_two_prd(x, y_prd3, y_prd), plt.title('C = 1, epsilon = 0.1')
plt.subplot(2,2,4)
plt_two_prd(x, y_prd4, y_prd), plt.title('C = 0.1, epsilon = 0.1')
plt.show()
plt.subplot(2, 2, 1)
plt_two_prd(x, y_prd5, y_prd), plt.title('C = 100, epsilon = 0.1')
plt.subplot(2, 2, 2)
plt_two_prd(x, y_prd6, y_prd), plt.title('C = 100, epsilon = 1.0')
plt.subplot(2, 2, 3)
plt_two_prd(x, y_prd7, y_prd), plt.title('C = 100, epsilon = 5.0')
plt.subplot(2, 2, 4)
plt_two_prd(x, y_prd8, y_prd), plt.title('C = 100, epsilon = 10')
plt.show()
当epsilon = 0.1 时, C 越小(脾气越小)数据容错率越高,模型拟合越随意。 所以在回归任务的时候C 要相对较大。
本质是正则化参数:该参数越大,使用的正则化越少。
当控制C = 100 即软间隔大小固定的时候, 随着 epsilon 增加,其模型拟合反而更差,这和SVM线性回归不同