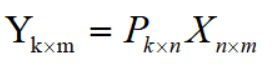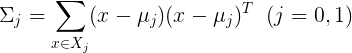ML-降维:PCA、SVD、LDA、MDS、LLE、LE算法总结
1.PCA主成分分析
PCA是不考虑样本类别输出的无监督降维技术,实现的是高维数据映射到低维的降维。
PCA原理这个介绍的不错:https://www.cnblogs.com/pinard/p/6239403.html
线性代数矩阵性质背景:特征值表示的是矩阵在特征值对应的特征向量方向上的伸缩大小;线性代数的本质这个课有不错介绍:https://www.bilibili.com/video/av6731067
步骤:
1)组成数据矩阵
def get_date():
m_vec = np.array([0, 0, 0])
cov_vec = np.array([[1, 0, 0], [0, 1, 0], [0, 0, 1]])
# 20个3维,且每个维度以m_vec为均值,以cov_vec为方差的多元正态分布随机数
c1 = np.random.multivariate_normal(m_vec, cov_vec, 20).T
m_vec2 = np.array([1, 1, 1])
cov_vec2 = np.array([[1, 0, 0], [0, 1, 0], [0, 0, 1]])
# 20个3维,且每个维度以m_vec2为均值,以cov_vec2为方差的多元正态分布随机数
c2 = np.random.multivariate_normal(m_vec2, cov_vec2, 20).T
# 合并构造一个3 x 40 的数据集
data = np.concatenate((c1, c2), axis=1)
return data
X = get_date()
2) 计算特征均值
mean_X = np.mean(X, axis=1).reshape(X.shape[0], 1)
3) 去中心化的新数据矩阵
dmean_X = X - mean_X4)计算协方差矩阵
m = dmean_X.shape[1]
C = np.dot(dmean_X, dmean_X.T) / m5)C的分解
计算C的特征值,特征向量,(大到小排列,特征值-向量一一对应)
eig_val_sc, eig_vec_sc = np.linalg.eig(C)6)取得投影矩阵
选择取C分解的前k(k 7)实现数据降维,n维到k维 然后用Yk.m替代Xn.m,作为降维数据集使用 可以看到降维到2的数据,还是有明显的区分的。 sklearn-PCA decomposition.PCA(n_components=None, copy=True, whiten=False) n_components: PCA算法中所要保留的主成分个数n,也即保留下来的特征个数n 可以看到,sklearn的效果和前面编程实现的一样。 梯度上升PCA 前面是基于矩阵分解,这个是用梯度下降: https://www.jianshu.com/p/1e9cab07d54d 原理参考:https://www.cnblogs.com/pinard/p/6251584.html 这里不加证明的写出如下总结: 对于任意矩阵Amxn,其奇异值是 若特征值<0,m 通常,按大小排列的奇异值,减少特别的快,在很多情况下,前10%甚至1%的奇异值的和就占了全部的奇异值之和的99%以上的比例。也就是说,我们也可以用最大的k个的奇异值和对应的左右奇异向量来近似描述矩阵。也就是说: sklearn实现: LDA是一种监督学习的降维技术,也就是说它的数据集的每个样本是有类别输出的。这点和PCA不同。核心思想是投影后类内方差最小,类间方差最大,如下右图(2维到1维),显然比左图更符合这个思想,LDA就是希望降维后的数据,能最大化的满足这个。 原理介绍:https://www.cnblogs.com/pinard/p/6244265.html 考虑二类别情况: 定义均值向量: 协方差: 希望同类更近,异类更远,意味着希望如下关系尽可能大: 定义类内散度矩阵: 类间散度矩阵: 目标变成了: 对上目标进行求解,其实是转为对某矩阵的分解,我的另一个文章的最后一张图有介绍:https://blog.csdn.net/jiang425776024/article/details/87607301 ,最后会得出结论,问题的求解等价于关系 多分类的情况也是一样的,最后的关键也是对 有数据集: 希望降维到d 1) 计算类内散度矩阵Sw:: 2)计算类间散度矩阵Sb,u为所有样本均值向量: 3) 计算矩阵 4)矩阵分解 5) 对样本集中的每一个样本特征xi转化为新的样本 6) 得到输出新样本集: numpy实现,加强对原理的理解: sklearn 实现3维到2维: 流形学习(Manifold Learning)的一类,多维缩放MDS要求原始空间中的样本之间的距离在低维空间中保持,思想是构造距离矩阵,然后矩阵分解,和前面的降维流程基本上是一致的,只是矩阵分解时的构造不一样,没有特别之处。 具体细节参考《机器学习》周志华 P228。或者博客:https://blog.csdn.net/AI_BigData_wh/article/details/78242052 MDS流程: sklearn API:https://scikit-learn.org/stable/modules/generated/sklearn.manifold.MDS.html Isomap:通过“改造一种原本适用于欧氏空间的算法”,达到了“将流形映射到一个欧氏空间”的目的。 流形Manifold:“嵌入在高维空间中的低维流形”,最直观的例子通常都会是嵌入在三维空间中的二维或者一维流形。比如说一块布,可以把它看成一个二维平面,这是一个二维的欧氏空间,现在我们(在三维)中把它扭一扭,它就变成了一个流形(当然,不扭的时候,它也是一个流形,欧氏空间是流形的一种特殊情况)。 MDS 是针对欧氏空间设计的,对于距离的计算也是使用欧氏距离来完成的。如果数据分布在流形上欧氏距离不适用。 Isomap通过把数据点连接起来构成一个邻接 Graph 来离散地近似原来的流形,而测地距离也相应地通过 Graph 上的最短路径来近似,通过这样的距离计算,把 MDS 中原始空间中距离的计算从欧氏距离换为了流形上的测地距离。 如下图:两点间的距离不再是欧氏距离!! 算法流程: 核心就是把MDS的距离计算改为了最短路径(Dijikstra、Floyd)等算法的计算。 sklearn API:https://scikit-learn.org/stable/modules/generated/sklearn.manifold.Isomap.html#sklearn.manifold.Isomap Isomap等距映射算法有一个问题就是他要找所有样本全局的最优解,当数据量很大,样本维度很高时,计算非常的耗时,鉴于这个问题,出现了只关注局部的LLE 局部线性嵌入(Locally Linear Embedding,以下简称LLE),也是流形学习算法,LLE关注于降维时保持样本局部的线性关系,由于LLE在降维时保持了样本的局部关系,它广泛的用于图像图像识别,高维数据可视化等领域。 参考:https://www.cnblogs.com/pinard/p/6266408.html LLE首先假设数据在较小的局部是线性的,也就是说,某一个数据可以由它邻域中的几个样本来线性表示,如原来的线性关系: 降维后的线性关系: 也就是说降维前后(局部)线性权重不变。 sklearn API:https://scikit-learn.org/stable/modules/generated/sklearn.manifold.locally_linear_embedding.html#sklearn.manifold.locally_linear_embedding 官方例子:https://scikit-learn.org/stable/auto_examples/manifold/plot_swissroll.html#sphx-glr-auto-examples-manifold-plot-swissroll-py 其思路和LLE很相似,也是基于图的降维算法,希望相互关联的点降维后的空间尽可能靠近,通过构建邻接矩阵,最后推导,矩阵分解等步骤,实现降维。 sklearn API:https://scikit-learn.org/stable/modules/generated/sklearn.manifold.SpectralEmbedding.html#sklearn.manifold.SpectralEmbedding k=2
# 从大到小特征值的k个索引
argsort_eigv = np.argsort(eig_val_sc)[-k:][::-1]
# 从大到小特征值的k个索引对应的特征向量,构造新矩阵
W = eig_vec_sc[argsort_eigv]# 构造的新矩阵对原数据X降维
new_X = np.dot(W, X)import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
def get_date():
m_vec = np.array([0, 0, 0])
cov_vec = np.array([[1, 0, 0], [0, 1, 0], [0, 0, 1]])
# 20个3维,且每个维度以m_vec为均值,以cov_vec为方差的多元正态分布随机数
c1 = np.random.multivariate_normal(m_vec, cov_vec, 20).T
m_vec2 = np.array([1, 1, 1])
cov_vec2 = np.array([[1, 0, 0], [0, 1, 0], [0, 0, 1]])
# 20个3维,且每个维度以m_vec2为均值,以cov_vec2为方差的多元正态分布随机数
c2 = np.random.multivariate_normal(m_vec2, cov_vec2, 20).T
# 合并构造一个3 x 40 的数据集
data = np.concatenate((c1, c2), axis=1)
return data
X = get_date()
k = 2
# ====基于线性代数实现PCA降维======
mean_X = np.mean(X, axis=1).reshape(X.shape[0], 1)
dmean_X = X - mean_X
m = dmean_X.shape[1]
C = np.dot(dmean_X, dmean_X.T) / m
# 矩阵分解 奇异值,特征向量
eig_val_sc, eig_vec_sc = np.linalg.eig(C)
# 从大到小特征值的k个索引
argsort_eigv = np.argsort(eig_val_sc)[-k:][::-1]
# 从大到小特征值的k个索引对应的特征向量,构造新矩阵
W = eig_vec_sc[argsort_eigv]
# 构造的新矩阵对原数据X降维
new_X = np.dot(W, X)
# 显示降维后的数据
plt.scatter(new_X.T[:, 0], new_X.T[:, 1], c='y')
plt.show()
from sklearn.decomposition import PCA
pca = PCA(n_components=3)
pca.fit(X)
print pca.explained_variance_ratio_
print pca.explained_variance_
X_new = pca.transform(X)#实现对X的降维
copy:表示是否在运行算法时,将原始训练数据复制一份。
若为True,则运行PCA算法后,原始训练数据的值不会有任何改变,因为是在原始数据的副本上进行运算;若为False,则运行PCA算法后,原始训练数据的值会改,因为是在原始数据上进行降维计算。
whiten: bool,缺省时默认为False,True使得每个特征具有相同的方差。
explained_variance_ratio_:计算了每个特征方差贡献率,所有总和为1
explained_variance_:方差值
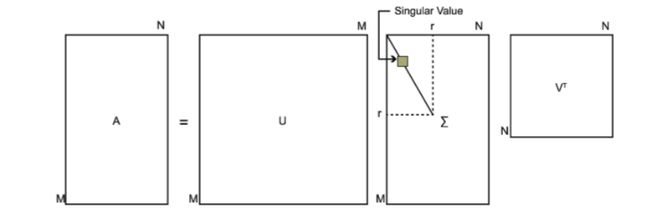
2.SVD
![]() 的特征值的平方根;
的特征值的平方根;![]() 的特征向量是:v1,v2,v3,则A的特征向量是:
的特征向量是:v1,v2,v3,则A的特征向量是:2.1SVD降维:
![]() ,可以实现降维。(线性代数中,特征向量意味着方向,对应的特征值意味着矩阵在特征向量上的拉伸程度,取大的k个特征值对应的特征向量,可以近似的描述这个拉伸过程,极大程度上保存原有数据形状)
,可以实现降维。(线性代数中,特征向量意味着方向,对应的特征值意味着矩阵在特征向量上的拉伸程度,取大的k个特征值对应的特征向量,可以近似的描述这个拉伸过程,极大程度上保存原有数据形状)import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.datasets import load_iris
# ======创建数据集========
iris = load_iris()
df = pd.DataFrame(iris.data, columns=iris.feature_names)
df['label'] = iris.target
df.columns = ['sepal length', 'sepal width', 'petal length', 'petal width', 'label']
labels = df.iloc[:, -1].values
data = df.iloc[:, :-1].values
samples, features = df.shape
print(samples, features)
# =======这里SVD========
U, s, V = np.linalg.svd(data)
# =====取前3维数据显示===========
newdata = U[:, :3]
fig = plt.figure()
ax = Axes3D(fig)
# =======每个类型的形状和颜色=====
marks = ['o', '*', '+']
colors = ['r', 'b', 'g']
for i in range(samples):
ax.scatter(newdata[i, 0], newdata[i, 1], newdata[i, 2], c=colors[int(labels[i])], marker=marks[int(labels[i])])
plt.show()
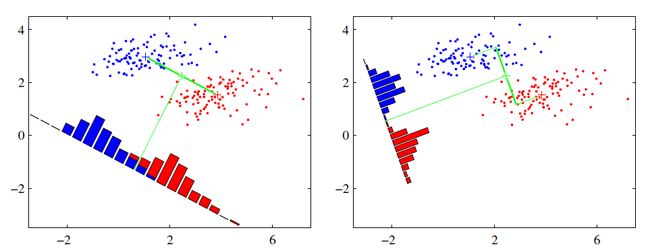
3 LDA
![]()
![]() ,只要对其矩阵分解得到
,只要对其矩阵分解得到![]() ,就找到了w的解(最大维度<2)。
,就找到了w的解(最大维度<2)。![]() 矩阵分解求w。
矩阵分解求w。3.1LDA算法流程
![]() ,
,![]()
![]()
![]() ,得到最大的d个特征值及对应的d个特征向量
,得到最大的d个特征值及对应的d个特征向量![]() ,得到投影矩阵W
,得到投影矩阵W![]()
![]()
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
iris = load_iris()
df = pd.DataFrame(iris.data, columns=iris.feature_names)
df['label'] = iris.target
df.columns = ['sepal length', 'sepal width', 'petal length', 'petal width', 'label']
data = np.array(df.iloc[:100, [0, 1, 2, -1]])
X = data[:, :-1]
Y = data[:, -1]
# 类别数
class_labels = np.unique(Y)
n_class = class_labels.shape[0]
# 类均值向量
mean_vec = []
for c in range(n_class):
mean_vec.append(np.mean(X[Y == class_labels[c]], axis=0))
# 类内散度矩阵Sw
SW = np.zeros((3, 3))
for cl, mv in zip(class_labels, mean_vec):
sc_matrix_class = np.zeros((3, 3))
for row in X[Y == cl]: # 所有X中类别=cl的样本
row, mv = row.reshape(3, 1), mv.reshape(3, 1)
sc_matrix_class += (row - mv).dot((row - mv).T)
SW += sc_matrix_class
# Sb
all_mean_vec = np.mean(X, axis=0)
n_features = X.shape[1]
SB = np.zeros((n_features, n_features))
for i, mea_v in enumerate(mean_vec):
n = X[Y == class_labels[i]].shape[0]
mea_v = mea_v.reshape(n_features, 1)
all_mean_vec = all_mean_vec.reshape(n_features, 1)
SB += n * (mea_v - all_mean_vec).dot((mea_v - all_mean_vec).T)
# Sw^-1 Sb矩阵分解
e_val, e_vec = np.linalg.eig(np.linalg.inv(SW).dot(SB))
dindex = np.argsort(e_val)[-2:][::-1]
W = e_vec[dindex]
new_X = np.dot(W, X.T)
plt.scatter(new_X.T[:, 0], new_X.T[:, 1])
plt.show()
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.datasets.samples_generator import make_classification
X, y = make_classification(n_samples=1000, n_features=3, n_redundant=0, n_classes=3, n_informative=2,
n_clusters_per_class=1, class_sep=0.5, random_state=10)
# fig = plt.figure()
# ax = Axes3D(fig, rect=[0, 0, 1, 1], elev=30, azim=20)
# ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker='o', c=y)
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
lda = LinearDiscriminantAnalysis(n_components=2)
lda.fit(X, y)
X_new = lda.transform(X)
plt.scatter(X_new[:, 0], X_new[:, 1], marker='o', c=y)
plt.show()
4 MDS 流形学习(Manifold Learning)
from sklearn.datasets import load_digits
from sklearn.manifold import MDS
X, _ = load_digits(return_X_y=True)
X.shape
embedding = MDS(n_components=2)
X_transformed = embedding.fit_transform(X[:100])
X_transformed.shape
4.1 Isomap 等度量映射
from sklearn.datasets import load_digits
from sklearn.manifold import Isomap
X, _ = load_digits(return_X_y=True)
X.shape
embedding = Isomap(n_components=2)
X_transformed = embedding.fit_transform(X[:100])
X_transformed.shape
5 LLE 局部线性嵌入
![]()
![]()
6 LE 拉普拉斯特征映射
from sklearn import manifold, datasets
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
fig = plt.figure()
ax = Axes3D(fig)
X, color = datasets.samples_generator.make_swiss_roll(n_samples=1500)
se = manifold.SpectralEmbedding(n_components=2, n_neighbors=10)
Y = se.fit_transform(X)
# 原3维
# ax.scatter(X[:, 0], X[:, 1], X[:, 2], c=color)
# 降维 2d
plt.scatter(Y[:, 0], Y[:, 1], c=color)
plt.show()