SKlearn库EM算法混合高斯模型参数说明及代码实现
SKlearn库EM算法混合高斯模型参数说明及代码实现
SKlearn库GaussianMixture类是EM算法在混合高斯分布的实现,现简单记录下其参数说明。
一、SKlearn混合高斯模型参数说明
GaussianMixture(n_components=1, covariance_type=’full’, tol=0.001, reg_covar=1e-06, max_iter=100, n_init=1, init_params=’kmeans’, weights_init=None, means_init=None,
precisions_init=None, random_state=None, warm_start=False, verbose=0, verbose_interval=10)
参数:
1. n_components:混合高斯模型个数,默认为1
2. covariance_type:协方差类型,包括{‘full’,‘tied’, ‘diag’, ‘spherical’}四种,分别对应完全协方差矩阵(元素都不为零),相同的完全协方差矩阵(HMM会用到),对角协方差矩阵(非对角为零,对角不为零),球面协方差矩阵(非对角为零,对角完全相同,球面特性),默认‘full’ 完全协方差矩阵
3. tol:EM迭代停止阈值,默认为1e-3.
4. reg_covar:协方差对角非负正则化,保证协方差矩阵均为正,默认为0
5. max_iter:最大迭代次数,默认100
6. n_init:初始化次数,用于产生最佳初始参数,默认为1
7. init_params: {‘kmeans’, ‘random’}, defaults to ‘kmeans’.初始化参数实现方式,默认用kmeans实现,也可以选择随机产生
8. weights_init:各组成模型的先验权重,可以自己设,默认按照7产生
9. means_init:初始化均值,同8
10. precisions_init:初始化精确度(模型个数,特征个数),默认按照7实现
11. random_state :随机数发生器
12. warm_start :若为True,则fit()调用会以上一次fit()的结果作为初始化参数,适合相同问题多次fit的情况,能加速收敛,默认为False。
13. verbose :使能迭代信息显示,默认为0,可以为1或者大于1(显示的信息不同)
14. verbose_interval :与13挂钩,若使能迭代信息显示,设置多少次迭代后显示信息,默认10次。
二、代码实现
首先,回顾EM算法基本流程:
1.E step
![]()
具体而言,是指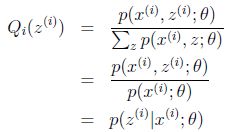
其中分子实际上可以写作:
![]()
=![]()
2.M step
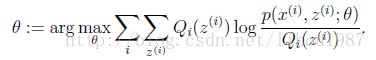
即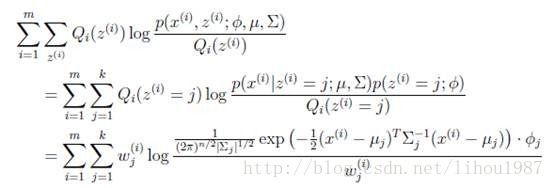
剩下的就是对参数求导的问题了。
接下来看源代码怎么实现的?
- 对应E step的代码
(1)首先将模型完全转换成对数计算,根据高斯密度函数公式分别计算k个组成高斯模型的log值,即logP(x|z)的值
def _estimate_log_gaussian_prob(X, means, precisions_chol, covariance_type):
# 计算精度矩阵的1/2次方log_det(代码精度矩阵是通过cholesky获取)
log_det = _compute_log_det_cholesky(
precisions_chol, covariance_type, n_features)
# 对应上面四种协方差类型,分别计算精度矩阵与(x-u)相乘那部分log_prob
if covariance_type == 'full':
log_prob = np.empty((n_samples, n_components))
for k, (mu, prec_chol) in enumerate(zip(means, precisions_chol)):
y = np.dot(X, prec_chol) - np.dot(mu, prec_chol)
log_prob[:, k] = np.sum(np.square(y), axis=1)
elif covariance_type == 'tied':
log_prob = np.empty((n_samples, n_components))
for k, mu in enumerate(means):
y = np.dot(X, precisions_chol) - np.dot(mu, precisions_chol)
log_prob[:, k] = np.sum(np.square(y), axis=1)
elif covariance_type == 'diag':
precisions = precisions_chol ** 2
log_prob = (np.sum((means ** 2 * precisions), 1) -
2. * np.dot(X, (means * precisions).T) +
np.dot(X ** 2, precisions.T))
elif covariance_type == 'spherical':
precisions = precisions_chol ** 2
log_prob = (np.sum(means ** 2, 1) * precisions -
2 * np.dot(X, means.T * precisions) +
np.outer(row_norms(X, squared=True), precisions))
# 最后计算出logP(x|z)的值
return -.5 * (n_features * np.log(2 * np.pi) + log_prob) + log_det
(2)P(x|z)*P(z)计算每个模型的概率分布P(x,z),求对数则就是相加了
def _estimate_weighted_log_prob(self, X):
return self._estimate_log_prob(X) + self._estimate_log_weights()
(3)最后开始计算每个模型的后验概率logP(z|x),即Q函数
def _estimate_log_prob_resp(self, X):
weighted_log_prob = self._estimate_weighted_log_prob(X)
#计算P(X)
log_prob_norm = logsumexp(weighted_log_prob, axis=1)
with np.errstate(under='ignore'):
# 忽略下溢,计算每个高斯模型的后验概率,即占比,对数则相减
log_resp = weighted_log_prob - log_prob_norm[:, np.newaxis]
return log_prob_norm, log_resp
(4)调用以上函数
#返回所有样本的概率均值,及每个高斯分布的Q值
def _e_step(self, X):
log_prob_norm, log_resp = self._estimate_log_prob_resp(X)
return np.mean(log_prob_norm), log_resp
2.对应M step
def _m_step(self, X, log_resp):
#根据上面获得的每个高斯模型的Q值(log_resp)。重新估算均值self.means_,协方差self.covariances_,当前符合各高斯模型的样本数目self.weights_(函数名起的像权重,实际指的是数目)
n_samples, _ = X.shape
self.weights_, self.means_, self.covariances_ = (
_estimate_gaussian_parameters(X, np.exp(log_resp), self.reg_covar,
self.covariance_type))
#更新当前各高斯模型的先验概率,即P(Z)
self.weights_ /= n_samples
#根据cholesky分解计算精度矩阵
self.precisions_cholesky_ = _compute_precision_cholesky(
self.covariances_, self.covariance_type)
然后重复以上过程,就完成了EM算法的实现啦。