- 读《人世间》有感
一0一
这个寒假,就如同朋友圈中的一段话:一闭眼,一睁眼假期还有5天,在一闭眼一睁眼假期还有12天;再一闭眼一睁眼假期还有20天;不敢睡,不敢睡啊……受疫情影响,这个假期变得漫长又煎熬,我也无时无刻不关注着疫情的变化。当然这样的一个假期,我还真得要感谢周翔,因为他有个爱看书的习惯,所以家里有不少他看过的书,可以让我随意挑选,因此也让我的假期不至于那么无所事事。这次我选了一本梁晓声的《人世间》,作为一名语文
- 《人世间》
南询yi
今日分享十点推文,《人世间》有感苏格拉底说:“天地只有三尺,而人在五尺开外,所以人人都要懂得低头。”深以为然。懂得低头,不是认输。而是于人世间找寻温存的成熟,于困境中寻觅柳暗花明的智慧,于争执中展示屈伸自如的格局。正如仰头不是骄傲,是要看见自己的天空;低头也不是认输,而是要看清自己的路。成大事者,不仅要抬头挺胸,还得低头看路。懂得低头,进退有度,不是认输,而是竭尽全力过好这一生。宫崎骏说过:“所有
- 读《人间鲁迅》有感
琳语读书
上周读完《闻一多传》后,我对中国近代知识分子产生了兴趣,这周继续读了《人间鲁迅》。厚厚的两本书,记录了一个人的一生,苦痛,彷徨和挣扎,虽然只读了一小部分,却也心潮澎湃。闻一多和鲁迅是完全不同的。鲁迅是沉郁的,现实的,寂寞的,抗争的。除了天生性格的不同外,环境的塑造也是非常之大。鲁迅少年经历了家庭的变故,看尽了人间冷暖,世态炎凉。这种经历促使他很早就观察思考人生,立志用文学来改变中国国民的劣根。闻一
- 女儿考研完报考雅思
捡拾流年
是否我过于焦虑?会不会无形间让女儿觉得压力太大了啊。2022年对于我们家来说是不平常的一年。女儿今年大四,为了准备考研,暑假也没回家,年初去了学校到了年末才回家。女儿自己一个人面对考研,没有参加培训,大四学校作业论文等课业也多,她同时也是很努力复习考研的。在疫情开放很多羊的时期,女儿终于顺顺利利参加12月24、25号的考研,我们和家人都觉得女儿回家来要好好休息调养。可女儿回到家,我再查阅考研信息,
- 【六项精进】20180930
Kinnfoo
一、学习与实践1.付出不亚于任何人的努力2.要谦虚,不要骄傲3.要每天反省4.活着,就要感谢5.积善行,思利他6.不要有感性的烦恼二、今日分享今天是9月的最后一个工作日,每个支行都在拼命地冲刺业绩,刚好今天同桌休假了,我就替他审核客户。一个上午就进件了6个客户,审核通过5个。这5个审核通过的客户里,1个因费率没谈拢而放弃,1个因车上发现GPS而被拒单,最终确认可放款的只有3个客户。感叹支行同事的不
- 如何培养兴趣
绽蕊向阳
今天读李笑来的书《与时间做朋友》,读到有关兴趣部分,深有感触。书中提到,好多人说对某事没有兴趣,实际上是没有能力把这件事做好,做这件事时的感受很不好,有挫败感,每个人对自己不擅长做不好的事情,都本能的容易逃避,所以就以为自己对这件事不感兴趣,他们真正感兴趣的是其他事情。可事实上,出现这种感觉应该仅仅是因为还没有开始做那件事情,也还没有在那件事情上遭受挫折而已。其实,很多人真的放弃原来做的事情,转去
- 今日有感,坚持分享第913天,2019.07.13
ZAF峰回路转
本周是假日里最忙碌的一周,连续四天晚上的课程,让我感觉到身体明显透支。昨天晚上读书会结束回到家,已经是十点半之后啦,忽然感觉身体不舒服,勉强支撑着洗漱完毕,没等上床休息,强烈的不适感警告我该吃药啦!感谢老公半夜到医院給我抓了药,今天早上当我对老公表达谢意的时候,老公说,不用感谢,我不是一直都是这样做的吗?多少年啦,今天竟然还谢谢!老公说的没错,可是以前总感觉那是他应该做的,如今感觉到,身边有一个在
- 绝招曝光!3小时高效利用ChatGPT写出精彩论文
kkai人工智能
chatgpt人工智能ai学习媒体
在这份指南中,我将深入解析如何利用ChatGPT4.0的高级功能,指导整个学术研究和写作过程。从初步探索研究主题,到撰写结构严谨的学术论文,我将一步步展示如何在每个环节中有效运用ChatGPT。如果您还未使用PLUS版本,可以参考相关教程。**初步探索与主题的确定**起初,我处于庞大的知识领域中,寻找一个可深入研究的领域。ChatGPT如同灯塔,通过深入分析最新研究趋势和领域热点,帮助我在广阔的学
- 自动写论文的网站推荐这5款实用类工具
小猪包333
写论文人工智能深度学习计算机视觉AI写作
在当今学术研究和写作领域,AI论文写作工具的出现极大地提高了写作效率和质量。这些工具不仅能够帮助研究人员快速生成论文草稿,还能进行内容优化、查重和排版等操作。以下是五款实用类工具推荐,特别是千笔-AIPassPaper。1.千笔-AIPassPaper千笔-AIPassPaper是一款功能强大且全面的AI论文写作助手,用户只需输入基本的研究需求和关键词,便能迅速生成一篇完整的论文。该工具利用先进的
- 推荐3家毕业AI论文可五分钟一键生成!文末附免费教程!
小猪包333
写论文人工智能AI写作深度学习计算机视觉
在当前的学术研究和写作领域,AI论文生成器已经成为许多研究人员和学生的重要工具。这些工具不仅能够帮助用户快速生成高质量的论文内容,还能进行内容优化、查重和排版等操作。以下是三款值得推荐的AI论文生成器:千笔-AIPassPaper、懒人论文以及AIPaperPass。千笔-AIPassPaper千笔-AIPassPaper是一款基于深度学习和自然语言处理技术的AI写作助手,旨在帮助用户快速生成高质
- 4款毕业论文参考文献格式生成器(附加详细步骤)
小猪包333
写论文人工智能深度学习计算机视觉AI写作
在撰写毕业论文时,参考文献的格式规范是至关重要的。为了帮助学生和学者们更高效地生成符合要求的参考文献格式,本文将详细介绍四款推荐的参考文献格式生成器,并提供详细的使用步骤。1.千笔-AIPassPaper千笔-AIPassPaper是一款先进的AI辅助论文写作工具,不仅能够自动生成大纲、开题报告,还能一键生成参考文献。AI论文,免费大纲,10分钟3万字https://www.aipaperpass
- AI论文写作推荐哪个好?分享5款AI论文写作带数据图表网站
小猪包333
写论文人工智能深度学习计算机视觉
在当今学术研究和写作领域,AI论文写作工具的出现极大地提高了写作效率和质量。这些工具不仅能够帮助研究人员快速生成论文草稿,还能进行内容优化、查重和排版等操作。以下是五款推荐的AI论文写作工具,包括千笔-AIPassPaper。千笔-AIPassPaper千笔-AIPassPaper是一款功能强大的AI论文写作助手,旨在帮助用户快速生成高质量的论文内容。AI论文,免费大纲,10分钟3万字https:
- AI论文题目生成器怎么用?9款论文写作网站简单3步搞定
小猪包333
写论文人工智能深度学习计算机视觉
在当今信息爆炸的时代,AI写作工具的出现极大地提高了写作效率和质量。本文将详细介绍9款优秀的论文写作网站,并重点推荐千笔-AIPassPaper。一、千笔-AIPassPaper千笔-AIPassPaper是一款功能强大的AI论文生成器,基于最新的自然语言处理技术,能够一键生成高质量的毕业论文、开题报告等文本内容。它不仅提供智能选题、文献推荐和论文润色等功能,还具有较高的用户评价。其文献综述生成功
- 毕业论文附录一般都写什么?大学生写论文是干嘛用的
写个原创论文
人工智能深度学习AI写作chatgpt论文阅读
毕业论文的附录通常包含一些在正文中不便于展示或详细阐述的内容,但对理解论文整体又具有重要意义的资料。具体来说,附录可能包含以下内容:AI论文,免费大纲,10分钟3万字,查重高于15%退费,支持数据图表!!AIPaperPass-AI论文写作指导平台AIPaperPass是AI原创论文写作平台,免费千字大纲,5分钟生成3万字初稿,提供答辩汇报ppt、开题报告、任务书等,40篇真实中英文知网参考文献,
- 【加密算法基础——RSA 加密】
XWWW668899
网络服务器笔记python
RSA加密RSA(Rivest-Shamir-Adleman)加密是非对称加密,一种广泛使用的公钥加密算法,主要用于安全数据传输。公钥用于加密,私钥用于解密。RSA加密算法的名称来源于其三位发明者的姓氏:R:RonRivestS:AdiShamirA:LeonardAdleman这三位计算机科学家在1977年共同提出了这一算法,并发表了相关论文。他们的工作为公钥加密的基础奠定了重要基础,使得安全通
- 2022-04-10
凤凰语言艺术吴老师
读刘院日更《再读稻盛和夫:习惯于用自己的承诺,倒逼自己成功》有感过去讲做人做事要“不言实行”,换言之,比起豪言壮语,默不作声、埋头实干才是美德。现如今社会,闷头干有时候也会失去动力。因为闷头干没有外界的监督,制定的计划只有自己知道,即使没有百分百完成,别人也不知道,久之就养成了得过且过的心态。就像当初自己花了不少钱报名学习日语一样,当时只是闷头学,没有开公失去了众人的监督,以致于后来因为工作和日常
- 读《幸福的勇气》有感
晓丽姐
1.三棱柱三棱柱,其中面朝来访者的两面是,可恶的他人与可怜的自己。来访者只能看到这两面,但第三面,猜猜是什么?其实是,应该怎么办?我们往往会沉浸在这样的故事中,他人是多么的可恶,我们是多么的可怜,而最重要的问题是,我应该怎么办?不妨花更多的心思在,我应该怎么办上面。2.课题分离课题分离,你做好自己的工作是自己的课题,而领导喜欢不喜欢你,要辞退你也是他的课题,我们要学会很好的课题分离。学习是孩子的事
- 【新教育-教师随笔】读《做最好的英语老师》有感
164c5aca7b79
伊川县直中学王素平《做最好的英语老师》这本书是作者这些年在他教学中得与失的总结。里面给我们提供了听力,单词,句子,阅读,作文等模块的教学方法,让我受益匪浅,现总结如下:一.语文教学给了我们什么启示?(1):现有的英语教材内容简单,枯燥,与学生的心智发展水平严重脱节。我们要给学生补中一些贴近学生生活,能感动和影响他们的经典作品。让学生学习知识的同时,有所感悟和思考,同时享受审美的乐趣!如AWiseO
- 《拖延心理学》(一)你为什么会拖延?|木盒笔记
纯se蓝调
《拖延心理学》是帮助你向拖延症宣战的一本书,作者简·博克和莱诺拉·袁是全球知名的拖延症治疗专家。大概每个人或多或少总会有一点拖延症的行为。比如明天要叫论文了,今天你还没有写好,你一边在焦虑症怎么办,一边又拿着手机漫无目的的刷新闻;比如你想了很久准备减肥,但是迟迟又没有行动,想着今天晚上少吃一点吧、明天我就开始运动。今天分析的笔记来告诉你“你为什么会拖延?”,解读人杨坚。有人说拖延就像巨大的泥沼,让
- 记上下班经甲秀楼有感
寒更香
文/洁净心念甲秀爽天气,旅人犹如织。凉风伴夏夜,归家悠哉行。图片发自App图片发自App图片发自App图片发自App图片发自App图片发自App
- 小学科学课堂管理规培有感
31c6a3d23d4e
今天进行了这学期的第一次新教师规培,又是干货满满的一次学习,两位有经验的老教师分享了自己很多年来有关科学教学的经验。苏老师的语言风趣幽默,通过他的课堂管理规则,懂得了语言的艺术。艳姐分享的培养小助手,对我的帮助很多,小组长,(做记录、整理材料、管理卫生)。既培养了学生,又减轻老师的工作量,还有评价表,学起来学起来!
- 2024年华为杯数学建模研赛C题思路代码+论文助攻
DS数模
2024华为杯数学建模华为2024华为杯2024研究生数学建模2024研赛
2024年华为杯研究生数学建模竞赛(以下简研赛)将于9月21日上午8时正式开始。下文包含:2024研赛思路解析、研赛参赛时间及规则信息说明、好用的数模技巧及如何备战数学建模竞赛C君将会第一时间发布选题建议、所有题目的思路解析、相关代码、参考文献、参考论文等多项资料,帮助大家取得好成绩。2024年研赛将于9月21日上午8时正式开始这里有些资料,大家可以看看:【2024最全国赛研赛数模资料包】C君珍贵
- 夏日随笔日记夏天的夜
住在城里的庄户孩子
浅聊微信朋友圈及其它文/王立虎(一)又是一个深夜了,夏天的夜显得有些浮躁有些闷热,透过窗户外面街道上街灯依旧明亮,照着匆忙的车与人回家。关上电脑,打开,还是先完成日更,一直坚持着努力着写着,虽没有什么优秀的大作出现,但有时候还是佩服自己对文学的执着和爱好,佩服自己的自律。写点吧,在这夜深人静的时候,独处着,习惯着,随笔写下自己一天的心情,有感悟,有事件,有温度,我想写下总是好的。也有人喜欢这个点来
- 轻量级模型解读——轻量transformer系列
lishanlu136
#图像分类轻量级模型transformer图像分类
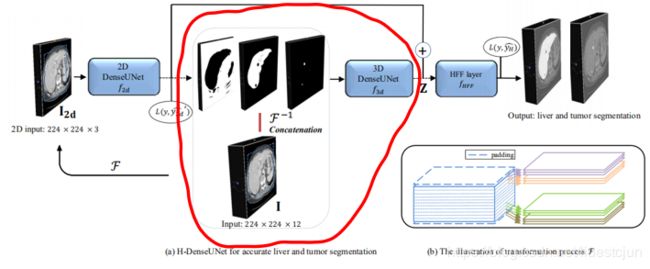
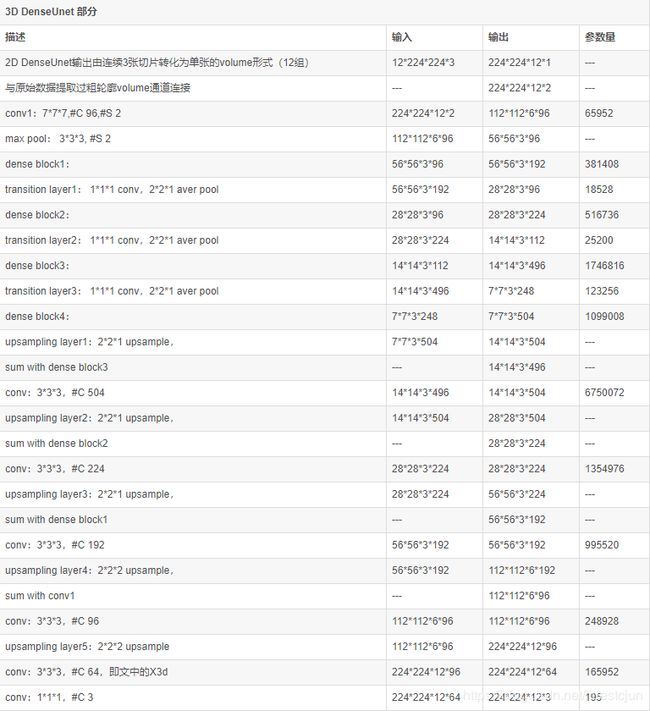
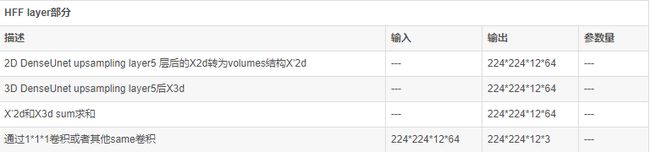
先占坑,持续更新。。。文章目录1、DeiT2、ConViT3、Mobile-Former4、MobileViTTransformer是2017谷歌提出的一篇论文,最早应用于NLP领域的机器翻译工作,Transformer解读,但随着2020年DETR和ViT的出现(DETR解读,ViT解读),其在视觉领域的应用也如雨后春笋般渐渐出现,其特有的全局注意力机制给图像识别领域带来了重要参考。但是tran
- 共读《绿野仙踪》有感 ——战胜自己就是勇敢的人
倚窗听雨_1ac2
今天我和孩子们一起共读的是《绿野仙踪》——路遇胆小狮。我发现这本书每一章的开头儿,都是对环境的描写。有干燥的堪萨斯州,有美丽的芒奇金人的国度,有通往翡翠城沿途优美的风景,有黑暗的森林......而这一章,重点描述了密林深处可怕的咆哮声。他们一行四人走在堆满黄树叶和枯树枝的黄砖路上。就在这样的情况下,他们遇到了一头大狮子。大狮子挥到了稻草人,扑倒了铁皮人。这危险时刻,托托虽小,却也敢面对强敌。它跑上
- 基于JavaWeb开发的Java+SpringMvc+vue+element实现上海汽车博物馆平台
网顺技术团队
成品程序项目javavue.js汽车课程设计springboot
基于JavaWeb开发的Java+SpringMvc+vue+element实现上海汽车博物馆平台作者主页网顺技术团队欢迎点赞收藏⭐留言文末获取源码联系方式查看下方微信号获取联系方式承接各种定制系统精彩系列推荐精彩专栏推荐订阅不然下次找不到哟Java毕设项目精品实战案例《1000套》感兴趣的可以先收藏起来,还有大家在毕设选题,项目以及论文编写等相关问题都可以给我留言咨询,希望帮助更多的人文章目录基
- 2019-04-22
平凡的人生Dian
六项精进打卡Day252一学习与实践1.付出不亚于任何人的努力2.要谦虚不要骄傲3.要每天反省4.活着,就要感谢5.积善行,思利他6.不要有感性的烦恼分享人总是对陌生人很宽容,对熟悉的人很挑剔。
- 深度学习-点击率预估-研究论文2024-09-14速读
sp_fyf_2024
深度学习人工智能
深度学习-点击率预估-研究论文2024-09-14速读1.DeepTargetSessionInterestNetworkforClick-ThroughRatePredictionHZhong,JMa,XDuan,SGu,JYao-2024InternationalJointConferenceonNeuralNetworks,2024深度目标会话兴趣网络用于点击率预测摘要:这篇文章提出了一种新
- 特别的520——家引师第二次团体会议
Erin曹
图片发自App前几天收到郭老师通知今天会议内容时,有点惊喜有点感动,还有感恩的,发现我要感恩的人好多哦!其实这个时间点我的身体感受是有点累的,有点困了想休息了,今天团体结束之后又到女儿的班级跟孩子们做了一次互动,到晚上九点钟才结束回家,之后班主任老师和学校德育主任分别与我沟通今天的互动情况,非常感恩老师和孩子们的敞开,让我更多一些了解孩子们,也让我确定接下来需要工作的方向!这是今晚的一个收获!图片
- 一路有你的美好Day525
果果儿guoguo132328
我的赞美日记:1.赞美自己完成早读后有多余时间就独自读了会儿书,早晨的时光可真宝贵呀!2.赞美自己开完一个会马不停蹄开下一个,这一天就开了四个会,我的腰还蛮坚强的。3.赞美自己对我的办公室特别有感情,上班时间一天不去就会想念。4.赞美自己难得和先生有一起下班的时光,于是去永辉超市各种买买买,久哥一直督促我们买买买,豪气。5.赞美自己和家人一起看了看电视。我的感恩日记:1.感恩公公悄悄就把久哥送去上
- 对股票分析时要注意哪些主要因素?
会飞的奇葩猪
股票 分析 云掌股吧
众所周知,对散户投资者来说,股票技术分析是应战股市的核心武器,想学好股票的技术分析一定要知道哪些是重点学习的,其实非常简单,我们只要记住三个要素:成交量、价格趋势、振荡指标。
一、成交量
大盘的成交量状态。成交量大说明市场的获利机会较多,成交量小说明市场的获利机会较少。当沪市的成交量超过150亿时是强市市场状态,运用技术找综合买点较准;
- 【Scala十八】视图界定与上下文界定
bit1129
scala
Context Bound,上下文界定,是Scala为隐式参数引入的一种语法糖,使得隐式转换的编码更加简洁。
隐式参数
首先引入一个泛型函数max,用于取a和b的最大值
def max[T](a: T, b: T) = {
if (a > b) a else b
}
因为T是未知类型,只有运行时才会代入真正的类型,因此调用a >
- C语言的分支——Object-C程序设计阅读有感
darkblue086
applec框架cocoa
自从1972年贝尔实验室Dennis Ritchie开发了C语言,C语言已经有了很多版本和实现,从Borland到microsoft还是GNU、Apple都提供了不同时代的多种选择,我们知道C语言是基于Thompson开发的B语言的,Object-C是以SmallTalk-80为基础的。和C++不同的是,Object C并不是C的超集,因为有很多特性与C是不同的。
Object-C程序设计这本书
- 去除浏览器对表单值的记忆
周凡杨
html记忆autocompleteform浏览
&n
- java的树形通讯录
g21121
java
最近用到企业通讯录,虽然以前也开发过,但是用的是jsf,拼成的树形,及其笨重和难维护。后来就想到直接生成json格式字符串,页面上也好展现。
// 首先取出每个部门的联系人
for (int i = 0; i < depList.size(); i++) {
List<Contacts> list = getContactList(depList.get(i
- Nginx安装部署
510888780
nginxlinux
Nginx ("engine x") 是一个高性能的 HTTP 和 反向代理 服务器,也是一个 IMAP/POP3/SMTP 代理服务器。 Nginx 是由 Igor Sysoev 为俄罗斯访问量第二的 Rambler.ru 站点开发的,第一个公开版本0.1.0发布于2004年10月4日。其将源代码以类BSD许可证的形式发布,因它的稳定性、丰富的功能集、示例配置文件和低系统资源
- java servelet异步处理请求
墙头上一根草
java异步返回servlet
servlet3.0以后支持异步处理请求,具体是使用AsyncContext ,包装httpservletRequest以及httpservletResponse具有异步的功能,
final AsyncContext ac = request.startAsync(request, response);
ac.s
- 我的spring学习笔记8-Spring中Bean的实例化
aijuans
Spring 3
在Spring中要实例化一个Bean有几种方法:
1、最常用的(普通方法)
<bean id="myBean" class="www.6e6.org.MyBean" />
使用这样方法,按Spring就会使用Bean的默认构造方法,也就是把没有参数的构造方法来建立Bean实例。
(有构造方法的下个文细说)
2、还
- 为Mysql创建最优的索引
annan211
mysql索引
索引对于良好的性能非常关键,尤其是当数据规模越来越大的时候,索引的对性能的影响越发重要。
索引经常会被误解甚至忽略,而且经常被糟糕的设计。
索引优化应该是对查询性能优化最有效的手段了,索引能够轻易将查询性能提高几个数量级,最优的索引会比
较好的索引性能要好2个数量级。
1 索引的类型
(1) B-Tree
不出意外,这里提到的索引都是指 B-
- 日期函数
百合不是茶
oraclesql日期函数查询
ORACLE日期时间函数大全
TO_DATE格式(以时间:2007-11-02 13:45:25为例)
Year:
yy two digits 两位年 显示值:07
yyy three digits 三位年 显示值:007
- 线程优先级
bijian1013
javathread多线程java多线程
多线程运行时需要定义线程运行的先后顺序。
线程优先级是用数字表示,数字越大线程优先级越高,取值在1到10,默认优先级为5。
实例:
package com.bijian.study;
/**
* 因为在代码段当中把线程B的优先级设置高于线程A,所以运行结果先执行线程B的run()方法后再执行线程A的run()方法
* 但在实际中,JAVA的优先级不准,强烈不建议用此方法来控制执
- 适配器模式和代理模式的区别
bijian1013
java设计模式
一.简介 适配器模式:适配器模式(英语:adapter pattern)有时候也称包装样式或者包装。将一个类的接口转接成用户所期待的。一个适配使得因接口不兼容而不能在一起工作的类工作在一起,做法是将类别自己的接口包裹在一个已存在的类中。 &nbs
- 【持久化框架MyBatis3三】MyBatis3 SQL映射配置文件
bit1129
Mybatis3
SQL映射配置文件一方面类似于Hibernate的映射配置文件,通过定义实体与关系表的列之间的对应关系。另一方面使用<select>,<insert>,<delete>,<update>元素定义增删改查的SQL语句,
这些元素包含三方面内容
1. 要执行的SQL语句
2. SQL语句的入参,比如查询条件
3. SQL语句的返回结果
- oracle大数据表复制备份个人经验
bitcarter
oracle大表备份大表数据复制
前提:
数据库仓库A(就拿oracle11g为例)中有两个用户user1和user2,现在有user1中有表ldm_table1,且表ldm_table1有数据5千万以上,ldm_table1中的数据是从其他库B(数据源)中抽取过来的,前期业务理解不够或者需求有变,数据有变动需要重新从B中抽取数据到A库表ldm_table1中。
- HTTP加速器varnish安装小记
ronin47
http varnish 加速
上午共享的那个varnish安装手册,个人看了下,有点不知所云,好吧~看来还是先安装玩玩!
苦逼公司服务器没法连外网,不能用什么wget或yum命令直接下载安装,每每看到别人博客贴出的在线安装代码时,总有一股羡慕嫉妒“恨”冒了出来。。。好吧,既然没法上外网,那只能麻烦点通过下载源码来编译安装了!
Varnish 3.0.4下载地址: http://repo.varnish-cache.org/
- java-73-输入一个字符串,输出该字符串中对称的子字符串的最大长度
bylijinnan
java
public class LongestSymmtricalLength {
/*
* Q75题目:输入一个字符串,输出该字符串中对称的子字符串的最大长度。
* 比如输入字符串“google”,由于该字符串里最长的对称子字符串是“goog”,因此输出4。
*/
public static void main(String[] args) {
Str
- 学习编程的一点感想
Cb123456
编程感想Gis
写点感想,总结一些,也顺便激励一些自己.现在就是复习阶段,也做做项目.
本专业是GIS专业,当初觉得本专业太水,靠这个会活不下去的,所以就报了培训班。学习的时候,进入状态很慢,而且当初进去的时候,已经上到Java高级阶段了,所以.....,呵呵,之后有点感觉了,不过,还是不好好写代码,还眼高手低的,有
- [能源与安全]美国与中国
comsci
能源
现在有一个局面:地球上的石油只剩下N桶,这些油只够让中国和美国这两个国家中的一个顺利过渡到宇宙时代,但是如果这两个国家为争夺这些石油而发生战争,其结果是两个国家都无法平稳过渡到宇宙时代。。。。而且在战争中,剩下的石油也会被快速消耗在战争中,结果是两败俱伤。。。
在这个大
- SEMI-JOIN执行计划突然变成HASH JOIN了 的原因分析
cwqcwqmax9
oracle
甲说:
A B两个表总数据量都很大,在百万以上。
idx1 idx2字段表示是索引字段
A B 两表上都有
col1字段表示普通字段
select xxx from A
where A.idx1 between mmm and nnn
and exists (select 1 from B where B.idx2 =
- SpringMVC-ajax返回值乱码解决方案
dashuaifu
AjaxspringMVCresponse中文乱码
SpringMVC-ajax返回值乱码解决方案
一:(自己总结,测试过可行)
ajax返回如果含有中文汉字,则使用:(如下例:)
@RequestMapping(value="/xxx.do") public @ResponseBody void getPunishReasonB
- Linux系统中查看日志的常用命令
dcj3sjt126com
OS
因为在日常的工作中,出问题的时候查看日志是每个管理员的习惯,作为初学者,为了以后的需要,我今天将下面这些查看命令共享给各位
cat
tail -f
日 志 文 件 说 明
/var/log/message 系统启动后的信息和错误日志,是Red Hat Linux中最常用的日志之一
/var/log/secure 与安全相关的日志信息
/var/log/maillog 与邮件相关的日志信
- [应用结构]应用
dcj3sjt126com
PHPyii2
应用主体
应用主体是管理 Yii 应用系统整体结构和生命周期的对象。 每个Yii应用系统只能包含一个应用主体,应用主体在 入口脚本中创建并能通过表达式 \Yii::$app 全局范围内访问。
补充: 当我们说"一个应用",它可能是一个应用主体对象,也可能是一个应用系统,是根据上下文来决定[译:中文为避免歧义,Application翻译为应
- assertThat用法
eksliang
JUnitassertThat
junit4.0 assertThat用法
一般匹配符1、assertThat( testedNumber, allOf( greaterThan(8), lessThan(16) ) );
注释: allOf匹配符表明如果接下来的所有条件必须都成立测试才通过,相当于“与”(&&)
2、assertThat( testedNumber, anyOf( g
- android点滴2
gundumw100
应用服务器android网络应用OSHTC
如何让Drawable绕着中心旋转?
Animation a = new RotateAnimation(0.0f, 360.0f,
Animation.RELATIVE_TO_SELF, 0.5f, Animation.RELATIVE_TO_SELF,0.5f);
a.setRepeatCount(-1);
a.setDuration(1000);
如何控制Andro
- 超简洁的CSS下拉菜单
ini
htmlWeb工作html5css
效果体验:http://hovertree.com/texiao/css/3.htmHTML文件:
<!DOCTYPE html>
<html xmlns="http://www.w3.org/1999/xhtml">
<head>
<title>简洁的HTML+CSS下拉菜单-HoverTree</title>
- kafka consumer防止数据丢失
kane_xie
kafkaoffset commit
kafka最初是被LinkedIn设计用来处理log的分布式消息系统,因此它的着眼点不在数据的安全性(log偶尔丢几条无所谓),换句话说kafka并不能完全保证数据不丢失。
尽管kafka官网声称能够保证at-least-once,但如果consumer进程数小于partition_num,这个结论不一定成立。
考虑这样一个case,partiton_num=2
- @Repository、@Service、@Controller 和 @Component
mhtbbx
DAOspringbeanprototype
@Repository、@Service、@Controller 和 @Component 将类标识为Bean
Spring 自 2.0 版本开始,陆续引入了一些注解用于简化 Spring 的开发。@Repository注解便属于最先引入的一批,它用于将数据访问层 (DAO 层 ) 的类标识为 Spring Bean。具体只需将该注解标注在 DAO类上即可。同时,为了让 Spring 能够扫描类
- java 多线程高并发读写控制 误区
qifeifei
java thread
先看一下下面的错误代码,对写加了synchronized控制,保证了写的安全,但是问题在哪里呢?
public class testTh7 {
private String data;
public String read(){
System.out.println(Thread.currentThread().getName() + "read data "
- mongodb replica set(副本集)设置步骤
tcrct
javamongodb
网上已经有一大堆的设置步骤的了,根据我遇到的问题,整理一下,如下:
首先先去下载一个mongodb最新版,目前最新版应该是2.6
cd /usr/local/bin
wget http://fastdl.mongodb.org/linux/mongodb-linux-x86_64-2.6.0.tgz
tar -zxvf mongodb-linux-x86_64-2.6.0.t
- rust学习笔记
wudixiaotie
学习笔记
1.rust里绑定变量是let,默认绑定了的变量是不可更改的,所以如果想让变量可变就要加上mut。
let x = 1; let mut y = 2;
2.match 相当于erlang中的case,但是case的每一项后都是分号,但是rust的match却是逗号。
3.match 的每一项最后都要加逗号,但是最后一项不加也不会报错,所有结尾加逗号的用法都是类似。
4.每个语句结尾都要加分