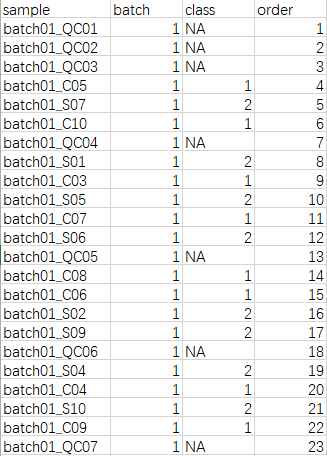
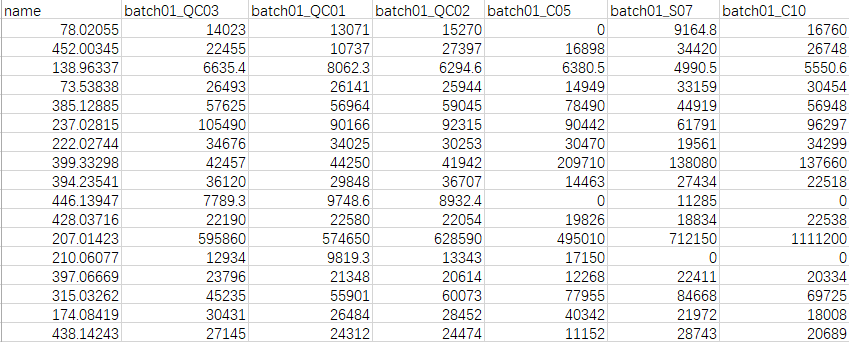
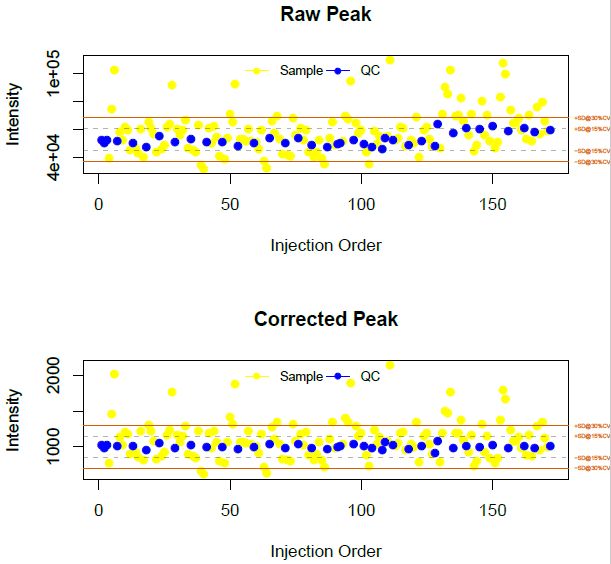
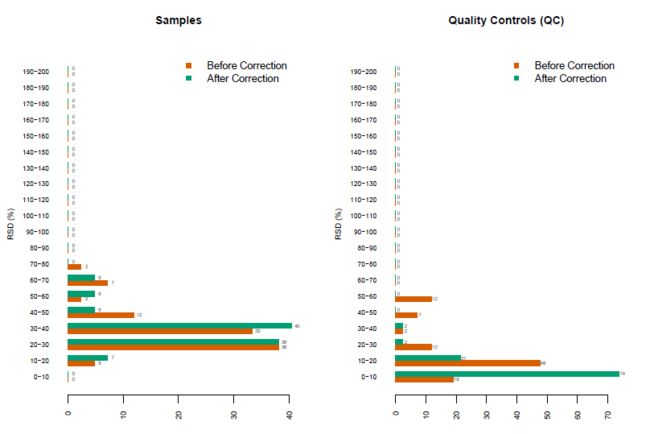
- 编写简单的小程序
又熟了
Python入门学习pythonflask
编写简单的小程序文章目录编写简单的小程序1.turtle的认识与使用1.1turtle常用的函数1.2用turtle画小蛇1.3begin_fill和end_fill绘制太阳花2.变量2.1变量的创建2.2命名规则2.3保留字及查看方法3.运算符3.1算数运算符3.2关系运算符3.3逻辑运算符4.注释与缩进5.赋值语句6.输出与输入7.数据类型7.1字符串的索引7.2列表8.字符编码8.2乱码问题
- 以光盘读写系统演示面向对象设计的原则与方法
CoderIsArt
C++11设计模式面向对象
面向对象设计(OOD)是软件开发中的核心方法,强调通过对象、类、继承、封装和多态等概念来构建系统。以下是面向对象设计的原则、方法及常用技术手段:一、面向对象设计原则(SOLID原则)单一职责原则(SRP,SingleResponsibilityPrinciple)一个类应只有一个职责,即只负责一项功能。优点:提高类的内聚性,降低耦合性,便于维护和扩展。开放-封闭原则(OCP,Open-Closed
- LeetCode 21Merge Two Sorted Lists 合并两个排序链表 Java
我欲混吃与等死
LeetCodeleetcode链表java
题目:将两个已排序的链表合并在一起。举例1:输入:list1=[1,2,4],list2=[1,3,4];输出:[1,1,2,3,4,4];举例2:输入:list1=[],list2=[];输出:[]举例3:输入:list1=[],list2=[0];输出:[0]解题思路:遍历两个链表,比较节点值来合并链表,当其中一个链表遍历完成时,将另一个链表剩余部分拼入新链表。/***Definitionfo
- Leetcode 160 Intersection of Two Linked Lists
xxxmmc
leetcode算法双指针
题意给定两个链表,找这两个链表第一个公共节点,如果没有返回nullptr题目链接https://leetcode.com/problems/intersection-of-two-linked-lists/description/题解两个指针分别从两个链表(记录为表A,表B)的表头出发,并且记录到表尾移动的步数,得到两个指针移动的步数之差xxx。步数之差为正数,那么把表A的指针移动xxx步,否则移
- 后端登录校验(一)——会话技术
develop_lost
服务器运维
一、登录校验1.什么是登录校验登录校验是服务器端对浏览器端(就是客户端)发送的请求进行校验。校验用户的登录情况,如果用户已经登录,则允许进行用户需要的业务操作,反之则禁止用户操作业务操作,并返回给用户一个错误的提示,让客户端跳转到的登录页面,进行登录操作。2.如何进行登录校验-先认清http协议:http协议是无状态协议,无状态是客户端的每一次请求都是独立的,这次的请求不会携带上一次的数据,服务器
- Java后端开发技术详解
小二爱编程·
java开发语言
Java作为一门成熟的编程语言,已广泛应用于后端开发领域。其强大的生态系统和广泛的支持库使得Java成为许多企业和开发者的首选后端开发语言。随着云计算、微服务架构和大数据技术的兴起,Java后端开发的技术栈也不断演进。本文将详细介绍Java后端开发的核心技术,包括Java基础、常见框架、数据库操作、缓存技术、异步编程等。1.Java基础:理解面向对象的编程Java是一种面向对象的编程语言,面向对象
- 基于springboot的社区团购系统设计
Olivia-gogogo
springboot后端java
一、引言在当今数字化时代,信息技术正以前所未有的速度渗透到社会的各个领域,深刻地改变着人们的生活和工作方式。教育领域也不例外,随着高等教育的普及和招生规模的不断扩大,大学生入学审核工作面临着越来越大的挑战。传统的人工入学审核方式已难以满足现代教育管理的需求,暴露出诸多弊端。传统人工入学审核方式效率低下。在每年的招生季,高校招生工作人员需要面对大量的入学申请材料,这些材料不仅数量庞大,而且种类繁多,
- Midscene.js自然语言驱动的网页自动化全指南
Hello.Reader
前端技术人工智能javascript自动化开发语言
一、概述网页自动化在数据抓取、UI测试和业务流程优化中发挥着重要作用。然而,传统工具如Selenium和Puppeteer要求用户具备编程技能,编写复杂的选择器和脚本维护成本高昂。Midscene.js通过自然语言接口革新了这一领域,用户只需描述任务(如“点击登录按钮”或“提取产品价格”),AI即可自动执行,大幅降低技术门槛。Midscene.js由web-infra-dev团队开发,开源于Git
- 【Pandas】pandas Series plot.box
liuweidong0802
PandasSeriespandas信息可视化数据分析
Pandas2.2SeriesPlotting方法描述Series.plot([kind,ax,figsize,…])用于绘制Series对象的数据可视化图表Series.plot.area([x,y,stacked])用于绘制堆叠面积图(StackedAreaPlot)Series.plot.bar([x,y])用于绘制垂直条形图(VerticalBarPlot)Series.plot.barh
- Vue 表单输入绑定,双向绑定
天马3798
Vuevue.jsjavascript前端Vue表单输入绑定Vue表单双向绑定
一、v-model指令用于Vue表单双向绑定用户Vue组件属性双向绑定v-model还可以用于各种不同类型的输入,、元素。它会根据所使用的元素自动使用对应的DOM属性和事件组合:文本类型的和元素会绑定valueproperty并侦听input事件;和会绑定checkedproperty并侦听change事件;会绑定valueproperty并侦听change事件。二、v-model使用案例文本姓名
- 使用Jupyter Notebook进行深度学习编程 - 深度学习教程
shandianfk_com
ChatGPTAIjupyter深度学习ide
大家好,今天我们要聊聊如何使用JupyterNotebook进行深度学习编程。深度学习是人工智能领域中的一项重要技术,通过模仿人脑神经网络的方式进行学习和分析。JupyterNotebook作为一个强大的工具,可以帮助我们轻松地进行深度学习编程,尤其适合初学者和研究人员。本文将带领大家一步步了解如何在JupyterNotebook中开展深度学习项目。一、什么是JupyterNotebook?Jup
- 【Linux】Hadoop-3.4.1的伪分布式集群的初步配置
孤独打铁匠Julian
Linuxlinuxhadoopubuntu
配置步骤一、检查环境JDK#目前还是JDK8最适合Hadoopjava-versionecho$JAVA_HOMEHadoophadoopversionecho$HADOOP_HOME二、配置SSH免密登录Hadoop需要通过SSH管理节点(即使在伪分布式模式下)sudoaptinstallopenssh-server#安装SSH服务(如未安装)cd~/.ssh/ssh-keygen-trsa#生
- **[特殊字符] 计算机领域创作挑战赛,邀你共绘文字海洋!**
爱编程的Loren
活动文章活动文章
亲爱的大学博主们: 大家好! 你是否热爱写作,渴望在文字的海洋中遨游,展示自己的创作才华?你是否对计算机领域充满热情,希望将你的知识和见解分享给更多人?如果你对这两点都给出了肯定的答案,那么这个创作活动就是为你量身定制的! **创作挑战赛火热开启** 我们诚挚地邀请你参加为期14天的创作挑战赛!这是一个以写作博客为目的的创作活动,旨在鼓励大学生博主们挖掘自己的创作潜能,展现自己的写作才
- 秒杀场景的设计思考
思无邪6675
后端
秒杀场景的设计思考在学习Redis的之后,一个绕不开的话题就是秒杀系统的设计。本文将从下面几个方面展开一下个人简单的理解:秒杀场景的介绍设计的核心思路怎么限流、削峰、异步planB总结秒杀场景的介绍秒杀场景是大家常说的高并发场景,但是实际上其与单纯的高并发还有一点不同,主要区别就是其流量来的猛增,几乎是一个垂直的增长,而非线性增长的并发。其具有如下特点:瞬时高并发读多写少不能超卖设计的核心思路在
- Spring Boot 项目 90% 存在这 15 个致命漏洞,你的代码在裸奔吗?
风象南
原创随笔javaspringboot后端web安全系统安全
文章首发公众号【风象南】SpringBoot作为一款广泛使用的Java开发框架,虽然为开发者提供了诸多便利,但也并非无懈可击,其安全漏洞问题不容忽视。本文将深入探讨SpringBoot常见的安全漏洞类型、产生原因以及相应的解决方案,帮助开发者更好地保障应用程序的安全。1.SQL注入漏洞漏洞描述:当应用程序使用用户输入的数据来构建SQL查询时,如果没有进行适当的过滤或转义,攻击者就可以通过构造恶意的
- golang jwt挖坑
qiang527052
golang个人笔记golangjwt
golangjwt使用golangjwt使用中遇到的一个坑,特此记录。具体描述:因为公司需要,现有架构jwt生成token的代码是java实现的,然后现在在golang中需要对此token进行解析。java用到的jar包:io.jsonwebtoken.jjwt0.9.0golang用到的库:github.com/dgrijalva/jwt-gojava生成token测试代码如下:publicst
- 「MySQL」日期时间格式化函数 DATE_FORMAT() 的使用详解
m0_74823827
mysqladbandroid
目录一、DATE_FORMAT()语法二、格式化字符串详解三、常见日期时间格式组合四、业务场景五、总结一、DATE_FORMAT()语法DATE_FORMAT()是MySQL中用于格式化日期时间的函数:语法:DATE_FORMAT(date,format_string)date:需要格式化的日期化时间值,一般是需要被格式化的日期时间类型(datetime类),但也可以是日期时间形式的字符串form
- SQL-触发器(trigger)的详解以及代码演示
m0_74823827
面试学习路线阿里巴巴sql数据库服务器
一、触发器的概念触发器是一种特殊的存储过程,但是触发器不存在输入和输出参数,所以不能被显式的去调用,而是与特定的表相关联,当表中的数据发生变化时,触发器被激活并执行其定义的SQL代码。触发器可以是行级触发器(针对每一行数据变化)或语句级触发器(针对整个SQL语句的变化),行级触发器允许对每一行单独处理,而语句级触发器则在整个操作完成后执行一次。触发器的功能强化约束:触发器能够实现比CHECK语句更
- Linux动静态库
遥逖
Linuxlinux运维服务器
Linux动静态库静态库动态库库的链接静态库核心特性:链接时机:在编译时将库代码直接嵌入到可执行文件中。文件格式:.a(Linux)、.lib(Windows)。内存占用:每个使用该库的可执行文件都会包含一份完整的库代码副本。创建静态库(.a文件)编写源代码:首先,编写你需要的库函数的源代码文件,例如mylib.c编译为目标文件:使用gcc/g++编译源码为目标文件(.o文件)g++-cmylib
- 华山论剑,大模型(deepseek qwq gemini)辩论人生意义
Lifeng66666666
语言模型语言模型人工智能
借助DeepDiscussion程序,通过让大模型(deepseekqwqgemini)讨论“人生意义是什么”这一挑战问题,我们得以客观观察目前这几种大模型的价值观,能力,不足。部分讨论过程:问题:人生的意义是什么?deepseek/deepseek-r1:free初始方案:针对“人生的意义是什么”这一终极问题,我的解决方案分为以下五个层次,融合东西方哲学智慧与实践心理学,并提供具体行动方向:一、
- 【el-cascader 级联选择器】懒加载 - 适用新增、编辑表单(编辑表单有初始数据)
Fighting_p
elementUIvue代码记录vue.jsjavascript前端
目录代码效果代码constant/formList.jsexportconstoperationFormDesc=(_this)=>{return{rule:{type:'cascader',label:'违规情况',layout:12,required:true,attrs:{'collapse-tags':true,props:{lazy:true,//multiple:true,/****【
- H743定时器输出PWM波方法(基于STM32CubeMX)
NW嵌入式开发
单片机开发stm32定时器PWM模式
0工具准备1.STM32CubeMX2.《STM32H743参考手册中文版》3.《stm32h743xi数据手册》1前言本文介绍基于STM32CubeMX,使用stm32h743xi,使用TIM15的通道2输出PWM波的方法。2H743定时器输出PWM波方法(基于STM32CubeMX)《STM32H743参考手册中文版》对PWM模式的描述如下:其中,有关PWM模式1和模式2的介绍见TIM15_C
- B3843 [GESP202306 三级] 密码合规
一台Redmi Note 12 Pro
算法c++数据结构
题目描述网站注册需要有用户名和密码,编写程序以检查用户输入密码的有效性。合规的密码应满足以下要求:。只能由a∼z之间26个小写字母、A∼Z之间26个大写字母、0∼9之间10个数字以及!@#$四个特殊字符构成。密码最短长度:6个字符,密码最大长度:12个字符。大写字母,小写字母和数字必须至少有其中两种,以及至少有四个特殊字符中的一个。输入格式输入一行不含空格的字符串。约定长度不超过100。该字符串被
- Opencv之计算机视觉一
闭月之泪舞
计算机视觉计算机视觉opencvpython
一、环境准备使用opencv库来实现简单的计算机视觉。需要安装两个库:opencv-python和opencv-contrib-python,版本可以自行选择,注意不同版本的opencv中的某些函数名和用法可能不同pipinstallopencv-python==3.4.18.65-ihttps://pypi.tuna.tsinghua.edu.cn/simplepipinstallopencv-
- STM8L1xx利用定时器实现毫秒和微妙延时
荣070214
STM8单片机单片机毫秒和微妙延时
采用单片机的定时计数器进行毫秒和微妙级延时,精度较准。检测溢出时产生的标志位来判断延时到达。下面以STM8L101芯片为例及配合代码说明。一、实现原理:1、初始化Timer2时钟源(附上相应代码)voidTIM2Init(void){TIM2_DeInit();CLK_PeripheralClockConfig(CLK_Peripheral_TIM2,ENABLE);TIM2->CR1&=((ui
- 大语言模型微调和大语言模型应用的区别?
AI Echoes
人工智能深度学习deepseek机器学习算法
大语言模型微调和大语言模型应用的区别?关键要点微调大型语言模型(LLM)是调整预训练模型以适应特定任务或领域的过程,研究表明这能显著提升性能。大型语言模型应用是指将LLM用于实际问题解决或任务执行,如聊天机器人或文本生成。微调和应用的不同在于:微调是准备阶段,应用是最终使用;使用微调模型通常在特定领域表现更好,而预训练模型更适合通用任务。什么是微调大型语言模型?微调大型语言模型是指取一个已经预训练
- 计算机视觉总结
Trank-Lw
计算机视觉深度学习人工智能
以下是针对上述问题的详细解答,并结合代码示例进行说明:1.改进YOLOv5人脸检测模块,复杂光照场景准确率从98.2%提升至99.5%优化具体过程:光照补偿:在数据预处理阶段,采用自适应光照补偿算法,对图像进行实时增强,以减少光照变化对人脸检测的影响。数据增强:在训练数据中增加复杂光照场景下的样本,如强光、弱光、背光等,通过数据增强提高模型对不同光照条件的适应性。模型调整:对YOLOv5模型的网络
- golang slice 切片数组下标的截取方式
用户昵称不能为空
golang开发语言后端
需求使用golang截取的下标,按照下标顺序截取代码//Youcaneditthiscode!//Clickhereandstarttyping.packagemainimport"fmt"funcRows(page,pageSizeint)(items[]int){alls:=[
- jupyter的一些操作
翻滚吧键盘
Debian12使用日记jupyteridepython
Linux,mac中可以直接通过在输入代码的地方输入pip等终端命令,并可以执行运行所有cell“Cell"菜单,然后选择“RunAll”选项来运行全部代码
- 自动驾驶系统的车辆动力学建模:自行车模型与汽车模型的对比分析
赛卡
自动驾驶自动驾驶数学建模pythonnumpymatplotlib算法
在自动驾驶系统的车辆动力学建模中,自行车模型(BicycleModel)和更复杂的汽车模型(如双轨模型或多体动力学模型)各有其适用场景和优缺点。以下是两者的详细对比及选择原因解析:1.模型定义与核心差异特性自行车模型复杂汽车模型(如双轨模型)简化假设将四轮车辆简化为两轮(前轮转向,后轮驱动)考虑四轮独立运动、悬架系统、轮胎侧偏特性自由度2-3自由度(位置x,y,航向角θ)6+自由度(含横向、俯仰、
- eclipse maven
IXHONG
eclipse
eclipse中使用maven插件的时候,运行run as maven build的时候报错
-Dmaven.multiModuleProjectDirectory system propery is not set. Check $M2_HOME environment variable and mvn script match.
可以设一个环境变量M2_HOME指
- timer cancel方法的一个小实例
alleni123
多线程timer
package com.lj.timer;
import java.util.Date;
import java.util.Timer;
import java.util.TimerTask;
public class MyTimer extends TimerTask
{
private int a;
private Timer timer;
pub
- MySQL数据库在Linux下的安装
ducklsl
mysql
1.建好一个专门放置MySQL的目录
/mysql/db数据库目录
/mysql/data数据库数据文件目录
2.配置用户,添加专门的MySQL管理用户
>groupadd mysql ----添加用户组
>useradd -g mysql mysql ----在mysql用户组中添加一个mysql用户
3.配置,生成并安装MySQL
>cmake -D
- spring------>>cvc-elt.1: Cannot find the declaration of element
Array_06
springbean
将--------
<?xml version="1.0" encoding="UTF-8"?>
<beans xmlns="http://www.springframework.org/schema/beans"
xmlns:xsi="http://www.w3
- maven发布第三方jar的一些问题
cugfy
maven
maven中发布 第三方jar到nexus仓库使用的是 deploy:deploy-file命令
有许多参数,具体可查看
http://maven.apache.org/plugins/maven-deploy-plugin/deploy-file-mojo.html
以下是一个例子:
mvn deploy:deploy-file -DgroupId=xpp3
- MYSQL下载及安装
357029540
mysql
好久没有去安装过MYSQL,今天自己在安装完MYSQL过后用navicat for mysql去厕测试链接的时候出现了10061的问题,因为的的MYSQL是最新版本为5.6.24,所以下载的文件夹里没有my.ini文件,所以在网上找了很多方法还是没有找到怎么解决问题,最后看到了一篇百度经验里有这个的介绍,按照其步骤也完成了安装,在这里给大家分享下这个链接的地址
- ios TableView cell的布局
张亚雄
tableview
cell.imageView.image = [UIImage imageNamed:[imageArray objectAtIndex:[indexPath row]]];
CGSize itemSize = CGSizeMake(60, 50);
&nbs
- Java编码转义
adminjun
java编码转义
import java.io.UnsupportedEncodingException;
/**
* 转换字符串的编码
*/
public class ChangeCharset {
/** 7位ASCII字符,也叫作ISO646-US、Unicode字符集的基本拉丁块 */
public static final Strin
- Tomcat 配置和spring
aijuans
spring
简介
Tomcat启动时,先找系统变量CATALINA_BASE,如果没有,则找CATALINA_HOME。然后找这个变量所指的目录下的conf文件夹,从中读取配置文件。最重要的配置文件:server.xml 。要配置tomcat,基本上了解server.xml,context.xml和web.xml。
Server.xml -- tomcat主
- Java打印当前目录下的所有子目录和文件
ayaoxinchao
递归File
其实这个没啥技术含量,大湿们不要操笑哦,只是做一个简单的记录,简单用了一下递归算法。
import java.io.File;
/**
* @author Perlin
* @date 2014-6-30
*/
public class PrintDirectory {
public static void printDirectory(File f
- linux安装mysql出现libs报冲突解决
BigBird2012
linux
linux安装mysql出现libs报冲突解决
安装mysql出现
file /usr/share/mysql/ukrainian/errmsg.sys from install of MySQL-server-5.5.33-1.linux2.6.i386 conflicts with file from package mysql-libs-5.1.61-4.el6.i686
- jedis连接池使用实例
bijian1013
redisjedis连接池jedis
实例代码:
package com.bijian.study;
import java.util.ArrayList;
import java.util.List;
import redis.clients.jedis.Jedis;
import redis.clients.jedis.JedisPool;
import redis.clients.jedis.JedisPoo
- 关于朋友
bingyingao
朋友兴趣爱好维持
成为朋友的必要条件:
志相同,道不合,可以成为朋友。譬如马云、周星驰一个是商人,一个是影星,可谓道不同,但都很有梦想,都要在各自领域里做到最好,当他们遇到一起,互相欣赏,可以畅谈两个小时。
志不同,道相合,也可以成为朋友。譬如有时候看到两个一个成绩很好每次考试争做第一,一个成绩很差的同学是好朋友。他们志向不相同,但他
- 【Spark七十九】Spark RDD API一
bit1129
spark
aggregate
package spark.examples.rddapi
import org.apache.spark.{SparkConf, SparkContext}
//测试RDD的aggregate方法
object AggregateTest {
def main(args: Array[String]) {
val conf = new Spar
- ktap 0.1 released
bookjovi
kerneltracing
Dear,
I'm pleased to announce that ktap release v0.1, this is the first official
release of ktap project, it is expected that this release is not fully
functional or very stable and we welcome bu
- 能保存Properties文件注释的Properties工具类
BrokenDreams
properties
今天遇到一个小需求:由于java.util.Properties读取属性文件时会忽略注释,当写回去的时候,注释都没了。恰好一个项目中的配置文件会在部署后被某个Java程序修改一下,但修改了之后注释全没了,可能会给以后的参数调整带来困难。所以要解决这个问题。
&nb
- 读《研磨设计模式》-代码笔记-外观模式-Facade
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
/*
* 百度百科的定义:
* Facade(外观)模式为子系统中的各类(或结构与方法)提供一个简明一致的界面,
* 隐藏子系统的复杂性,使子系统更加容易使用。他是为子系统中的一组接口所提供的一个一致的界面
*
* 可简单地
- After Effects教程收集
cherishLC
After Effects
1、中文入门
http://study.163.com/course/courseMain.htm?courseId=730009
2、videocopilot英文入门教程(中文字幕)
http://www.youku.com/playlist_show/id_17893193.html
英文原址:
http://www.videocopilot.net/basic/
素
- Linux Apache 安装过程
crabdave
apache
Linux Apache 安装过程
下载新版本:
apr-1.4.2.tar.gz(下载网站:http://apr.apache.org/download.cgi)
apr-util-1.3.9.tar.gz(下载网站:http://apr.apache.org/download.cgi)
httpd-2.2.15.tar.gz(下载网站:http://httpd.apac
- Shell学习 之 变量赋值和引用
daizj
shell变量引用赋值
本文转自:http://www.cnblogs.com/papam/articles/1548679.html
Shell编程中,使用变量无需事先声明,同时变量名的命名须遵循如下规则:
首个字符必须为字母(a-z,A-Z)
中间不能有空格,可以使用下划线(_)
不能使用标点符号
不能使用bash里的关键字(可用help命令查看保留关键字)
需要给变量赋值时,可以这么写:
- Java SE 第一讲(Java SE入门、JDK的下载与安装、第一个Java程序、Java程序的编译与执行)
dcj3sjt126com
javajdk
Java SE 第一讲:
Java SE:Java Standard Edition
Java ME: Java Mobile Edition
Java EE:Java Enterprise Edition
Java是由Sun公司推出的(今年初被Oracle公司收购)。
收购价格:74亿美金
J2SE、J2ME、J2EE
JDK:Java Development
- YII给用户登录加上验证码
dcj3sjt126com
yii
1、在SiteController中添加如下代码:
/**
* Declares class-based actions.
*/
public function actions() {
return array(
// captcha action renders the CAPTCHA image displ
- Lucene使用说明
dyy_gusi
Lucenesearch分词器
Lucene使用说明
1、lucene简介
1.1、什么是lucene
Lucene是一个全文搜索框架,而不是应用产品。因此它并不像baidu或者googleDesktop那种拿来就能用,它只是提供了一种工具让你能实现这些产品和功能。
1.2、lucene能做什么
要回答这个问题,先要了解lucene的本质。实际
- 学习编程并不难,做到以下几点即可!
gcq511120594
数据结构编程算法
不论你是想自己设计游戏,还是开发iPhone或安卓手机上的应用,还是仅仅为了娱乐,学习编程语言都是一条必经之路。编程语言种类繁多,用途各 异,然而一旦掌握其中之一,其他的也就迎刃而解。作为初学者,你可能要先从Java或HTML开始学,一旦掌握了一门编程语言,你就发挥无穷的想象,开发 各种神奇的软件啦。
1、确定目标
学习编程语言既充满乐趣,又充满挑战。有些花费多年时间学习一门编程语言的大学生到
- Java面试十问之三:Java与C++内存回收机制的差别
HNUlanwei
javaC++finalize()堆栈内存回收
大家知道, Java 除了那 8 种基本类型以外,其他都是对象类型(又称为引用类型)的数据。 JVM 会把程序创建的对象存放在堆空间中,那什么又是堆空间呢?其实,堆( Heap)是一个运行时的数据存储区,从它可以分配大小各异的空间。一般,运行时的数据存储区有堆( Heap)和堆栈( Stack),所以要先看它们里面可以分配哪些类型的对象实体,然后才知道如何均衡使用这两种存储区。一般来说,栈中存放的
- 第二章 Nginx+Lua开发入门
jinnianshilongnian
nginxlua
Nginx入门
本文目的是学习Nginx+Lua开发,对于Nginx基本知识可以参考如下文章:
nginx启动、关闭、重启
http://www.cnblogs.com/derekchen/archive/2011/02/17/1957209.html
agentzh 的 Nginx 教程
http://openresty.org/download/agentzh-nginx-tutor
- MongoDB windows安装 基本命令
liyonghui160com
windows安装
安装目录:
D:\MongoDB\
新建目录
D:\MongoDB\data\db
4.启动进城:
cd D:\MongoDB\bin
mongod -dbpath D:\MongoDB\data\db
&n
- Linux下通过源码编译安装程序
pda158
linux
一、程序的组成部分 Linux下程序大都是由以下几部分组成: 二进制文件:也就是可以运行的程序文件 库文件:就是通常我们见到的lib目录下的文件 配置文件:这个不必多说,都知道 帮助文档:通常是我们在linux下用man命令查看的命令的文档
二、linux下程序的存放目录 linux程序的存放目录大致有三个地方: /etc, /b
- WEB开发编程的职业生涯4个阶段
shw3588
编程Web工作生活
觉得自己什么都会
2007年从学校毕业,凭借自己原创的ASP毕业设计,以为自己很厉害似的,信心满满去东莞找工作,找面试成功率确实很高,只是工资不高,但依旧无法磨灭那过分的自信,那时候什么考勤系统、什么OA系统、什么ERP,什么都觉得有信心,这样的生涯大概持续了约一年。
根本不是自己想的那样
2008年开始接触很多工作相关的东西,发现太多东西自己根本不会,都需要去学,不管是asp还是js,
- 遭遇jsonp同域下变作post请求的坑
vb2005xu
jsonp同域post
今天迁移一个站点时遇到一个坑爹问题,同一个jsonp接口在跨域时都能调用成功,但是在同域下调用虽然成功,但是数据却有问题. 此处贴出我的后端代码片段
$mi_id = htmlspecialchars(trim($_GET['mi_id ']));
$mi_cv = htmlspecialchars(trim($_GET['mi_cv ']));
贴出我前端代码片段:
$.aj