最近似乎有这样一种观点,生物信息就是用来发低分水刊的,快像meta分析一样被淘汰、被嫌弃了。
对于这种看法,真正的生信分析师听到后只会莞尔一笑,「really?;are you kidding?」别问我怎么知道的,因为科研猫团队就是这么专业的团队。想做高质量的生信分析,就找科研猫,全985博士团队,人手一篇10分+,人均100+案例分析经验,绝对靠谱~
生物信息这几年发展可谓如火如荼,特别是在新的测序技术推进下,在科研领域及临床应用上都大放异彩。
科研领域中,从简单的公共数据挖掘,“短平快”地发表纯数据分析的小文章;到挖掘大型测序数据,结合实验验证的高级别CNS顶级文章,生物信息都发挥着重要作用。
临床应用上,从分析肿瘤测序数据,筛选基因突变,指导抗肿瘤靶向用药;到病原体宏基因组测序,鉴定发热待查患者的致病病原体,生物信息一直引领精准医疗的开展。不管是何领域,都可谓至关重要。
咱们今天就看一下通过挖掘TCGA数据库,3年内接连发表3篇《Nucleic Acids Research》(IF:11.1),相关研究均是同一主要完成人/作者。
3篇文献都是有关与eQTL的,那么什么是eQTL。
首先QTL(Quantitative Trait Locus)是数量性状位点,比如身高、奶牛的产奶量、癌症的生存时间等,其对应的性状的变异是一个连续的范围,其对应的控制基因的位点就是一个数量性状位点。而eQTL(expression Quantitative Trait Loci, eQTL)就是控制数量性状表达位点,即能调控数量性状基因(如身高基因)表达水平高低的那些基因的位点。可以理解为eQTL是QTL的上游调控基因,控制其表达情况。
eQTL(expression Quantitative Trait Loci, eQTL)的准确定义:是染色体上一些能特定调控mRNA和蛋白质表达水平的区域,其mRNA/蛋白质的表达水平量与数量性状成比例关系。eQTL可分为顺式作用eQTL和反式作用eQTL,顺式作用eQTL就是某个基因的eQTL定位到该基因所在的基因组区域,表明可能是该基因本身的差别引起的mRNA水平变化;反式作用eQTL是指某个基因的eQTL定位到其他基因组区域,表明其他基因的差别控制该基因mRNA水平的差异。
eQTL就是把基因表达作为一种性状,研究遗传突变与基因表达的相关性: 就好像研究遗传突变与身高的相关性一样。简单地说, 遗传学研究经常发现一些致病或易感突变,这些突变怎样导致表型有时候不太直观;所以用某个基因的差异表达作为过渡:突变A-->B基因表达变化-->表型。
早年可以通过同时做一个个体的SNP芯片和cDNA芯片, 在全基因组水平上研究突变与表达的相关性, 这种研究需要较多个体(例如1000个); 现在随着深度测序的出现,很多人开始用RNA-Seq在较少量个体中研究allele-specific expression,本质上就是eQTL。
背景知识介绍完了,我们看一下今天安排的3篇文献吧。
01
这研究由美国德州大学健康科学中心韩冷教授和华中科技大学公共卫生学院缪小平教授共同合作完成,龚静博士为第一作者。该研究报道了针对33种肿瘤类型、近万样本的eQTL分析。
全基因组关联研究(GWAS)可以为疾病或性状的遗传病因研究提供线索。分析显示,GWAS发现的与疾病相关的风险位点大多数位于基因的非编码区域并在调控区域富集,这些结果提示疾病相关位点可能主要通过调控基因表达来发挥作用。
所以越来越多的研究将遗传变异与基因表达数据进行整合,故而经常用到eQTLs分析。有研究在通过肿瘤样本和正常样本的对比之后,发现大量eQTLs具有肿瘤特异性,然而癌症的eQTL分析目前的研究领域内是非常缺乏的。
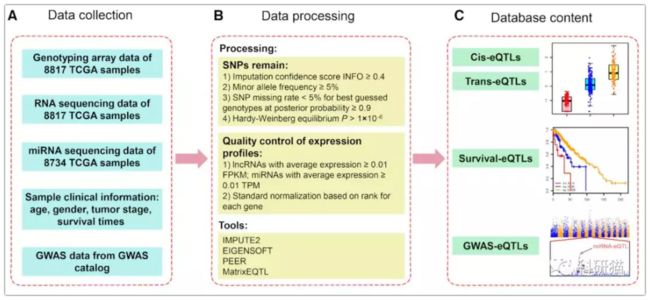
作者通过挖掘TCGA数据库中33种肿瘤类型的9196个肿瘤样本的基因型和基因表达数据,共发现了5606570个可以顺式调控基因表达的cis-eQTLs和231210个可以远程调控基因的trans-eQTLs。
另外,作者通过整合eQTLs与临床预后信息和GWAS数据,发现了22212个与生存期相关的eQTLs,以及337131与GWAS分析位点重合的eQTLs。
pancanQTL数据库概述:(A)pancanQTL四个数据集,包括cis-eQTLs,trans-eQTLs,生存相关的eQTLs和GWAS相关的eQTLs。(B)PancanQTL的搜索框。(C)cis-eQTLs 数据集中eQTL示意图。(D)与生存时间相关的eQTL Kaplan-Meier plot示意图。图片来源:Nucleic Acids Research
作者还构建了数据库平台pancanQTL,供其他研究者免费查询、浏览。该平台上线一个月,就有来自全球13个国家近500次的数据访问。
评价:该研究可谓是高质量的数据挖掘研究。利用公共数据库中全面的表型资料和测序数据,不仅仅节约了研究的时间成本和经济成本,更是对全球已有数据库的整合和开发。该研究有助于增强遗传变异在肿瘤的发生与发展中发挥的潜在生物学功能的理解,促进遗传和癌症相关研究领域的发展。由此可见,数据挖掘才是后基因组时代的主角。
02
华中农大信息学院、生物医学与健康学院的研究团队在“Nucleic Acids Research”杂志先后发表题为「ncRNA-eQTL: a database to systematically evaluate the effects of SNPs on non-coding RNA expression across cancer types」的数据库研究。
研究团队在接受采访时打了以个这样的比喻「如果我们的遗传信息是一座山,遗传学家知道这山里面有金子,但他们用的是锄头。我们的工作就是给他们打造‘挖矿机’,帮他们把这些金子更快地挖出来;并把我们已经挖到的东西展现出来,构建成资源库,方便他们使用。」
目前虽然有一些ncRNA得到深入研究,但也仅仅是冰山一角。在该研究中,研究团队基于TCGA的癌症多组学数据和全基因组关联分析(GWAS)数据,开发了新的生物信息计算方法,系统鉴定了能影响ncRNA(包括lncRNA和miRNA)的表达数量性状基因座(ncRNA-eQTL),进一步整合了GWAS数据和临床信息,鉴定了与GWAS表型或临床信息相关的ncRNA-eQTL,并搭建了ncRNA-eQTL的数据平台。
在33种癌症中,共鉴定出到与lncRNA相关的600多万种顺式eQTLs和70多万种反式eQTLs。他们将已鉴定的eQTLs和癌症病人生存信息关联分析后,确定了与患者总生存时间相关的8235个长非编码RNA-eQTLs和116个microRNA-eQTLs。
用户可通过ncRNA-eQTL数据库查询和下载所有ncRNA-eQTL结果,包括不同癌症类型的顺式和反式ncRNA-eQTL、患者存活时间相关的ncRNA-eQTL,GWAS 相关的ncRNA-eQTL。此外,该平台提供批量查询功能,可跨癌症类型比较多个功能性ncRNA-eQTL或基因。该平台为探索SNP、非编码RNA和癌症表型之间的关联及其潜在的生物学机制提供极大便利。
评价:本篇的通讯作者就是咱们今天看的第一篇文献的first author,不难看出还是类似的思路,只是切入点不同。2017年的那篇是比较宏观分析了eQTLs和癌症表型的关系,本研究提取了ncRNA 和SNP进行着重分析,紧跟时代热点。
03
研究发现,可选择性多聚腺苷酸化(APA)可通过识别转录本上不同的PolyA加尾信号,使转录本具有不同长度的3’端非翻译区(3’UTR),从而影响其生物学功能。SNP是人类遗传变异的最常见类型,在人类复杂性状和疾病中发挥重要作用。近来发现,一些遗传变异可造成APA功能失调,APA也与癌症的发生发展密切相关。
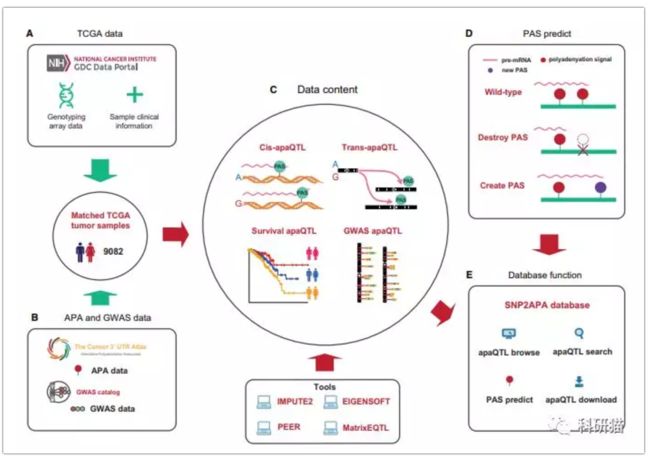
研究团队,通过整合TCGA数据库中多种癌症样本的基因型数据和APA数据,系统地鉴定了影响APA事件的遗传变异(apaQTL),并根据基因调控方式定义了顺式和反式apaQTL。
在进一步整合GWAS数据后,鉴定了落在GWAS区域的tag SNP及其连锁不平衡LD区域中的apaQTL,并利用样本的临床信息鉴定预后相关的apaQTL。研究团队搭建了SNP2APA数据库展示这些结果,同时设计了软件在线预测SNP对多聚腺苷酸化信号的影响。
评价:跟第二篇ncRNA 和SNP关联分析不同的是,这次的切入点是可选择性多聚腺苷酸化(APA)和SNP,文章主体依旧是相似的流程,换了更新的切入点。相信未来该团队会从其他的切入点,进行更多的数据挖掘和网站开发,期待他们在QTL方面更多的研究。
小结
最近不知道从哪里吹出来的风,说生物信息文章等同灌水文章。我只能说这些人真的是“一叶障目,不见泰山”。就像我们在文章中解读的这三篇文献,3年3篇NAR「核酸研究」。NAR可以说是生命科学领域内非常老牌的杂志了,能够在上面发表一篇文章可谓是很多科研人员的奋斗目标。这三篇文献中没有涉及任何实验,纯数据挖掘,不是一样发表10分+?我想,这足以给数据挖掘正名了吧。
还有人说生物信息就像前几年的Meta分析一样,用不了多久就该黄了。在这里,我们不妄议Meta分析的文章,更不会妄自菲薄。生物信息是一门专业学科,在很多高校中都设立了生物信息专业。一门学科,一个专业,你敢说它水吗?此外,需要引起我们重视的是,近几年的政府工作报告中反复强调大数据的重要性,以致相关产业得到高速发展,在生命科学和临床大数据处理中,生物信息一直扮演着核心的角色。
总而言之,灌水文处处有,文章水不水是作者的关系,跟生物信息,亦或是Meta分析无干。端正科研态度,善用研究方法,紧跟科学热点,高质量的科研成果自然水到渠成。
参考文献:
doi: 10.1093/nar/gkx861
doi: 10.1093/nar/gkz711
doi: 10.1093/nar/gkz793
部分图片来源于网络,如有侵权,请联系删除。
参考资料:
1:https://www.cnblogs.com/Acceptyly/p/3904108.html
2:https://mp.weixin.qq.com/s/Yr7Tfepuwq0BxWLB1C7nCg
3:https://mp.weixin.qq.com/s/W__R_o8UtHrbnXvmOH6RoA
4:http://news.sciencenet.cn/htmlnews/2019/10/431274.shtm
关注“科研猫”公众号,联系客服
胖雨小姐姐
or
折耳猫小姐姐
领取超多~超多~科研干货
往期干货链接
R语言从入门到精通系列
从今天开始,每天学点R语言~
R语言从入门到精通:Day1
R语言从入门到精通:Day2
R语言从入门到精通:Day3
R语言从入门到精通:Day4
R语言从入门到精通:Day5
R语言从入门到精通:Day6
R语言从入门到精通:Day7
R语言从入门到精通:Day8
R语言从入门到精通:Day9
R语言从入门到精通:Day10
R语言从入门到精通:Day11
R语言从入门到精通:Day12
科研作图系列
【国庆特辑】崛起的中国,全球70年科研成果排名
【科研猫·绘图】高级科研做图 – AI入门
【科研猫·绘图】今夏最热的“热图”
【科研猫·绘图】看·箱线图·如何美丽动人
【科研猫·绘图】优雅版·小提琴图
【科研猫·绘图】缤纷版·韦恩图
【科研猫·绘图】朋友圈最火热的“火山图”
【科研猫·绘图】bar(霸)图绘制之霸气满屏
【科研猫·绘图】GSEA分析全攻略,带视频分享
【科研猫·绘图】团团“圆圆”,来个不一样的月饼
【科研猫·绘图】献礼国庆70年,R语言绘制中国地图
【科研猫·绘图】趣味 饼图 -从“披萨”到“圆形彩虹”
网络图
从网络图探寻基因互作的蛛丝马迹(1)
从网络图探寻基因互作的蛛丝马迹(2)
从网络图探寻基因互作的蛛丝马迹(3)
从网络图探寻基因互作的蛛丝马迹(4)
从网络图探寻基因互作的蛛丝马迹(5)
生存分析系列
【科研猫】生存分析的正确姿势(1)视频+R代码
【科研猫·出品】TCGA超大批量生存分析教程
GEO数据挖掘系列
GEO数据库挖掘(1)--SCI文章速成
GEO数据库挖掘(2)--快速锁定目标数据
挖掘GEO速成SCI文章系列教程(3)-R语言基础
重磅:GEO数据库挖掘教程(4)一体化分析代码
GO/KEGG功能富集系列
3分钟了解GO/KEGG功能富集分析
干货预警:3分钟搞定GO/KEGG功能富集分析(2)
终极篇:3分钟搞定GO/KEGG功能富集分析-柱状图
终极篇:3分钟搞定GO/KEGG功能富集分析-气泡图
TCGA数据挖掘系列
隔壁实验室的“秃鹫”师兄又发SCI啦--TCGA数据挖掘实战
TCGA数据挖掘终结者:cBioPortal
更多科研新鲜资讯、文献精读和生物信息技能
请关注科研猫公众号
未经许可请勿随意转载,
版权事宜由上海辰明律师事务所提供法务支持