1. BAMStats
BAMStats是一款从BAM文件中计算覆盖度和其他数据,并生成这些数据的描述性统计的工具
1.1 安装
wget https://jaist.dl.sourceforge.net/project/bamstats/BAMStats-1.25.zip
unzip BAMStats-1.25.zip
rm -f BAMStats-1.25.zip
cd BAMStats-1.25/
1.2 使用
java -jar BAMStats-1.25.jar -h
-d, --distances: edit distance statistics(不清楚是什么指标)
-f, --features: bed or gtf
-l, --lengths: mapped read length
-m, --mapped: coverage statistics, for mapped regions only
-q, --qualities: mapping quality (MAPQ) statistics
-s, --starts: start position statistics
-v, --view: output format, 'simple' or 'html', default 'simple'
-i, --infile
-o, --outfile
1.2.1 错误信息
参考基因组的染色体数目太多(可能参考基因组上有一些染色体的小片段还没有定位)
java -jar -Xmx6g BAMStats-1.25.jar -i *.smr.bam -o test --view simple
ERROR: Sorry, 1471 are too many references to display sensibly in 'html' format. Either try 'simple', or use the GUI version.
系统设置不对,不能使用GUI
java -jar -Xmx4g BAMStats-GUI-1.25.jar
Exception in thread "AWT-EventQueue-0" java.awt.HeadlessException:
No X11 DISPLAY variable was set, but this program performed an operation which requires it.
第二个错误的解决办法见https://www.cnblogs.com/princessd8251/p/4000016.html,但需要管理员权限。这里为了演示,只取了bam的前1000000行(集中在第一条染色体),并修改header。
java -jar -Xmx6g BAMStats-1.25.jar -i head_and_part.sam -o test
lsx test
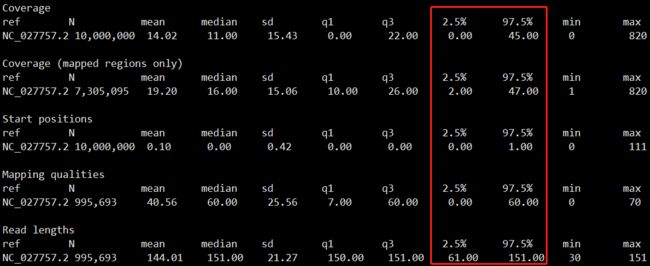
ref N mean median sd q1 q3 2.5% 97.5% min max
NC_027757.2 10,000,000 14.02 11.00 15.43 0.00 22.00 0.00 45.00 0 820
把几个选项都加上,看看增加了些什么
java -jar -Xmx6g BAMStats-1.25.jar -d -l -m -q -s -v simple -i head_and_part.sam -o test1
lsx test1
median, q1, q3是四分位数, 2.5%和97.5%也是类似的
改为网页输出,看看有什么变化
java -jar -Xmx6g BAMStats-1.25.jar -d -l -m -q -s -v html -i head_and_part.sam -o test2
cd test2.data/
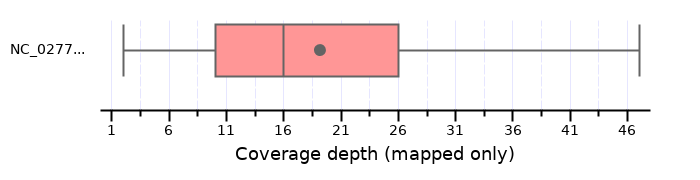
生成了很多图片和网页链接,每一个链接呈现如下的一个板块,并且同时包括直方图、累计直方图、箱线图
Coverage.html
EditDistances.html
MappedCoverage.html
MappingQuality.html
ReadLengths.html
StartPositions.html
以Coverage distribution (mapped only)为例
1.3 注意
- BAMStats通常需要分配2至6GB的内存(Java程序可以使用-Xmx来设置)
- SAM或者BAM格式均可,但需要排序
2. samtools depth/stats
2.1 samtools depth得到每一个碱基上的测序深度,可以加上bed文件
samtools depth -b bed *.smrb.bam > bedbase_depth.txt
chr1 1146705 104
chr1 1146706 107
chr1 1146707 107
chr1 1146708 107
chr1 1146709 109
然后自己编写脚本(划定指定区域进行分析),就能得到测序深度与覆盖度的关系,类似于在20X, 30X, 50X等深度下,覆盖度是百分之多少。也可以通过划定窗口的方式,看一系列窗口的平均深度,然后从整条染色体的角度分析哪块儿深度低了,哪块儿深度高了,能说明什么问题等等。
2.2 samtools stats能对比对后的bam进行很全面的统计
samtools stats *.smrb.bam > bamstat.txt
grep ^SN bamstat.txt | cut -f 2-
sequences: 134013154
reads mapped: 133144413
reads mapped and paired: 132860280
reads unmapped: 868741
reads properly paired: 123414550
reads duplicated: 13863532
还有碱基数目, average length, average quality, insert size average等等统计量
还可以得到可视化的一些结果
plot-bamstats -p ./ bamstat.txt
3. GATK DepthOfCoverage
在捕获测序的评估中比较适用,因为捕获测序一般有bed范围,也需要在不同深度下评价。
${java_path} -Xmx4g -jar ${gatk3_path} -T DepthOfCoverage \
-R ${ref} \
-o ${out_dir}${sample}/${sample} \
-I *.smrb.bam \
-L ${bed} \
--omitDepthOutputAtEachBase --omitIntervalStatistics \
-ct 1 -ct 5 -ct 10 -ct 20 -ct 30 -ct 50 -ct 100 可以随意设置,且注意是大于号
会生成四个文件
sample_cumulative_coverage_counts
sample_cumulative_coverage_proportions
sample_statistics
sample_summary
重点看一下summary文件
sample_id total mean granular_third_quartile granular_median granular_first_quartile %_bases_above_1 %_bases_above_5 %_bases_above_10 %_bases_above_20 %_bases_above_30 %_bases_above_50 %_bases_above_100
17L0292145 417397895 461.76 500 443 322 99.9 99.7 99.6 99.4 99.2 98.7 97.1
Total 417397895 461.76 N/A N/A N/A
4. bamdst
这个之前总结过,https://www.jianshu.com/p/ac7b8e4d76e4
reference
http://bamstats.sourceforge.net/