第9章 方差分析
名义型或有序因子也可作为预测变量进行建模。当包含的因子是解释变量时,关注的重点通常从预测转向组别差异的分析,这种分析方法称作**方差分析(ANOVA)。
9.1 术语速成
9.2 ANOVA模型拟合
ANOVA也是广义线性模型的特例,可以用lm()函数分析ANOVA模型。不过本章基本使用aov()函数。两者结果等同。
9.2.1 aov( )函数
aov( )函数的语法为`aov(formula, data = dataframe)。
9.2.2 表达式中各项的顺序
基础性的效应需要放在表达式前面。首先是协变量,然后是主效应,接着是双因素的交互项,再接着是三因素的交互项,以此类推。
car包中的Anova()函数(不要与标准anova()函数混淆)提供了类型Ⅱ和类型Ⅲ方法的选项,而aov()函数使用的是类型Ⅰ的方法。
9.3 单因素方差分析
单因素方差分析中,比较分类因子定义的两个或多个组别中的因变量均值。
代码清单9-1 单因素方差分析
> library(multcomp)
> attach(cholesterol)
> table(trt)
trt
1time 2times 4times drugD drugE
10 10 10 10 10
> aggregate(response, by=list(trt), FUN=mean)
Group.1 x
1 1time 5.78197
2 2times 9.22497
3 4times 12.37478
4 drugD 15.36117
5 drugE 20.94752
> aggregate(response, by=list(trt), FUN=sd)
Group.1 x
1 1time 2.878113
2 2times 3.483054
3 4times 2.923119
4 drugD 3.454636
5 drugE 3.345003
> fit <- aov(response ~ trt)
> summary(fit)
Df Sum Sq Mean Sq F value Pr(>F)
trt 4 1351.4 337.8 32.43 9.82e-13 ***
Residuals 45 468.8 10.4
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> library(gplots)
> plotmeans(response ~ trt, xlab="Treatment", ylab="Response",
+ main="Mean Plot\nwith 95% CI")
> detach(cholesterol)
ANOVA对治疗方式( trt)的F检验非常显著(p<0.0001),说明五种疗法的效果不同。
gplots包中的plotmeans()可绘制带有置信区间的组均值图形。
9.3.1 多重比较
探究各变量之间的不同。TukeyHSD()函数提供了对各组均值差异的成对检验。
代码清单9-2 Tukey HSD的成对组间比较
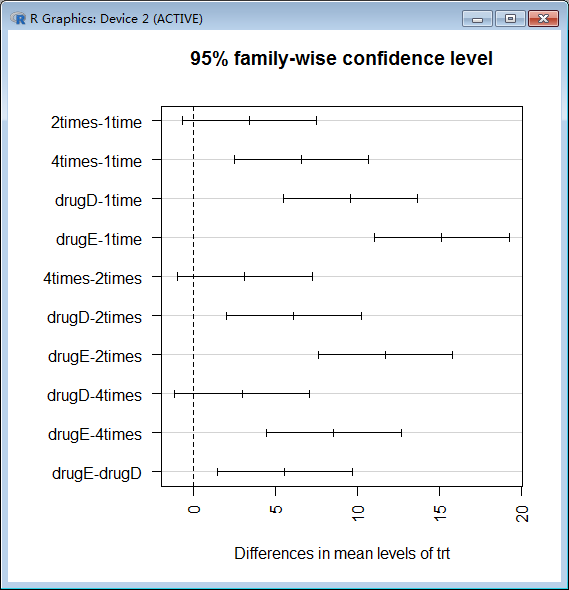
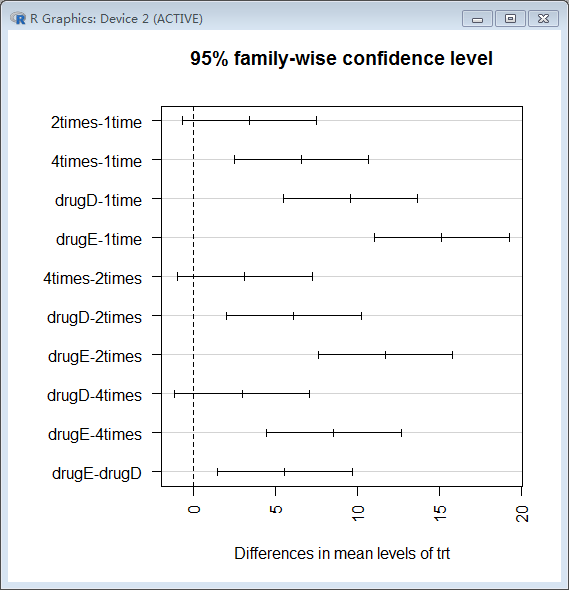
> TukeyHSD(fit)
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = response ~ trt)
$trt
diff lwr upr p adj
2times-1time 3.44300 -0.6582817 7.544282 0.1380949
4times-1time 6.59281 2.4915283 10.694092 0.0003542
drugD-1time 9.57920 5.4779183 13.680482 0.0000003
drugE-1time 15.16555 11.0642683 19.266832 0.0000000
4times-2times 3.14981 -0.9514717 7.251092 0.2050382
drugD-2times 6.13620 2.0349183 10.237482 0.0009611
drugE-2times 11.72255 7.6212683 15.823832 0.0000000
drugD-4times 2.98639 -1.1148917 7.087672 0.2512446
drugE-4times 8.57274 4.4714583 12.674022 0.0000037
drugE-drugD 5.58635 1.4850683 9.687632 0.0030633
> par(las=2) #标签文字方向
> par(mar=c(5, 8, 4, 2)) #图形边距:下、左、上、右
> plot(TukeyHSD(fit))
1time和2times的均值差异不显著( p=0.138),而1time和4times间的差异非常显著(p<0.001)。
图中置信区间包含0组别说明差异不显著(p>0.05)。
multcomp包中的glht()函数提供了多重均值比较更为全面的方法,适用于线性模型及广义线性模型。下面的代码重现了Tukey HSD检验,并用不同图形展示结果。
> library(multcomp)
> par(mar=c(5, 4, 6, 2))
> tuk <- glht(fit, linfct=mcp(trt="Tukey"))
有相同字母的组说明均值差异不显著。
从结果看,一天四次5mg剂量比一天一次20mg效果更佳。
9.3.2 评估检验的假设条件
单因素方差分析中,假设因变量服从正态分布,各组方差相等。结果的有效性取决于统计检验时数据满足假设条件的程度。
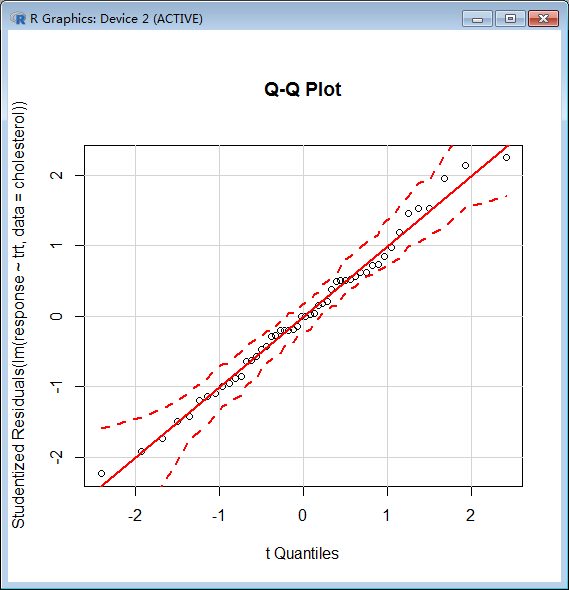
Q-Q图可用来检验正态性假设,若数据落在95%的置信区间内,说明满足正态性假设。
> library(car)
> qqPlot(lm(response ~ trt, data=cholesterol),
+ simulate=TRUE, main="Q-Q Plot", labels=FALSE)
R提供了一些方差齐性检验的函数。如下代码做Bartlett检验:
> bartlett.test(response ~ trt, data=cholesterol)
Bartlett test of homogeneity of variances
data: response by trt
Bartlett's K-squared = 0.57975, df = 4, p-value = 0.9653
p=0.97,五组方差无显著不同。
其他检验如Fligner-Killeen检验(fligner.test()函数)和Brown-Forsythe检验(HH包中的hov()函数)。它们的检验结果与Bartlett检验相同。
方差齐性分析对离群点非常敏感。car包中的outlierTest()函数可检测离群点:
> library(car)
> outlierTest(fit)
No Studentized residuals with Bonferonni p < 0.05
Largest |rstudent|:
rstudent unadjusted p-value Bonferonni p
19 2.251149 0.029422 NA
当p>1时产生NA,因此说明数据中没有离群点。
综上,根据Q-Q图、Bartlett检验和离群点检验,该数据似乎可以用ANOVA模型很好地拟合。这些方法证明了结果的可靠。
9.4 单因素协方差分析
单因素协方差分析(ANCOVA)扩展了单因素方差分析(ANOVA),包含一个或多个定量的协变量。
代码清单9-3 单因素ANCOVA
> data(litter, package="multcomp")
> table(dose)
dose
0 5 50 500
20 19 18 17
> aggregate(litter$weight, by=list(litter$dose), FUN=mean) #此处用数据框$变量名
Group.1 x
1 0 32.30850
2 5 29.30842
3 50 29.86611
4 500 29.64647
> fit <- aov(litter$weight ~ litter$gesttime + dose)
> summary(fit)
Df Sum Sq Mean Sq F value Pr(>F)
litter$gesttime 1 134.3 134.30 8.049 0.00597 **
dose 3 137.1 45.71 2.739 0.04988 *
Residuals 69 1151.3 16.69
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
利用table()函数可以看到,不同剂量下产仔数不同。
aggregate()函数获得各组均值,未用药幼崽体重均值最高。
ANCOVA的F检验说明:gesttime与weight有关,控制gesstime,dose与weight有关。
由于使用了协变量,可以求去除协变量效应后的组均值。使用effects包中的effects()函数来计算调整的均值:
> library(effects)
> effect("litter$dose", fit)
Error in model.frame.default(litter$weight ~ litter$gesttime + litter$dose + :
参数'litter'的种类(list)不对
本例中,调整的均值与aggregate()函数得出的未调整的均值类似。
F检验表明了不同处理方式造成体重均值不同,但无法表明哪种处理方式与其他方式不同。使用multcomp包对所有均值进行比较。multcomp包还可以检验用户自定义的均值假设。
代码清单9-4 对用户定义的对照的多重比较
> library(multcomp)
> contrast <- rbind("no drug vs. drug" = c(3, -1, -1, -1))
> summary(glht(fit, linfct=mcp(dose=contrast)))
Error in mcp2matrix(model, linfct = linfct) :
Variable(s) ‘dose’ have been specified in ‘linfct’ but cannot be found in ‘model’!
> #遇错未实现
9.4.1 评估检验的假设条件
ANCOVA与ANOVA相同,都需要正态性和同方差性假设,9.3.2中方法可以检验这些假设条件。
另外ANCOVA还假订回归斜率相同。模型包含交互项时,可对回归斜率的同质性进行检验。
代码清单9-5 检验回归斜率的同质性
> library(multcomp)
> fit2 <- aov(weight ~ gesttime*dose, data=litter)
> summary(fit2)
Df Sum Sq Mean Sq F value Pr(>.4.2 koF)
gesttime 1 134.3 134.30 8.289 0.00537 **
dose 3 137.1 45.71 2.821 0.04556 *
gesttime:dose 3 81.9 27.29 1.684 0.17889
Residuals 66 1069.4 16.20
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
交互效应不显著,支持了斜率相等的假设。
若假设不成立,可尝试变换协变量或因变量,或使用能对每个斜率独立解释的模型,或使用不需假设回归斜率同质性的非参数ANCOVA方法。sm包中的sm.ancova()函数为后者提供了一个例子。
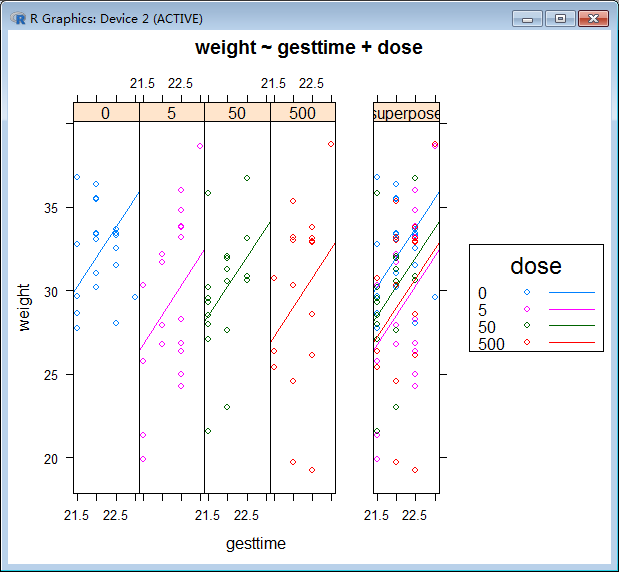
9.4.2 结果可视化
HH包中的ancova()函数可以绘制因变量、协变量和因子之间的关系图。例如代码:
> library(HH)
> ancova(weight ~ gesttime + dose, data=litter)
Analysis of Variance Table
Response: weight
Df Sum Sq Mean Sq F value Pr(>F)
gesttime 1 134.30 134.304 8.0493 0.005971 **
dose 3 137.12 45.708 2.7394 0.049883 *
Residuals 69 1151.27 16.685
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
9.5 双因素方差分析
在双因素方差分析中,受试者被分配到两因子的交叉类别组中。
> attach(ToothGrowth)
> table(supp, dose)
dose
supp 0.5 1 2
OJ 10 10 10
VC 10 10 10
> aggregate(len, by=list(supp, dose), FUN=mean)
Group.1 Group.2 x
1 OJ 0.5 13.23
2 VC 0.5 7.98
3 OJ 1.0 22.70
4 VC 1.0 16.77
5 OJ 2.0 26.06
6 VC 2.0 26.14
> aggregate(len, by=list(supp, dose), FUN=sd)
Group.1 Group.2 x
1 OJ 0.5 4.459709
2 VC 0.5 2.746634
3 OJ 1.0 3.910953
4 VC 1.0 2.515309
5 OJ 2.0 2.655058
6 VC 2.0 4.797731
> fit <- aov(len ~ supp*dose)
> summary(fit)
Df Sum Sq Mean Sq F value Pr(>F)
supp 1 205.4 205.4 12.317 0.000894 ***
dose 1 2224.3 2224.3 133.415 < 2e-16 ***
supp:dose 1 88.9 88.9 5.333 0.024631 *
Residuals 56 933.6 16.7
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
table语句表明该设计为均衡设计(各设计单元中样本大小相同),aggregate获得各单元均值和标准差。summary()函数得到方差分析表,可以看到主效应(supp和dose)和交互效应都非常显著。
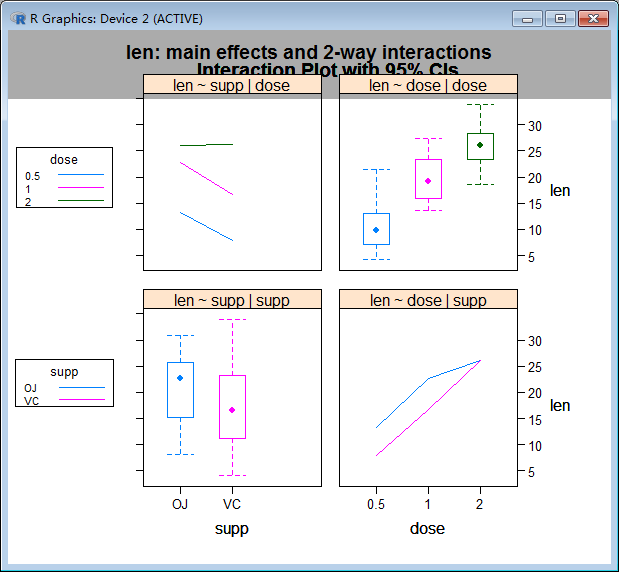
有多种结果可视化处理,
interaction.plot()函数
> interaction.plot(dose, supp, len, type="b", col=c("red", "blue"), pch=c(16, 18), main="Interaction between Dose and Supplement Type")
gplots包中plotmeans()函数
> library(gplots)
> plotmeans(len ~ interaction(supp, dose, sep=" "),
+ connect=list(c(1, 3, 5),c(2, 4, 6)), #1,3,5为一组连线
+ col=c("red","darkgreen"),
+ main = "Interaction Plot with 95% CIs",
+ xlab="Treatment and Dose Combination")
最后,还可用HH包中的interaction2wt()函数可视化结果:
> library(HH)
> interaction2wt(len~supp*dose)
9.6 重复测量方差分析
所谓重复测量方差分析,即受试者被测量不止一次。本节重点关注含一个组内和一个组间因子的重复测量方差分析。
代码清单9-7 含一个组内因子和一个组间因子的重复测量方差分析
> CO2$conc <- factor(CO2$conc)
> wlbl <- subset(CO2, Treatment='chilled')
> fit <- aov(uptake ~ (conc*Type) + Error(Plant/(conc)), wlbl)
> summary(fit)
Error: Plant
Df Sum Sq Mean Sq F value Pr(>F)
Type 1 3366 3366 22.49 0.00079 ***
Residuals 10 1497 150
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Error: Plant:conc
Df Sum Sq Mean Sq F value Pr(>F)
conc 6 4069 678.1 101.322 < 2e-16 ***
conc:Type 6 374 62.4 9.324 3.23e-07 ***
Residuals 60 402 6.7
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
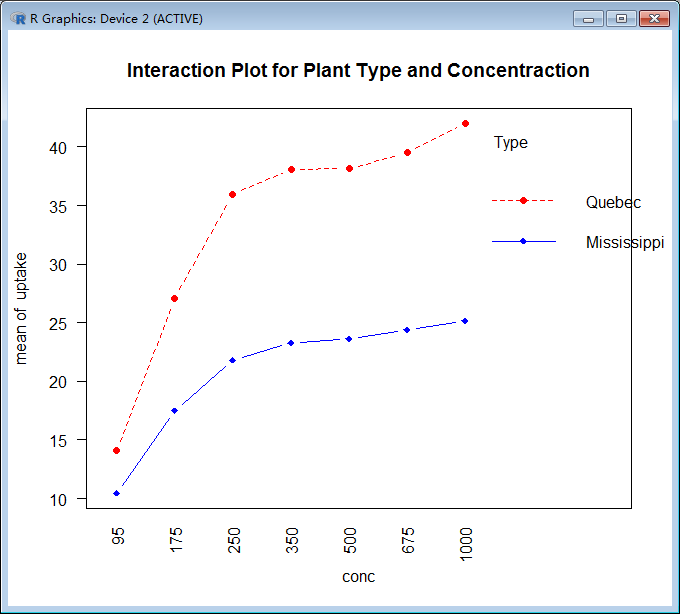
> par(las=2)
> par=(mar=c(10, 4, 4, 2))
> with(wlbl, interaction.plot(conc, Type, uptake, type="b", col=c("red", "blue"), pch=c(16, 18), main="Interaction Plot for Plant Type and Concentraction"))
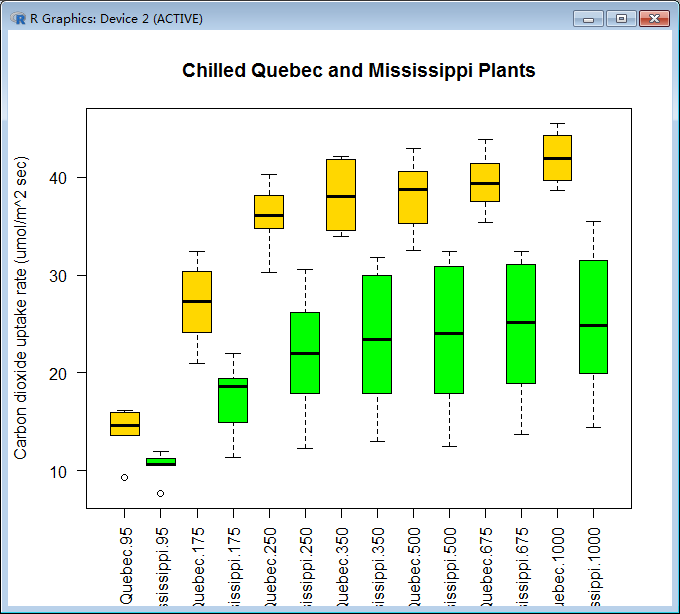
> boxplot(uptake ~ Type*conc, data=wlbl, col=(c("gold", "green")),
+ main = "Chilled Quebec and Mississippi Plants",
+ ylab = "Carbon dioxide uptake rate (umol/m^2 sec)")
方差分析表明在0.01水平下,主效应类型和浓度以及交叉效应类型x浓度都非常显著。
通常处理的数据集是宽格式(wide format),即列是变量,行是观测值,而且一行一个受试对象。9.4节中litter数据库即为此类。
不过在处理重复测量设计时,需要有长格式(long format)数据才能拟合模型。长格式中,因变量的每次测量都要放到它独有的行中,CO2数据集即是该种形式。5.6.3节中的reshape包可方便地将数据转换为相应的格式。
以上分析均针对单个因变量的情况。
9.7 多元方差分析
当因变量(结果变量)不止一个时,可用多元方差分析(MANOVA)对它们同时进行分析。
代码清单9-8 单因素多元方差分析
> library(MASS)
> attach(UScereal)
> y <- cbind(calories, fat, sugars)
> aggregate(y, by=list(shelf), FUN=mean)
Group.1 calories fat sugars
1 1 119.4774 0.6621338 6.295493
2 2 129.8162 1.3413488 12.507670
3 3 180.1466 1.9449071 10.856821
> cov(y)
calories fat sugars
calories 3895.24210 60.674383 180.380317
fat 60.67438 2.713399 3.995474
sugars 180.38032 3.995474 34.050018
> fit <- manova(y ~ shelf)
> summary(fit)
Df Pillai approx F num Df den Df Pr(>F)
shelf 1 0.19594 4.955 3 61 0.00383 **
Residuals 63
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> summary.aov(fit)
Response calories :
Df Sum Sq Mean Sq F value Pr(>F)
shelf 1 45313 45313 13.995 0.0003983 ***
Residuals 63 203982 3238
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Response fat :
Df Sum Sq Mean Sq F value Pr(>F)
shelf 1 18.421 18.4214 7.476 0.008108 **
Residuals 63 155.236 2.4641
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Response sugars :
Df Sum Sq Mean Sq F value Pr(>F)
shelf 1 183.34 183.34 5.787 0.01909 *
Residuals 63 1995.87 31.68
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
manova()函数能对组间差异进行多元检验。
summary.aov()函数对每一个变量做单因素方差分析。
9.7.1 评估假设检验
单因素多元方差分析有两个前提假设:
- 多元正态性(因变量组成的向量服从多元正态分布),可用Q-Q图检验。
- 方差-协方差矩阵同质性(各组的协方差矩阵相同),通常用Box's M检验,该函数自寻。
代码清单9-9 检验多元正态性
> center <- colMeans(y)
> n <- nrow(y)
> p <- ncol(y)
> cov <- cov(y)
> d <- mahalanobis(y, center, cov)
> coord <- qqplot(qchisq(ppoints(n), df=p),
+ d, main="Q-Q Plot Assessing Multivariate Normality",
+ ylab="Mahalanobis D2")
> abline(a=0, b=1)
> identify(coord$x, coord$y, labels=row.names(UScereal))
若数据服从多元正态分布,那么点将落在斜率为1、截距项为0的直线上。
9.7.2 稳健多元方差分析
如果多元正态性或者方差-协方差均值假设都不满足,或担心多元离群点。可以考虑用稳健或非参数版本的MANOVA检验。稳健单因素MANOVA可通过rrcov包中的Wilks.test()函数实现。vegan包中的adonis()函数提供了非参数的MANOVA的等同形式。下面是Wilks.test()的应用。
代码清单9-10 稳健单因素MANOVA
fit.lm <- lm(response ~ trt, data=cholesterol)
summary(fit.lm)
contrasts(cholesterol$trt)
#系统bug无数据
9.8 用回归来做ANOVA
9.2节提到ANOVA和回归都是广义线性模型的特例。因此本章所有的设计都可用lm()函数来分析。但是,为更好理解结果,需明白在拟合模型是,R是如何理解类别型变量的。
> #aov()函数拟合模型
> fit.aov <- aov(response ~ trt, data=cholesterol)
> summary(fit)
Df Pillai approx F num Df den Df Pr(>F)
shelf 2 0.4021 5.1167 6 122 0.0001015 ***
Residuals 62
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> #lm()函数拟合模型
> fit.lm <- lm(response ~ trt, data=cholesterol)
> summary(fit.lm)
Call:
lm(formula = response ~ trt, data = cholesterol)
Residuals:
Min 1Q Median 3Q Max
-6.5418 -1.9672 -0.0016 1.8901 6.6008
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 5.782 1.021 5.665 9.78e-07 ***
trt2times 3.443 1.443 2.385 0.0213 *
trt4times 6.593 1.443 4.568 3.82e-05 ***
trtdrugD 9.579 1.443 6.637 3.53e-08 ***
trtdrugE 15.166 1.443 10.507 1.08e-13 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 3.227 on 45 degrees of freedom
Multiple R-squared: 0.7425, Adjusted R-squared: 0.7196
F-statistic: 32.43 on 4 and 45 DF, p-value: 9.819e-13
因为线性模型要求预测变量是数值型,当lm()函数遇到因子时,会用一系列与因子水平对应的数值对照变量来代替因子。如果因子有k个水平,将会创建k-1个对照变量。R提供了五种创建对照变量的内置方法。默认情况下,对照处理用于无序因子,正交多项式用于有序因子。
contrasts()函数查看编码过程:
> contrasts(cholesterol$trt)
2times 4times drugD drugE
1time 0 0 0 0
2times 1 0 0 0
4times 0 1 0 0
drugD 0 0 1 0
drugE 0 0 0 1
修改lm()中默认的对照方法。例如使用Helmert对照:
> fit.lm <- lm(response ~ trt, data=cholesterol, contrasts="contr.helmert")
> #修改
> options(contrasts = c("contr.SAS", "contr.helmert"))
9.9 小结
基本实验和准实验设计的分析方法:ANOVA/ANCOVA/MANOVA。组内和组间设计,单因素ANOVA、单因素ANCOVA、双因素ANOVA、重复测量ANOVA和单因素MANOVA。
下章介绍功效分析,功效分析可以帮助我们在给定置信度的情况下,判断达到要求效果所需样本大小。
——2017.3.7
——于510