原文题目:
Integrative Genomic Analysis of Cholangiocarcinoma Identifies Distinct IDH-Mutant Molecular Profiles
简介:
Farshidfar等人提出了TCGA的胆管癌标记分析。通过多平台分析,他们发现了一个独特的亚型丰富的IDH突变体。该亚型显示线粒体增多,染色质修饰基因表达减少,包括ARID1A潜在的表观遗传沉默。其他IDH突变型肝癌在分子结构上类似于胆管癌。
重点:
1.IDH突变体CCAs具有不同的mRNA、拷贝数和DNA甲基化的功能。
2.IDH突变体CCAs具有高线粒体和低染色质修饰基因表达。
3.IDH突变体CCAs甲基化了ARID1A启动子,表达量较低。
4.其他IDH突变型肝癌与CCA具有多平台相似性。
介绍:
胆管癌(CCAs)是一组胆道上皮细胞(胆管细胞)恶性肿瘤,包括肝内、肝门周和肝外胆道树发生的侵袭性癌。在现在标准化疗情况下,大多数终末期病人平均生存期不到1年。由于肿瘤的高复发率,即使是接受了明显治愈性切除手术的患者,其预后也很差。尽管肝内(iCCA)、肝门周和肝外CCAs具有相同的形态学特征,并在传统的临床试验中被聚集,但很明显,不同解剖位置的肿瘤在肿瘤生物学和遗传学上存在重要差异。进一步阐明这些异质肿瘤的分子改变,并在每个解剖组中发现有意义的亚型,是开发更合理、更特异和更有效治疗方法的重要步骤。
胆管癌是第二常见的原发性肝肿瘤,其病因有:血吸虫感染、原发性硬化性胆管炎、乙型或丙型肝炎病毒(HBV/HCV)感染、胆结石、先天性胆道畸形、肝硬化和接触芳香毒素等。另外,吸烟、酗酒、糖尿病等也是肝内胆管癌的高危因素。考虑到影响突变谱的危险因素的多样性和CCA亚型的不同细胞起源,对胆道癌变的机制仍有不完全的认识。
先前的研究表明,iCCAs在上皮癌中具有一定特异性,其异柠檬酸脱氢酶1和2 (IDH1/IDH2)基因的错义突变率相对较高,此类酶参与编码代谢酶,在中心碳代谢中相互转换异柠檬酸和α-酮戊二酸。这些突变在急性髓系白血病、低级别胶质瘤和胶质母细胞瘤以及软骨肉瘤中也很常见,它们发生在特定的位置,并提高新形态酶活性,导致产生高水平的代谢物(R)-2-羟基戊二酸盐(2HG) 。2HG不参与正常的代谢过程,而是干扰利用-酮戊二酸作为辅酶的酶的功能,包括组蛋白和DNA去甲基酶,并抑制线粒体电子传递链。鉴于这种复杂性,有必要在iCCA中建立一个更综合的分子改变模型,以更好地定义致癌回路,并推导出一个分类系统,将肿瘤分组为具有生物学意义的亚型,可用于指导治疗。
在本研究中,我们描述了38例CCA(血吸虫阴性、非肝内CCA、非肝炎)的分子特征,并将疾病分为几组。我们发现一类CCAs具有不同的转录组、拷贝数和甲基化特征,这些特征被高度富集IDH突变样本。我们还确定了我们在外部数据集中验证的途径和甲基化模式,这可能有助于开发更有效的治疗方法。最后,CCA与胰腺癌和肝癌(HCC)的跨平台比较进一步强调了不同肿瘤亚型的存在。
方法及结果:
1.样本
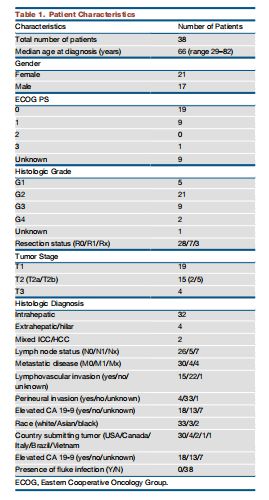
该研究分析了38个CCA的样本(限制非肝内、非血吸虫、非HBV/HCV等条件),这种相对整体的样品均匀性使我们样品集中已知的和潜在的分子异质性来源最小化。
TCGA分析由以下平台组成:全外显子组测序;Affymetrix SNP6.0拷贝数数组;RNA测序(RNA-seq),包括miRNA和lncRNA;DNA甲基化;并利用192个抗体的反相蛋白阵列(RPPA)。
2.基因水平突变、融合及拷贝数改变
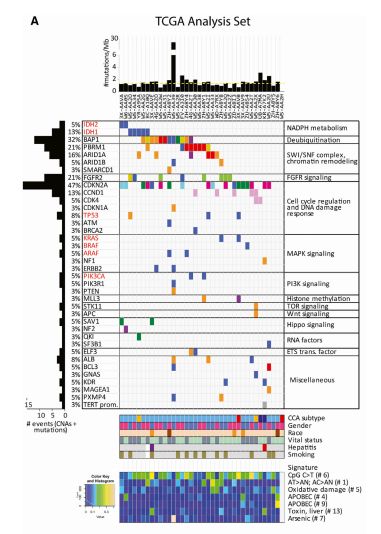
首先注释了特异基因的改变。全外显子组测序发现了2831个体细胞突变,包括1869个非沉默编码突变。目标捕获,43个选定基因的深度测序验证证实了77个突变,并新发现9个。与TCGA评估的其他癌症相比,该CCA突变率为中等,并与胰腺导管腺癌(PDAC)类似。
与之前的研究一致,我们确定了肿瘤抑制基因ARID1A、ARID1B、BAP1, PBRM1、TP53、STK11和PTEN的失活突变,癌基因IDH1、IDH2、KRAS、BRAF和PIK3CA中的热点获得功能突变。
3.过滤正常肝基因,找到一个IDH突变富集的mRNA亚群
接下来我们通过无监督的层次聚类分析mRNA的表达,只选择最可变的2%。由此产生的两个聚类显示了基因的大差异表达(图A),并具有肝脏相关特征的强富集。然而,这些聚类与任何其他分子或临床参数无关。
鉴于大多数肝脏信号基因在正常肝脏中的表达水平比肿瘤高1000 - 20000倍,我们认为肝脏信号表达在一定程度上反映了即使是少量正常肝脏的污染。与这一假设相一致,组织学分析显示肝高聚类中正常肝污染有升高的趋势(图C),而基于DNA的肿瘤纯度估计没有发现这一趋势。这表明高表达的基质基因可能混淆了mRNA的表达聚类,尤其是在只使用最顶端的可变基因时。
为减少影响,我们过滤掉386个来自GTEx正常组织表达的基因。对剩下的15427基因进行主成分分析(PCA),发现了3个集群(图A、B)。
通过正交偏最小二乘判别分析(OPLSDA),确定了1150个基因的一个子集对聚类分离的贡献最大(图A)。有趣的是,cluster 1包括所有7例IDH1或IDH2热点突变,而cluster 2富含肝外或肝门周CCA, cluster 3包含全部5个FGFR2融合。通过与之前生成的微阵列基因表达数据集GSE26566的比较,验证了这一点。
对数据集之间共享的976个最强鉴别基因进行层次聚类(图C)。在这个包含40个样本的验证队列中,确定了三个与TCGA数据集集群相似的主要集群。值得注意的是,大多数IDH1/2突变位于与富含IDH突变体的TCGA cluster 1最相似的簇中。因此,肝脏特征的去除揭示了以生物学相关方式分离样本的转录簇。
4.IDH突变子群富集了线粒体和色素修饰物的特征
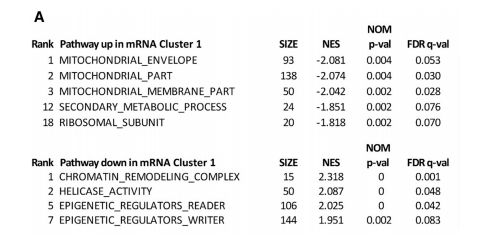
接下来我们对mRNA簇进行基因集富集分析(GSEA),发现在IDH突变cluster 1中涉及线粒体结构和功能的基因表达丰富。这包括编码柠檬酸循环酶、线粒体核糖体蛋白、电子传递链成分和线粒体结构成分的基因显著上调,与氧化磷酸化和线粒体生物发生控制的改变一致。(以下统称线粒体基因表达)。
线粒体基因高表达与IDH和PBRM1突变样本显著相关,与FGFR2融合样本低表达显著相关(图B,图D为浓缩的“线粒体评分”);
为了保持线粒体基因差异表达的潜在功能相关性,我们确定了一个相对较高的线粒体拷贝数在IDH突变体样本中,FGFR2融合样本的数量较低(图3E)。对GSE26566数据集的检查提供了这些发现的外部验证,再次确认了线粒体基因标志高表达的肿瘤中IDH突变体的富集(图3)。
GSEA还发现了线粒体基因修饰物在IDH突变的mRNA集群中显著低表达。(图A)
5.聚类分析确定了四个子组
接下来,我们将其他平台(拷贝数、甲基化、miRNA、lncRNA和蛋白质)纳入聚类分配(COCAs)中。
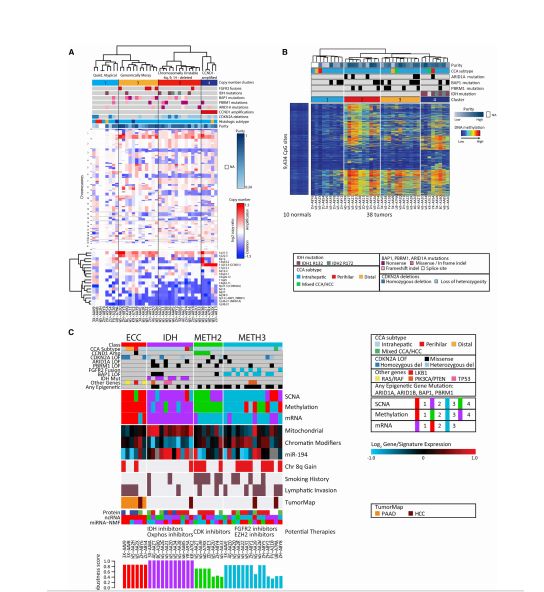
首先,基于SNP阵列杂交数据的拷贝数(图A)显示了四个基因组簇。
接下来,使用显示癌症特异性DNA甲基化变化的CpG位点对样本进行无监督聚类,确定CCA队列中的四个亚组(图B)。
我们观察到,拷贝数和甲基化簇一般与图3(上图)中识别的mRNA簇相匹配,表明数据能够检测共享的生物学机制。
我们使用mRNA、拷贝数和甲基化来进行古柯分析(COCA),以识别生物学上一致的聚类。我们优化了一个四集群解决方案(参见实验),而不是由任何一个平台所主导的(图C)。
6.在IDH突变体中,ARID1A启动子高甲基化和低表达
我们研究了IDH突变体特异性DNA高甲基化是否可能针对IDH古柯亚型中表达减少的基因。
将IDH突变体高甲基化位点与IDH古柯亚型中表达较低的基因交叉,筛选两个平台之间的基因特异性耐蚀关系,我们确定了24个基因,推测其表达受IDH突变体高甲基化调控(图A和图B)。为了验证这个列表,我们将它与匹配的对象交叉引用CCA甲基化数据集GSE32079和表达数据集GSE26566。
7.与线粒体基因修饰物标志相关的lncRNAs
8.与HCC及PDAC的比较
参考来源:生信技能树
友情链接:
课程分享
生信技能树全球公益巡讲
(https://mp.weixin.qq.com/s/E9ykuIbc-2Ja9HOY0bn_6g)
B站公益74小时生信工程师教学视频合辑
(https://mp.weixin.qq.com/s/IyFK7l_WBAiUgqQi8O7Hxw)
招学徒:
(https://mp.weixin.qq.com/s/KgbilzXnFjbKKunuw7NVfw)
欢迎关注公众号:青岛生信菜鸟团