叶绿体基因组类的文章做系统发育分析通常会用到三个数据集:
- 完整的叶绿体基因组序列
- 蛋白编码基因的CDS序列
- 基因间隔区和内含子区
比如论文Completion of the chloroplast genomes of five chines Juglans and their contribution to chloroplast phylogeny 中系统发育分析部分提到 The analyses were carried out based on the following three datasets: (1) the complete cp DNA sequences; (2) protein coding sequences; (3) the introns and spacers.
更新20190930
终于找到一款软件可以非常方便的完成这件事 PhyloSuite
软件下载地址 https://github.com/dongzhang0725/PhyloSuite/releases/tag/1.1.16
通过微信搜索 PhyloSuite应该可以找到很多比较全面的教程,都是软件的作者写的,这篇文章主要记录使用这款软件提取叶绿体基因组的蛋白编码基因,基因间区
- 首先准备需要的叶绿体基因组genbank格式的注释文件
(这款软件可以输入序列号批量获取genbank文件,具体使用方法可以自行搜索)
放到同一个文件夹下
打开软件
File —— import files
Choose Files
将genbank文件读入进来
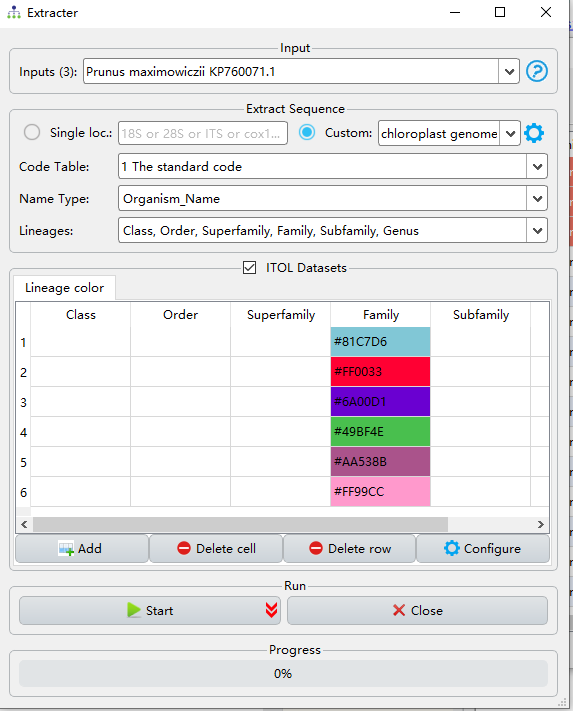
然后把文件选中,点击右键可以选择Extract
将Custom处改为chloroplast genome即可,然后点击start。结果就自动保存啦!
有需要的同学赶紧试一下吧!
欢迎大家关注我的公众号
小明的数据分析笔记本
我们自己手中的数据通常是完整的叶绿体基因组序列和对应的注释文件,以genbank文件为例,自己之前获得CDS序列的方式是 利用 http://www.bioinformatics.org/sms2/ 这个在线工具的 Genbank feature extractor 功能,然后再对结果文件做简单的处理;如何获得内含子区和基因间隔区的序列是一直困扰自己的问题。之前想写一个简单的脚本来做这件事但是一直没有想没明白如何根据注释文件获得基因间隔区和内含子区的位置坐标。今天尝试了一下Geneious这款软件,发现可以直接利用这款软件获得基因间隔区和蛋白编码基因的序列,简单记录:
打开软件后
- File——New folder 新建一个文件夹并对其命名
- 单击选中新建的文件夹后依次点击File——import导入自己的genbank注释文件
-
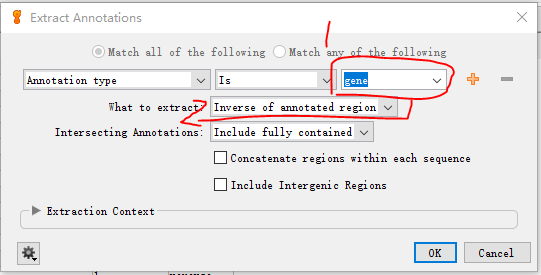
单击选中导入的文件后依次点击tools——extraction annotations
1处下拉箭头可以选提取的类型是gene,CDS,trna,rrna;如果选择的是gene,在2处可以选Annotated Region 或者 Inverse of annotated region,如果选择的是inverse的话提取的应该就是基因间隔区的序列
问题:1处对应的下拉箭头可以选的提取类型不包括intron,如何获得intron的序列自己还没有想到比较容易的办法,目前能想到的做法是:因为叶绿体基因包含内含子的并不多,大概范围可能在10~20个之间,所以手动提取应该也不会浪费很多时间。
未完,想到好的办法提取intron的序列后再来补充
更新
简单的python脚本 根据genbank文件提取CDS序列并翻译成蛋白质
之前自己的解决办法一直是利用上文提到的网址,也尝试过写一个简单的python脚本,但是一直没有能够实现,今天脑子一闪,突然看懂了 组学大讲堂 公众号 分享的文章 NCBI批量下载数据,省时又省力 里 第三部分内容 从 genbank文件里提取序列的内容,简单记录:
使用Biopython的SeqIO模块解析genbank文件,形成一个可迭代的对象,这个对象有也个features的属性,返回的是包含很多数据的一个列表,其中extract用该是一个方法,用来提取序列,同时还有id,location,type,qualifiers;qualifiers是一个字典,里面也有好多数据。(关于python里的属性和方法还有其他基本概念自己还得好好看一下!)
from Bio import SeqIO
for record in SeqIO.parse("Malus_baccata.gb","gb"):
fw_CDS = open("Malus_baccata.gb.CDS","w")
fw_pep = open("Malus_baccata.gb.pep","w")
for feature in record.features:
if feature.type == 'CDS':
fw_CDS.write(">%s\n%s\n"%(feature.qualifiers['gene'][0],features.extract(record.seq)))
fw_pep.write(">%s\n%s\n"%(feature.qualifiers['gene'][0],features.qualifiers['translation'][0]))
else:
continue
fw_CDS.close()
fw_pep.close()
需要脚本的可以给我留言即可
from Bio import SeqIO
import os
file_names = os.listdir("genbank_file")
for filename in file_names:
for record in SeqIO.parse("genbank_file/"+filename,'gb'):
fw = open(filename+".CDS","w")
for feature in record.features:
if feature.type == 'CDS':
fw.write(">%s\n%s\n"%(feature.qualifiers['gene'][0],feature.extract(record.seq)))
else:
continue
fw.close()
突然想到了自己刚刚开始接触基因家族的文章的时候根据gff注释文件手动提取CDS序列的日子......
更新20181109
想到了一种方式可以得到叶绿体基因组基因间隔区的序列,基本思路是根据genbank文件获得基因的位置坐标,然后将坐标区间内的序列统一替换成某个字符(自己采用的是替换成M),最后用replace()函数将序列字符串中的M统一替换成为空字符,
替换字符串中指定位置的字符参考https://blog.csdn.net/u012063507/article/details/79271698
第一种:纯基因间隔区,不包含内含子区
from Bio import SeqIO
def sub(string,a,b):
new = []
for i in string:
new.append(i)
for point in range(a,b):
new[point] = "M"
return ''.join(new)
for rec in SeqIO.parse("Malus_baccata.gb","gb"):
sequence = str(rec.seq)
for feature in rec.features:
if feature.type == "gene":
for part in feature.location.parts:
start = part.start
end = part.end
sequence = sub(sequence,start,end)
else:
continue
final_seq = sequence.replace("M","")
print(len(final_seq))
fw = open("1.txt","w")
fw.write(">%s\n%s\n"%("intergene",final_seq))
fw.close()
第二种:内含子也包括在其中
from Bio import SeqIO
def sub(string,a,b):
new = []
for i in string:
new.append(i)
for point in range(a,b):
new[point] = "M"
return ''.join(new)
for rec in SeqIO.parse("Malus_baccata.gb","gb"):
sequence = str(rec.seq)
for feature in rec.features:
if feature.type == "CDS" or feature.type == "tRNA" or feature.type == "rRNA":
for part in feature.location.parts:
start = part.start
end = part.end
sequence = sub(sequence,start,end)
else:
continue
final_seq = sequence.replace("M","")
print(len(final_seq))
fw = open("2.txt","w")
fw.write(">%s\n%s\n"%("intergene",final_seq))
fw.close()
更新20181201
(我的天哪!已经12月份啦!)
提取多个物种叶绿体基因组共有的CDS序列
使用genbank格式的叶绿体基因组注释文件,放到同一个文件夹内
import os
from Bio import SeqIO
cp_genbank = os.listdir("./")
gene_name_set = []
for fileName in cp_genbank:
Coding_sequence = {}
for rec in SeqIO.parse(fileName,"gb"):
for feature in rec.features:
if feature.type == "CDS":
Coding_sequence[feature.qualifiers["gene"][0]] = str(feature.extract(rec.seq))
gene_name_set.append(list(Coding_sequence.keys()))
a = set(gene_name_set[0]).intersection(set(gene_name_set(1)))
for i in range(2,len(gene_name_set)):
a = a.intersection(set(gene_name_set[i]))
geneName = list(a)
fw = open("Coding_region.fasta","w")
for fileName in cp_genbank:
fw.write(">"+fileName+"\n")
Coding_sequence = {}
for rec in SeqIO.parse(fileName,"gb"):
for feature in rec.features:
if feature.type == "CDS":
Coding_sequence[feature.qualifiers["gene"][0]] = str(feature.extract())
for gene_name in geneName:
fw.write(str(Coding_sequence[gene_name])+"\n")
fw.close()
更新20190108
- 单行fasta改成多行
使用Biopython的SeqIO模块将fasta写入到文件中的时候序列是放在一行,如果很长的话(比如叶绿体基因组通常在150kbp左右)看着好像不太舒服,再长一点打开文本都有点困难,还是准换成多行比较好一点(每行60个),记录简单的python脚本
from Bio import SeqIO
i = 0
with open("output_filename.fasta","w") as fw:
for rec in SeqIO.parse("input.fasta","fasta"):
fw.write(">%s\n"%(rec.id))
while i < len(rec.seq):
fw.write("%s\n"%(str(rec.seq[i:i+60])))
i = i + 60
更新
好友问fasta文件中大于号所在的行,自己的理解是
>Seq1
AGCTAGCT
>Seq2
TGCATGCA
>Seq3
CGATCGAT
变成
AGCTAGCT
TGCATGCA
CGATCGAT
python脚本
fr = open("input_file.fasta","r") #读入需要处理的fasta文件
fw = open("output_file.txt","w") #新建文本文件,把结果写到这个文件里
for line in fr:
if line[0] == ">":
continue
else:
fw.write(line)
fr.close()
fw.close() #关闭文件
python读入文件时是一行一行读进来的,所以加一个简单的判断,如果这一行第一个字符是“>”就跳过,如果不是就将这一行写入新文件里;这里需要注意的是python的第一个字符是从零开始的。