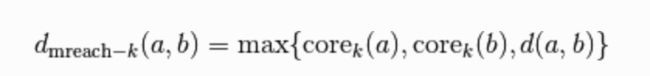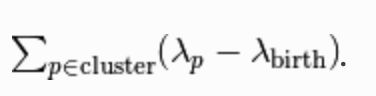聚类方法之 HDBSCAN —— 层次DBSCAN 的原理分析
HDBSCAN
HDBSCAN(Hierarchical Density-Based Spatial Clustering of Applications with Noise)是由Campello,Moulavi和Sander开发的聚类算法。 它通过将DBSCAN转换为分层聚类算法来扩展DBSCAN,然后基于聚类稳定性,使用了提取平面聚类地技术。
和传统DBSCAN最大的不同之处在于,HDBSCAN可以处理密度不同的聚类问题。
本文和HDBSCAN的论文不同,将不从DBSCAN出发。相反的,本文将从该算法如何与Robust Single Linkage(鲁棒单链接算法)紧密联系出发,并在其上面进行平面群集提取。
在我们开始之前,我们将加载我们需要的库,以及设置我们的可视化代码。
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import sklearn.datasets as data
%matplotlib inline
sns.set_context('poster')
sns.set_style('white')
sns.set_color_codes()
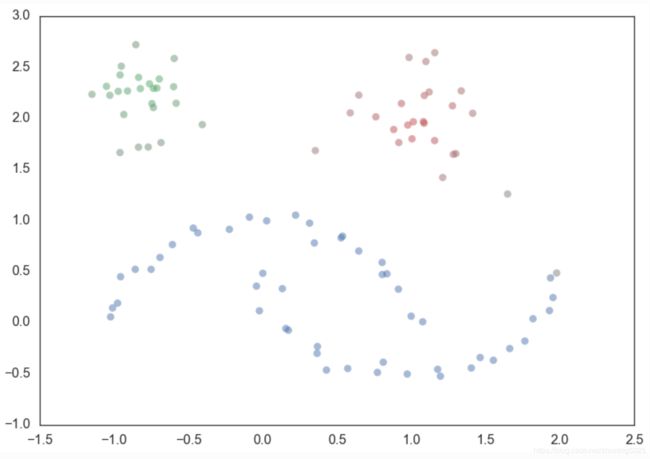
plot_kwds = {'alpha' : 0.5, 's' : 80, 'linewidths':0}接下来我们需要的是一些数据。 为了说明一个例子,我们需要数据量相当小,以便我们可以看到发生了什么。 sklearn具有生成样本聚类数据的工具,因此我将利用它并创建一百个数据点的数据集。
moons, _ = data.make_moons(n_samples=50, noise=0.05)
blobs, _ = data.make_blobs(n_samples=50, centers=[(-0.75,2.25), (1.0, 2.0)], cluster_std=0.25)
test_data = np.vstack([moons, blobs])
plt.scatter(test_data.T[0], test_data.T[1], color='b', **plot_kwds)现在,解释HDBSCAN的最佳方法就是实现它,刚开始不要因为作为一个调包侠。 所以让我们加载hdbscan库并开始工作。
import hdbscan
clusterer = hdbscan.HDBSCAN(min_cluster_size=5, gen_min_span_tree=True)
clusterer.fit(test_data)
HDBSCAN(algorithm='best', alpha=1.0, approx_min_span_tree=True,
gen_min_span_tree=True, leaf_size=40, memory=Memory(cachedir=None),
metric='euclidean', min_cluster_size=5, min_samples=None, p=None)那么现在我们已经聚集了数据 ,但算法背后究竟发生了什么? 我们可以将其分解为一系列步骤:
- 根据密度/稀疏度变换空间(Transform the space)
- 构建距离加权图的最小生成树(Build the minimum spanning tree)
- 构建集群层次结构(Build the cluster hierarchy)
- 根据最小簇大小压缩集群层次结构(Condense the cluster tree)
- 从压缩树中提取稳定群集(Extract the clusters)
根据密度/稀疏度变换空间(Transform the space)
为了找到簇,就如我们希望在稀疏的噪声海洋中找到高密度的岛屿 - 并且噪声的假设很重要。因为真实数据是混乱的,它可能具有异常值,数据损坏和噪声。
聚类算法的核心是单链路聚类(single linkage clustering),它可能对噪声非常敏感:一个噪声数据点如果正好在两个簇之间,那么它可能就可以充当岛屿之间的桥梁,将它们粘合在一起(导致错误的聚类)。 我们希望我们的算法对噪声具有鲁棒性,因此我们需要找到一种方法来实现“降低海平面”,然后再运行单链路聚类算法。
但我们应该如何在不进行聚类的情况下描述“海洋”和“土地”的特征?只要我们能够估算出密度,我们就可以将低密度点视为“海洋”。 这里的目标不是完全准确地区分“海洋”和“陆地” ,因为这是聚类的第一步,而不是输出 。只是为了使我们的聚类核心算法对噪声更加鲁棒。因此,为了“海洋”的识别,我们希望降低海平面,更具体地来说,这意味着使“海洋”点与彼此和“土地”之间的距离更远。
然而,这只是直觉分析。 那么它在实践中如何实现? 我们需要一个计算量很小的密度估计方法,最简单的是KNN( kth nearest neighbor)。 如果我们有数据的距离矩阵(无论如何我们将迫切需要),我们可以简单地读取它。 让我们将其形式化并(遵循DBSCAN,LOF和HDBSCAN文献)定义核心距离:点 x (参数 k )的核心距离 。
现在我们需要一种方法来分散低密度点(具有高核心距离)。 这样做的简单方法是在点之间定义一个新的距离来度量。我们将调用(再次遵循文献)相互可达度量距离(mutual reachability distance)。 我们定义相互可达度量距离如下:
其中d(a, b) 是a和b之间的距离。 在这个度量下,密集点(具有低核心距离)保持彼此相同的距离,但是稀疏点被推开至少远离任何其他点的核心距离。 这有效地“降低了海平面”,及散布了稀疏的“海洋”点,同时保持“土地”不受影响。
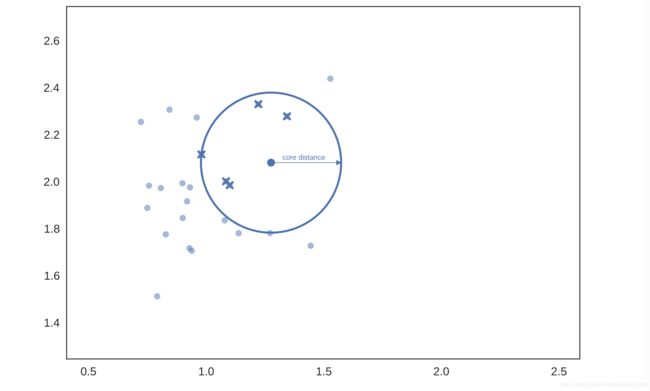
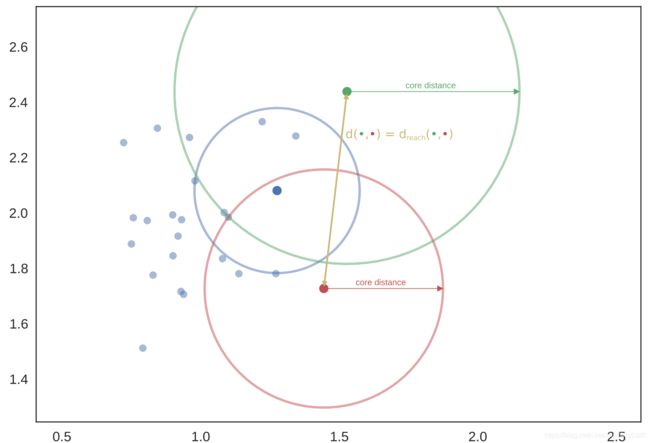
这里需要注意的是,这效果显然取决于k的选择; 较大的k值将更多的点归在“海洋”中。 从图片中可以更好的解释上述内容,所以让我们设k的值为5。 然后,对于给定的点,我们可以根据核心距离来绘制一个圆圈,圆圈接触到第六个(包括计算点本身)最邻近的点,如下所示:
选择另一个点,做同样的计算,但这次是一组不同的邻居(其中一个是我们挑选的第一个点)。
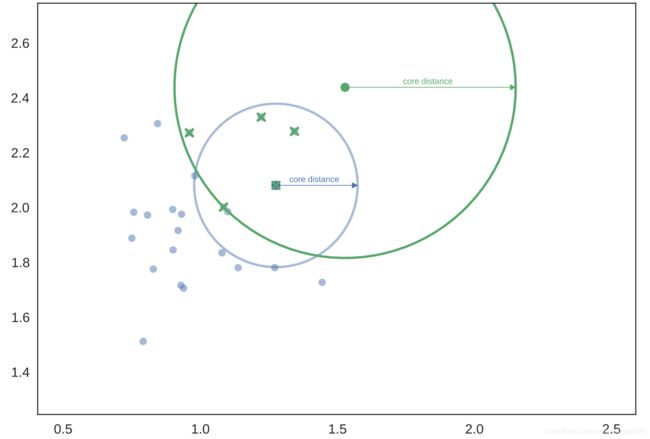
为了更好的测量,我们运用另一组六个最近邻和另一个半径略有不同的圆。
现在,如果我们想知道蓝点和绿点之间的相互可达度量距离,我们可以通过从绘制绿色和蓝色之间的距离来开始:
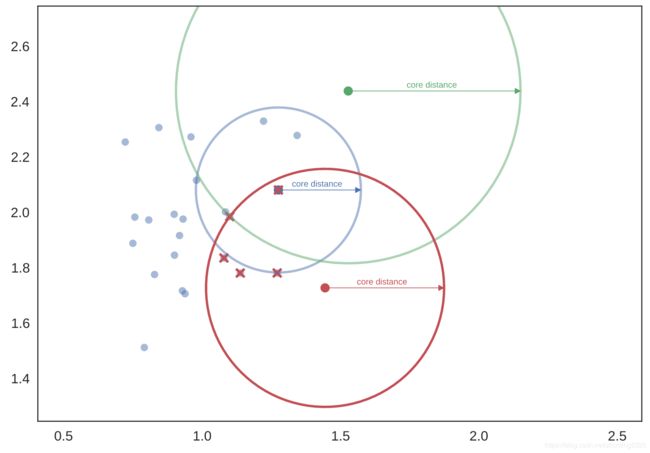
穿过蓝色圆圈,但不穿过绿色圆圈 - 绿色的核心距离大于蓝色和绿色之间的距离。 因此,我们需要将蓝色和绿色之间的相互可达度量距离标记为更大的 - 等于绿色圆圈的半径。
另一方面,从红色到绿色的相互可达度量距离简单地是从红色点到绿色点的距离,因为该距离大于任一核心距离(即距离箭头穿过两个圆圈)。
通常,有一个证明,表明以相互可达度量距离作为一种变换方式,可以让单链路聚类更加贴合地去拟合我们的样本层次分布,无论我们的样本的真实密度是什么样的。
构建最小生成树(Build the minimum spanning tree)
至此,我们在数据集上已有了一个新的相互可达度量距离,然后我们希望可以在密集的数据上找到“岛屿”。当然,密集的区域也是相对的,不同的岛屿可能有不同的密度。从概念上来说,我们将要做的是:
将数据视为加权图,数据点作为顶点,而任意两个顶点之间边的权重,等于两点之间的相互可达度量距离。
现在我们开始考虑一个阈值,从一个较高的值开始,然后逐步降低。我们丢弃加权图中权重超过这个阈值的所有边。在我们丢弃边缘的同时,我们将断开的图连接到已有的分组。最终,我们将在不用的阈值水平上获得一个层次的结构(从完全连接到完全断开连接)。
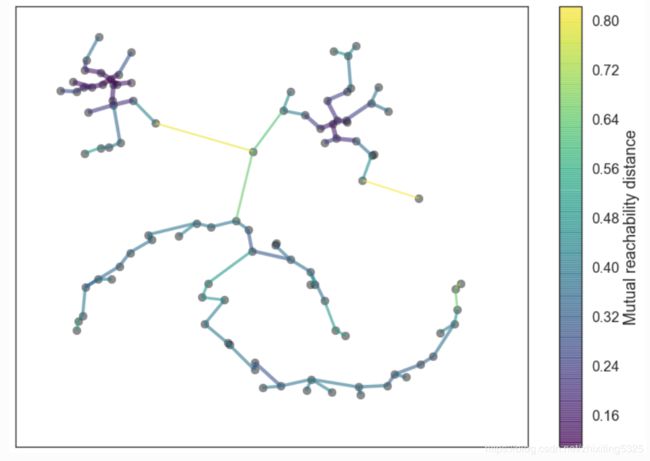
然而在实践过程中,这个操作是非常昂贵的,因为有n^2条边,我们并不希望算法的时间复杂度到这个级别。所以我们引入了最小生成树,因为只要断开最小生成树中的一条边,就可以获得一个完全分离的组。
我们可以通过Prim算法非常有效地生成一个最小生成树。你可以看到下面HDBSCAN的最小生成树,需要注意的是:该最小生成树的权重是两点间的相互可达度量距离。在这个案例中,我们令k=5。
当数据存储于度量空间时,我们可以运用更快的方法,例如 Dual Tree Boruvka 来构建最小生成树。
clusterer.minimum_spanning_tree_.plot(edge_cmap='viridis',
edge_alpha=0.6,
node_size=80,
edge_linewidth=2)构建集群层次结构(Build the cluster hierarchy)
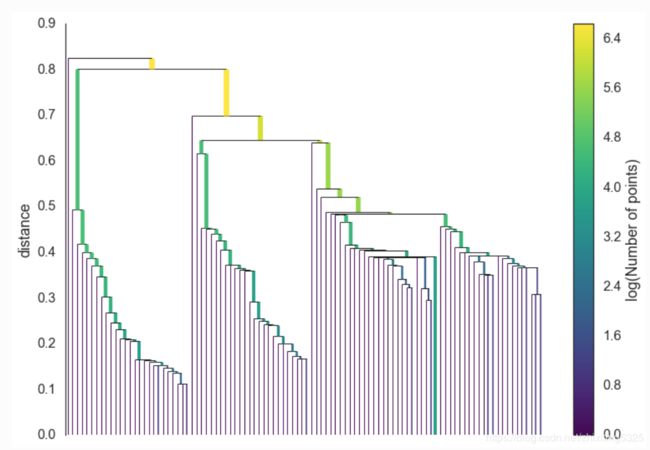
给定最小生成树,下一步是将其转换为连接组件的层次结构。 这最容易以相反的顺序完成:按距离(按递增顺序)对树的边进行排序,然后迭代,为每个边创建一个新的合并集群。 这里唯一有困难的部分是,对每一条边,如何选择是否将该边连接在的两个簇聚在一起。但这很容易通过union-find的数据结构来实现。 我们可以将结果绘制成树形图,如下所示:
clusterer.single_linkage_tree_.plot(cmap='viridis', colorbar=True)这个方法可以让我们得到最终点,也就是单链接的最终点。但我们想要的更多,虽然集群的层次结构很好,但是我们需要的其实是一组平面聚群。我们可以在上图中会议一条水平线,并选择它所切割的簇来实现这一点。但实际上,这也就是DBSCAN做的事情,但问题是,我们如何知道这条线在哪绘制? DBSCAN只是将其作为一个参数(非常不直观)。 更糟糕的是,我们真的想要处理变密度簇,任意选择的切割线都是一个固定的密度水平。 理想情况下,我们希望能够在不同的地方切割树以选择我们的聚类。 这就是HDBSCAN的下一步开始并与强大的单一链接产生差异的地方。
压缩聚类树(Condense the cluster tree)
类群提取的第一步是将大而复杂的类群层次结构缩小为一个较小的树。正如你在上面的层次结构中所看到的那样,通常情况下,群集拆分是从群集中分离出来的一个或两个点; 这是关键点 - 我们不希望将其视为一个分裂为两个新集群的集群,而是将其视为一个“丢失点”的持久集群(这句话博主不是非常的理解,如果你有更好的见解请留言讨论)。为了使这个具体化,我们需要引入一个最小簇大小的概念,我们将其作为HDBSCAN的参数。
一旦我们得到最小簇大小的值,我们就可以遍历层次结构,并在每次拆分时判断:由拆分创建的新簇之一是否具有比最小簇大小更少的点。如果我们的点数少于最小簇大小,我们将其声明为“点落在类群之外”并让较大的群集保留父节点的群集标识,从而标记出哪些点落在了群集之外,以及这种情况发生的时候 的距离值。
另一方面,如果拆分为两个集群(每个集群至少与最小集群大小一样大),然后我们视其为真正的集群拆分,并让该拆分在树中持续存在。在遍历整个层次结构并执行此操作后,我们最终得到一个小得多的树,其中包含更少的节点,每个节点都有关于该节点上集群大小如何随着距离变化的数据。我们可以将其可视化出来,类似于上面的树状图 - 我们可以再次使用线的宽度表示聚类中的点数。然而,这一次,这个宽度会随着离群点而变化。对于使用最小簇大小为5的数据,结果如下所示:
clusterer.condensed_tree_.plot()这更易于查看和处理,尤其是与我们当前的测试数据集一样简单的聚类问题。 但是,我们仍然需要选择要用作平面聚类的群集。通过前述的内容,应该会给你一些关于如何做到这一点的思路。
提取簇(Extract the clusters)
直观地说,我们希望选择群集应该具有持续存在并具有更长生命周期的特征; 短寿命集群可能仅仅是单链接方法的工件。 看看之前的图片,我们可以认为我们想要选择那些在图中具有最大颜色面积的类群。 如果要进行平面聚类,我们需要添加进一步的要求,如果你选择一个类群,则无法再选择所有该集群的后代。 事实上,这个直观概念正是HDBSCAN所做的。 当然,我们需要将这个理念具象化成为一个具体的算法。
首先,我们需要一个与距离不同的度量来考虑簇的持续性; 在这里我们将用距离的倒数来度量,即![]() 。
。 ![]() 对于给定的集群,我们可以将值
对于给定的集群,我们可以将值![]() 定义为当集群拆分成功,并成为它自己的子集群时的
定义为当集群拆分成功,并成为它自己的子集群时的![]() 值;将
值;将![]() 定义为当群集被分成较小的群集时的
定义为当群集被分成较小的群集时的![]() 值。 反过来,对于给定的集群,对于该集群中的每个点p,我们可以将值
值。 反过来,对于给定的集群,对于该集群中的每个点p,我们可以将值 ![]() 定义为该点 '被从该集群中剔除' 的
定义为该点 '被从该集群中剔除' 的![]() 值。现在,对于每个集群,我们令它的稳定性为
值。现在,对于每个集群,我们令它的稳定性为
声明所有叶节点都是选定的簇。 现在通过树进行遍历(反向拓扑排序顺序)。 如果子集群的稳定性之和大于父集群的稳定性,那么我们将集群稳定性设置为子稳定性的总和。 另一方面,如果群集的稳定性大于其子节点的总和,那么我们将群集声明为选定群集并归并其所有后代。 一旦我们到达根节点,我们将当前选定的集群称为平面聚类并返回该节点。
上述的过程可能是冗长而复杂的,但它实际上只是执行我们的 “选择具有最大总墨水面积的图中的聚类” 并受到后代约束(我们之前所提及的)的过程。 我们通过该算法可以在压缩树的树形图中选择簇,运行下面的代码你可以得到你期望的结果:
clusterer.condensed_tree_.plot(select_clusters=True, selection_palette=sns.color_palette())现在我们已经拥有了类群,运用 sklearn 的 API将其转换为集群标签就可以了。 不在选定聚类中的任何一个点都被视为是一个噪点(并指定标签-1)。 我们可以做更多的事情:对于每个集群,我们为该集群中的每个点p都有![]() ; 如果我们简单地将这些值标准化(因此它们的范围从0到1),那么我们可以衡量集群中每个点的是该集群成员资格的强度。 hdbscan库将其作为clusterer对象的probabilities_属性返回。 因此,有了标签和会员优势,我们可以制作标准图,根据聚类标签选择点的颜色,并根据成员的强度去饱和颜色(并使非聚集点纯灰色)。
; 如果我们简单地将这些值标准化(因此它们的范围从0到1),那么我们可以衡量集群中每个点的是该集群成员资格的强度。 hdbscan库将其作为clusterer对象的probabilities_属性返回。 因此,有了标签和会员优势,我们可以制作标准图,根据聚类标签选择点的颜色,并根据成员的强度去饱和颜色(并使非聚集点纯灰色)。
palette = sns.color_palette()
cluster_colors = [sns.desaturate(palette[col], sat)
if col >= 0 else (0.5, 0.5, 0.5) for col, sat in
zip(clusterer.labels_, clusterer.probabilities_)]
plt.scatter(test_data.T[0], test_data.T[1], c=cluster_colors, **plot_kwds)这就是HDBSCAN的工作方式。 这可能看起来有点复杂 - 算法中有相当多的可抑制部分 - 但最终每个部分实际上非常简单并且可以很好地进行优化。
博主附:
在去天津比赛的火车上把这篇写完了,感觉对这个算法有了更深刻的认识,其实这种算法也是揉合了很多工程性的方法,然后达到了很好的效果。
文中还是有一些地方博主有一些疑惑,希望大家有见解的话多多留言交流。
Reference:
How HDBSCAN Works