简单的SIR模型传播--numpy/matplotlib(某师范大学学生的期中大作业)
SIR模型是传染病模型中最经典的模型,其中S表示易感者,I表示感染者,R表示移出者。模型中把传染病流行范围内的人群分成三类:S类,易感者(Susceptible),指未得病者,但缺乏免疫能力,与感病者接触后容易受到感染;I类,感病者(Infective),指染上传染病的人,它可以传播给S类成员;R类,移出者(Removal),指被隔离,或因病愈而具有免疫力的人。其中S被附近节点感染的概率为beta,如果S状态节点有m个I状态的话,S在下个时间段被感染的概率就是1-(1-beta)^m,如果本身就是I节点,那么下个阶段康复的概率为 u。
然后我们可以用下面代码创建ER图(邻接矩阵)
def CreateER(N, p):
mat = np.random.rand(N, N)
mat = np.where(mat>p, 0, 1)
for i in range(N):
mat[i, i] = 0
mat[i, :] = mat[:, i]
return mat
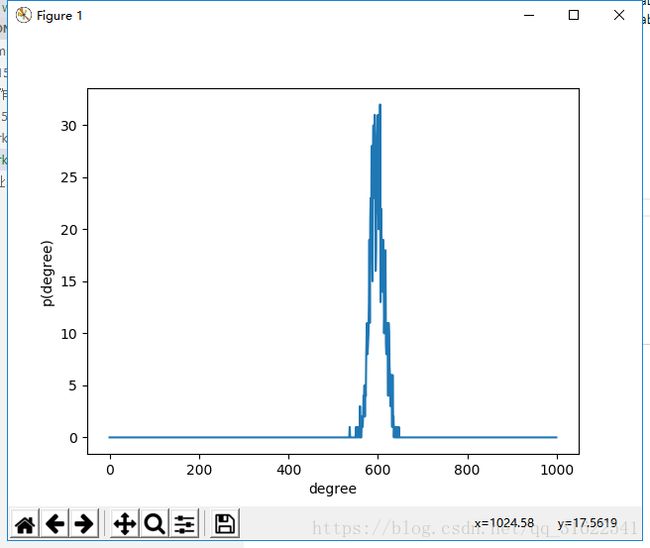
接着我们可以通过计算直方图来进行区间统计,代码如下:
def Distribution(mat):
(a, b) = mat.shape
Count = np.array([mat[i, :].sum() for i in range(a)])
hist = np.histogram(Count, bins=1000, range=(0,1000))
plt.plot(hist[0])
plt.xlabel('degree')
plt.ylabel('p(degree)')
plt.show()
return hist最终生成相应的SIR模型
# 对一个mat进行一次SIR的传播 S 1 -- I 2 -- R 3 普通人--1 感染者--2 恢复者
def SIRSpread(mat, beta, mu, vec):
nvec = np.array(vec)
for i in range(vec.size):
if vec[i] == 1:
num = 0
for j in range(vec.size):
if mat[i,j] == 1 and vec[j] == 2:
num = num + 1
prob = 1 - (1-beta)**num
rand = random.random()
if rand < prob:
nvec[i] = 2
elif vec[i] == 2:
rand = random.random()
if rand < mu:
nvec[i] = 3
return nvec
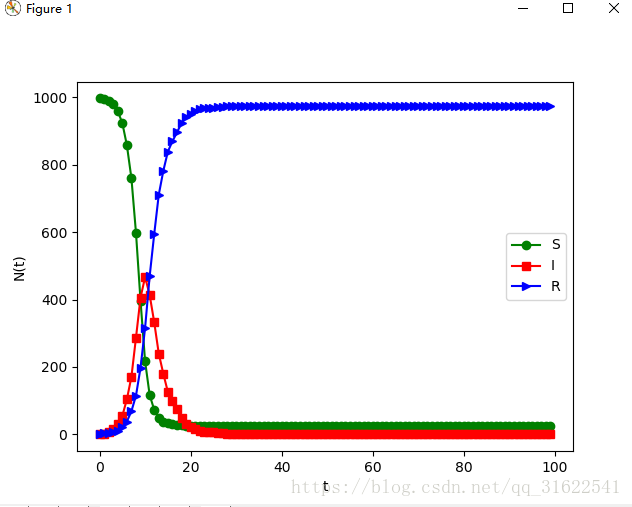
# 设置传播次数,来进行传播,并返回每个阶段S,I,R的数量
def MultiSpread(N, beta, mu, t):
mat = CreateER(N, 0.01)
vec = np.array([1 for i in range(N)])
rNum = random.randint(0, N-1)
vec[rNum] = 2
S = []
I = []
R = []
for i in range(t):
vec = SIRSpread(mat, beta, mu, vec)
S.append(np.where(np.array(vec)==1, 1, 0).sum())
I.append(np.where(np.array(vec)==2, 1, 0).sum())
R.append(np.where(np.array(vec)==3, 1, 0).sum())
return S,I,R实验结果如图所示