遗传算法(Genetic Algorithm)原理详解和matlab代码解析实现及对应gaot工具箱实现代码
原理-算例-代码
1.定义
遗传算法(Genetic Algorithm, GA)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法。
主要特点
- 直接对结构对象进行操作,不存在求导和函数连续性的限定;
- 具有内在的隐并行性和更好的全局寻优能力;
- 采用概率化的寻优方法,不需要确定的规则就能自动获取和指导优化的搜索空间,自适应地调整搜索方向。
对象:一种群体中的所有个体
基本操作: 选择、交叉和变异
核心内容:
- 参数编码
- 初始群体的设定
- 适应度函数的设计
- 遗传操作设计
- 控制参数设定
2 常用词汇:
基因型(genotype):
性状染色体的内部表现。
编码样式,把自变量转换为如二进制数串。
方法有:二进制编码法、浮点编码法、符号编码法。
表现型:
染色体决定的性状的外部表现
自变量取值。
编码(coding):
DNA中遗传信息在一个长链上按一定的模式排列。
遗传编码可看作从表现型到基因型的映射。
解码(decoding):
基因型到表现型的映射。
个体(individual):
指染色体带有特征的实体;
就是一个基因型的样本
种群(population):
个体的集合,该集合内个体数称为种群
样本集合
适应度(fitness):
度量某个物种对于生存环境的适应程度。
评价指标函数
3 形象理解
既然我们把函数曲线理解成一个一个山峰和山谷组成的山脉。那么我们可以设想所得到的每一个解就是一只袋鼠,我们希望它们不断的向着更高处跳去,直到跳到最高的山峰。所以求最大值的过程就转化成一个“袋鼠跳”的过程。
下面介绍介绍“袋鼠跳”的几种方式。
- 爬山算法:一只袋鼠朝着比现在高的地方跳去。它找到了不远处的最高的山峰。但是这座山不一定是最高峰。这就是爬山算法,它不能保证局部最优值就是全局最优值。
- 模拟退火:袋鼠喝醉了。它随机地跳了很长时间。这期间,它可能走向高处,也可能踏入平地。但是,它渐渐清醒了并朝最高峰跳去。这就是模拟退火算法。
- 遗传算法:有很多袋鼠,它们降落到喜玛拉雅山脉的任意地方。这些袋鼠并不知道它们的任务是寻找珠穆朗玛峰。但每过几年,就在一些海拔高度较低的地方射杀一些袋鼠。于是,不断有袋鼠死于海拔较低的地方,而越是在海拔高的袋鼠越是能活得更久,也越有机会生儿育女。就这样经过许多年,这些袋鼠们竟然都不自觉地聚拢到了一个个的山峰上,可是在所有的袋鼠中,只有聚拢到珠穆朗玛峰的袋鼠被带回了美丽的澳洲。
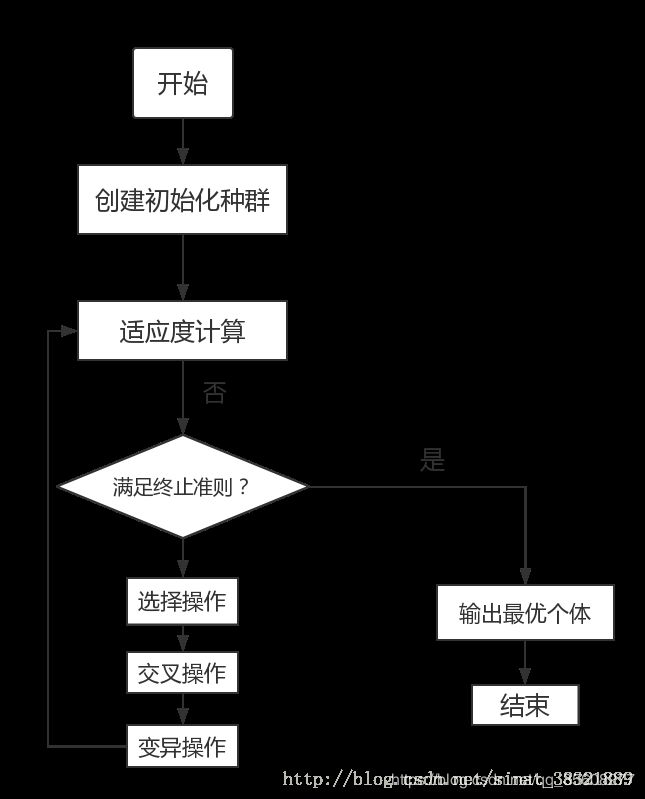
遗传算法的一般步骤:
- 随机产生种群。
- 根据策略判断个体的适应度,是否符合优化准则,若符合,输出最佳个体及其最优解,结束。否则,进行下一步。
- 依据适应度选择父母,适应度高的个体被选中的概率高,适应度低的个体被淘汰。
- 用父母的染色体按照一定的方法进行交叉,生成子代。
- 对子代染色体进行变异。
- 由交叉和变异产生新一代种群,返回步骤2,直到最优解产生。
具体二进制编码方式、轮盘选择、单点交叉、基本位变异是最为简单好理解的。参考
https://blog.csdn.net/u012422446/article/details/68061932
https://www.jianshu.com/p/ae5157c26af9
在看代码实现前,先看这个例子,会手动计算后更易理解。
算法例子
https://blog.csdn.net/u012422446/article/details/68061932
matlab代码实现
下面就用代码实现袋鼠如何爬上这座山的最高峰
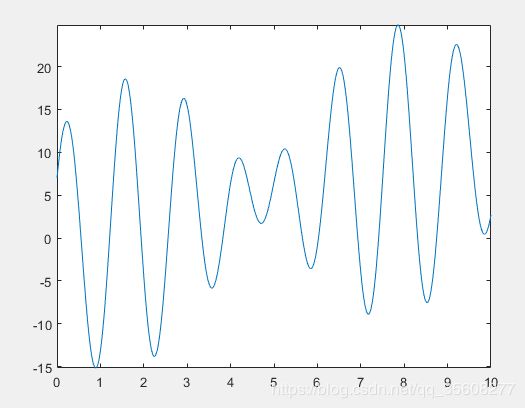
% 即求下列函数的最大值 %
% f(x)=x+10*sin(5x)+7*cos(4x) x∈[0,10] %
涉及了一个染色体长度的计算:
就是在自变量区间,多长的染色体满足精度要求。
将 x 的值用一个10位的二值形式表示为二值问题,一个10位的二值数提供的分辨率是每位 (10-0)/(2^10-1)≈0.01
将变量域 [0,10] 离散化为二值域 [0,1023], x=0+10*b/1023, 其中 b 是 [0,1023] 中的一个二值数。
按照算法流程图进行:
初始化-进入循环(计算目标函数值-计算适应度-选择-交叉-变异)-到达迭代次数-输出循环过程中达到的最优值。
每个函数保存为一个m文件
0 主函数
% 下面举例说明遗传算法 %
% 求下列函数的最大值 %
% f(x)=x+10*sin(5x)+7*cos(4x) x∈[0,10] %
% 将 x 的值用一个10位的二值形式表示为二值问题,一个10位的二值数提供的分辨率是每为 (10-0)/(2^10-1)≈0.01
% 将变量域 [0,10] 离散化为二值域 [0,1023], x=0+10*b/1023, 其中 b 是 [0,1023] 中的一个二值数。 %
% %
%--------------------------------------------------------------------------------------------------------------%
%--------------------------------------------------------------------------------------------------------------
% 2.0 主程序
%遗传算法主程序
%Name:genmain05.m
%要求精度不大于0.01,
clear
clc
popsize=20; %群体大小
chromlength=10; %字符串长度(个体长度)
pc=0.6; %交叉概率,只有在随机数小于pc时,才会产生交叉
pm=0.001; %变异概率
pop=initpop(popsize,chromlength); %随机产生初始群体
for i=1:2000 %20为遗传代数
[objvalue]=calobjvalue(pop); %计算目标函数
fitvalue=calfitvalue(objvalue); %计算群体中每个个体的适应度
[newpop]=selection(pop,fitvalue); %复制
[newpop1]=crossover(newpop,pc); %交叉
[newpop2]=mutation(newpop1,pm); %变异
[objvalue]=calobjvalue(newpop2); %计算目标函数
fitvalue=calfitvalue(objvalue); %计算群体中每个个体的适应度
[bestindividual,bestfit]=best(newpop2,fitvalue); %求出群体中适应值最大的个体及其适应值
y(i)=bestfit; %返回的 y 是自适应度值,而非函数值
x(i)=decodechrom(bestindividual,1,chromlength)*10/1023; %将自变量解码成十进制
pop=newpop2;
end
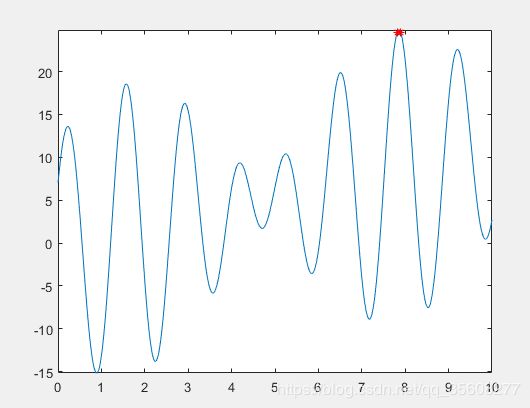
fplot('x+10*sin(5*x)+7*cos(4*x)',[0 10])
hold on
plot(x,y,'r*')
hold on
[z ,index]=max(y); %计算最大值及其位置
x5=x(index) %计算最大值对应的x值
ymax=z
1. 初始化
主函数中调用,生成20个个体,按照精度要求选择染色体的长度
initpop.m
% 2.1初始化(编码)
% initpop.m函数的功能是实现群体的初始化,popsize表示群体的大小,chromlength表示染色体的长度(二值数的长度),
% 长度大小取决于变量的二进制编码的长度(在本例中取10位)。
%初始化
function pop=initpop(popsize,chromlength)
pop=round(rand(popsize,chromlength));
% rand随机产生每个单元为 {0,1} 行数为popsize,列数为chromlength的矩阵,
% round对矩阵的每个单元进行圆整四舍五入。这样产生的初始种群。
2 计算目标函数值
calobjvalue.m
% 2.2.3 计算目标函数值
% calobjvalue.m函数的功能是实现目标函数的计算,其公式采用本文示例仿真,可根据不同优化问题予以修改。
%遗传算法子程序
%Name: calobjvalue.m
%实现目标函数的计算,将 二值域 中的数转化为 变量域的数
function [objvalue]=calobjvalue(pop)
temp1=decodechrom(pop,1,10); %将pop每行转化成十进制数
x=temp1*10/1023; %在精度不大于0.01时,最小整数为1023,即需要10位二进制
objvalue=x+10*sin(5*x)+7*cos(4*x); %计算目标函数值
decodechrom.m
(主要针对多个变量时候的一条染色体拆分,此处的例子不用拆分,pop1=pop)
% 2.2.2 将二进制编码转化为十进制数(2)
% decodechrom.m函数的功能是将染色体(或二进制编码)转换为十进制,参数spoint表示待解码的二进制串的起始位置
% (对于多个变量而言,如有两个变量,采用20为表示,每个变量10位,则第一个变量从1开始,另一个变量从11开始。本例为1),
% 参数1ength表示所截取的长度(本例为10)。
%遗传算法子程序
%Name: decodechrom.m
%将二进制编码转换成十进制
function pop2=decodechrom(pop,spoint,length) %1 10
pop1=pop(:,spoint:spoint+length-1);
pop2=decodebinary(pop1); %将pop每行转换成十进制值,结果是20*1的矩阵
decodebinary.m
%产生 [2^n 2^(n-1) ... 1] 的行向量,然后求和,将二进制转化为十进制
function pop2=decodebinary(pop)
[px,py]=size(pop); %求pop行和列数
for i=1:py
pop1(:,i)=2.^(py-i).*pop(:,i);
end
pop2=sum(pop1,2); %求pop1的每行之和
3 计算适应度函数
calfitvalue.m
为了计算后续概率,把小于0的目标函数值设为0
% 2.3 计算个体的适应值
%遗传算法子程序
%Name:calfitvalue.m
%计算个体的适应值
function fitvalue=calfitvalue(objvalue)
[px,py]=size(objvalue); %目标值有正有负
for i=1:px
if objvalue(i)>0
temp=objvalue(i);
else
temp=0.0;
end
fitvalue(i)=temp;
end
fitvalue=fitvalue';
4 选择
轮盘法选择。
原理如下
下面以轮盘赌选择为例给大家讲解一下:
假如有5条染色体,他们的适应度分别为5、8、3、7、2。
那么总的适应度为:F = 5 + 8 + 3 + 7 + 2 = 25。
那么各个个体的被选中的概率为:
α1 = ( 5 / 25 ) * 100% = 20%=0.2
α2 = ( 8 / 25 ) * 100% = 32%=0.32
α3 = ( 3 / 25 ) * 100% = 12%=0.12
α4 = ( 7 / 25 ) * 100% = 28%=0.28
α5 = ( 2 / 25 ) * 100% = 8%=0.08
怎么用代码实现呢?
思想:
把α1-α5,按比例顺序,放到长度为1的一维的线上,开始随机数生成。
理解成向线上随机撒一个点。撒到哪一段就是选择了哪个个体。
eg:
点到0.25时候,处在α1之后,在α2段,所以α2个体被选择一次。
再随机生成0.55,在α3段,所以α3个体被选择一次。
为了便于排序,先把点生成好,按顺序排列,从小到大的进行比较。
即如下代码:
selection.m
function [newpop]=selection(pop,fitvalue)
totalfit=sum(fitvalue); %求适应值之和
fitvalue=fitvalue/totalfit; %单个个体被选择的概率
fitvalue=cumsum(fitvalue); %如 fitvalue=[1 2 3 4],则 cumsum(fitvalue)=[1 3 6 10],不明白为什么要累加
[px,py]=size(pop); %20*10
ms=sort(rand(px,1)); %从小到大排列
fitin=1;
newin=1;
while newin<=px %选出20个新个体,有重复情况,和上面介绍的方法不太一样
if(ms(newin))5 交叉
单点交叉,以0.6的概率对相邻的两个个体染色体随机选择交叉,并随机选择交叉的位置。
crossover.m
% 2.5 交叉
% 交叉(crossover),群体中的每个个体之间都以一定的概率 pc 交叉,即两个个体从各自字符串的某一位置
% (一般是随机确定)开始互相交换,这类似生物进化过程中的基因分裂与重组。例如,假设2个父代个体x1,x2为:
% x1=0100110
% x2=1010001
% 从每个个体的第3位开始交叉,交又后得到2个新的子代个体y1,y2分别为:
% y1=0100001
% y2=1010110
% 这样2个子代个体就分别具有了2个父代个体的某些特征。利用交又我们有可能由父代个体在子代组合成具有更高适合度的个体。
% 事实上交叉是遗传算法区别于其它传统优化方法的主要特点之一。
%遗传算法子程序
%Name: crossover.m
%交叉
function [newpop]=crossover(pop,pc) %pc=0.6
[px,py]=size(pop);
newpop=ones(size(pop));
for i=1:2:px-1 %步长为2,是将相邻的两个个体进行交叉
if(rand6 变异
mutation.m
% 2.6 变异
% 变异(mutation),基因的突变普遍存在于生物的进化过程中。变异是指父代中的每个个体的每一位都以概率 pm 翻转,
%即由“1”变为“0”,或由“0”变为“1”。
%遗传算法的变异特性可以使求解过程随机地搜索到解可能存在的整个空间,因此可以 在一定程度上 求得全局最优解。
%遗传算法子程序
%Name: mutation.m
%变异
function [newpop]=mutation(pop,pm)
[px,py]=size(pop);
newpop=ones(size(pop));
for i=1:px
if(rand7 求出每一代 中最大得适应值及其个体
best.m
% 2.7 求出群体中最大得适应值及其个体
%遗传算法子程序
%Name: best.m
%求出第 t 代群体中适应值最大的值
function [bestindividual,bestfit]=best(pop,fitvalue)
[px,py]=size(pop);
bestindividual=pop(1,:);
bestfit=fitvalue(1);
for i=2:px
if fitvalue(i)>bestfit
bestindividual=pop(i,:);
bestfit=fitvalue(i);
end
end
gaot 工具箱实现遗传算法
注:gaot工具箱要加入路径
main.m
%% I. 清空环境变量
%optimtool solver 中选择 GA
%添加 gaot工具箱
clear all
clc
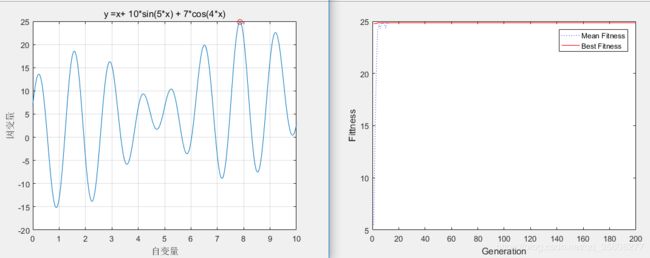
%% II. 绘制函数曲线
x = 0:0.01:10;
y =x+ 10*sin(5*x)+7*cos(4*x);
figure
plot(x, y)
xlabel('自变量')
ylabel('因变量')
title('y =x+ 10*sin(5*x) + 7*cos(4*x)')
grid
%% III. 初始化种群
initPop = initializega(50,[0 10],'fitness'); %种群大小;变量变化范围;适应度函数的名称
%看一下initpop 第二列代表 适应度函数值
%% IV. 遗传算法优化
[x endPop bpop trace] = ga([0 10],'fitness',[],initPop,[1e-6 1 1],'maxGenTerm',200,...
'normGeomSelect',0.08,'arithXover',2,'nonUnifMutation',[2 25 3]);
%变量范围上下界;适应度函数;适应度函数的参数;初始种群;精度和显示方式;终止函数的名称;
%终止函数的参数;选择函数的名称;选择函数的参数;交叉函数的名称;交叉函数的参数;变异函数的名称;变异函数的参数
% X 最优个体 endpop 优化终止的最优种群 bpop 最优种群的进化轨迹 trace 进化迭代过程中
%最优的适应度函数值和适应度函数值矩阵
%% V. 输出最优解并绘制最优点
x
hold on
plot (endPop(:,1),endPop(:,2),'ro')
%% VI. 绘制迭代进化曲线
figure(2)
plot(trace(:,1),trace(:,3),'b:')
hold on
plot(trace(:,1),trace(:,2),'r-')
xlabel('Generation'); ylabel('Fittness');
legend('Mean Fitness', 'Best Fitness')
fitness.m
function [sol, fitnessVal] = fitness(sol, options)
x = sol(1);
fitnessVal = x+10*sin(5*x)+7*cos(4*x);
%如果求最小值 可以 1/ 10*sin(5*x)+7*cos(4*x);
end
ref
https://blog.csdn.net/xuehuafeiwu123/article/details/52327559
https://blog.csdn.net/lee_shuai/article/details/52510713
https://blog.csdn.net/on2way/article/details/40084449
https://blog.csdn.net/on2way/article/details/40084581