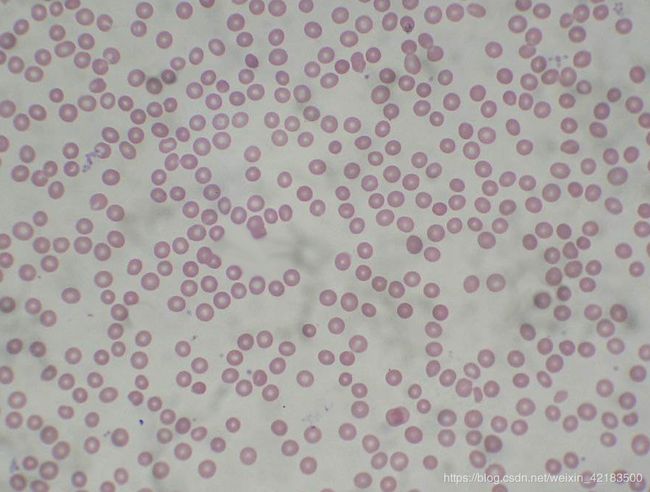
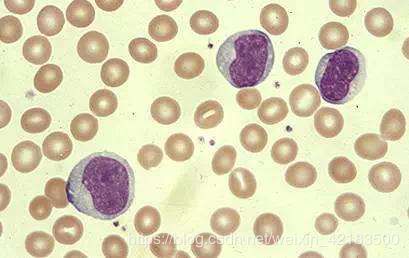
医学图像处理之python opencv实用范例源码
开发环境:
python3.6 + opencv-python 4.2
opencv-python 版的whl文件安装包下载地址: http://mirrors.aliyun.com/pypi/simple/opencv-python/
细胞计数的范例代码网上很多,但偏书面性和学习性,而且错误多形成误导,下面来点实用的.
一.快速上手一个最简范例:
上面左边是输入图像,右边是输出结果图(识别到的细胞总数)
下面代码运行的中间处理图像,用于调试观察:
主处理过程: 1.灰度化 -----> 2.二值化-----> 3.形态学运算-----> 4.轮廓查找
import cv2
import numpy as np
img=cv2.imread(r'111.png',cv2.IMREAD_COLOR) #读入一张RGB图片,忽略ALPHA通道
cv2.imshow("draw00",img)
gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY) #将图片转为灰度图片来分析
gray1 = cv2.GaussianBlur(gray,(3,3),0)# 高斯滤波(低通,去掉高频分量,图像平滑)
gray1 = cv2.GaussianBlur(gray1,(3,3),0)
#上面处理了多次,为消除细胞体内的浓淡深浅差别,避免在后续运算中产生孔洞
gray2=255-gray1 #0~255反相,为了适应腐蚀算法
cv2.imshow("draw11",gray2)
#ret, thresh = cv2.threshold(gray2, 80,255, cv2.THRESH_BINARY) # 固定 阈值处理 二值化图像
ret, thresh = cv2.threshold(gray2, 0, 255, cv2.THRESH_BINARY + cv2.THRESH_OTSU)#自动阈值处理
cv2.imshow("draw22",thresh)
#下面为了去除细胞之间的粘连,以免把两个细胞计算成一个
kernel=np.ones((2,2),np.uint8) #进行腐蚀操作,kernel=初值为1的2*2数组
erosion=cv2.erode(thresh,kernel,iterations=10) #腐蚀:卷积核沿着图像滑动,如果与卷积核对应的原图像的所有像素值都是1,那么中心元素就保持原来的像素值,否则就变为零。
cv2.imshow("draw33",erosion)
#下面为恢复一点SIZE,因为上面多次腐蚀,块太小了,所以这里膨胀几次
dilation = cv2.dilate(erosion,kernel,iterations = 5)
cv2.imshow("draw44",dilation)
contours,hirearchy=cv2.findContours(dilation, cv2.RETR_TREE, cv2.CHAIN_APPROX_SIMPLE)
# 上面查找出块轮廓(实现细胞计数)
#对连通域面积进行比较
contours_out=[] #建立空list,放减去最小面积的数
for i in contours:
if (cv2.contourArea(i)>2 and cv2.contourArea(i)<3000 and i.shape[0]>2):
# 排除面积太小或太大的块轮廓 而且排除 "点数不足2"的离散点块
contours_out.append(i)
total_num=len(contours_out)
print("Count=%d"%total_num) #输出计算细包个数
#下面生成彩色结构的灰度图像,为的是在上面画彩色标注
color_of_gray_img=cv2.cvtColor(dilation,cv2.COLOR_GRAY2BGR)
cv2.drawContours(color_of_gray_img,contours_out,-1,(50,50,250),2) #用红色线,描绘块轮廓
#求连通域重心 以及 在重心坐标点描绘数字
for i,j in zip(contours_out,range(total_num)):
M = cv2.moments(i)
cX=int(M["m10"]/M["m00"])
cY=int(M["m01"]/M["m00"])
cv2.putText(color_of_gray_img, str(j+1), (cX+10, cY), 1,1, (50, 250, 50), 1) #在中心坐标点上描绘数字
strout="Count=%d"%total_num #输出计算细包个数
print(strout)
cv2.putText(color_of_gray_img, strout, (2, 12), 1,1, (250, 150, 150), 1)
#输出结果图片
cv2.imshow("draw55",color_of_gray_img)
cv2.waitKey()
#cv2.destroyWindow()
#输入图像你可以直接抓图(上面那张),存成jpg或png,和上面的python代码放在同一个目录下运行.
如果上面的源码你已经运行成功了,那么恭喜你,可以进行下一步了.
二.进阶篇,从学习阶段,向实用阶段的过渡:
上面三张图片差异很大,用上面的花枪代码就不行了,下面上兼容性强的代码,能一定程度上打断细胞粘连:
#-----全代码开始------#
import cv2
import numpy as np
import matplotlib.pyplot as plt
def fill_food(img):
#这是网上下的一个简要的孔洞填充函数,在边界上有问题,不能产品化,需要改进
#而且有的细胞没有完全闭合的孔洞它不能填好(在不能用闭运算的前提下,需另行单独开发填洞算子)
contours, hierarchy = cv2.findContours(img, cv2.RETR_TREE, cv2.CHAIN_APPROX_SIMPLE)
h,w=img.shape[:2]
max_area_lmit=h*w/4
min_area_lmit=2
len_contour = len(contours)
contour_list = []
drawing = np.zeros_like(img, np.uint8)
for i in range(len_contour):
if (cv2.contourArea(contours[i])>min_area_lmit and cv2.contourArea(contours[i])
and contours[i].shape[0]>2):
img_contour = cv2.drawContours(drawing, contours, i, (255, 255, 255), -1)
contour_list.append(img_contour)
out = sum(contour_list)
return out
#下面这个是固定长度的,也不适用于图像大小变化的情况,需要改进,而且计算速度慢,但切断粘连细胞的效果很好.
def clear_bridge_line(img,iterations): #清除桥接线,这里用的是9点模版,左右各2个点是桥头,中间5个点为桥身
h,w=img.shape[:2]
ibo=img.copy()
ib=ibo.copy()
for n in range(iterations):
for r in range(9,h-9):
for c in range(9,w-9):
if( (ib[r,c]==255 and ib[r,c+1]==255 and ib[r,c+2]==255 and ib[r,c-1]==255 and ib[r,c-2]==255) \
and ( ib[r,c+3]==255 and ib[r,c+4]==255 and ib[r,c-3]==255 and ib[r,c-4]==255 ) \
and ( (ib[r-1,c]==0 and ib[r-1,c+1]==0 and ib[r-1,c+2]==0 and ib[r-1,c-1]==0 and ib[r-1,c-2]==0) \
or (ib[r+1,c]==0 and ib[r+1,c+1]==0 and ib[r+1,c+2]==0 and ib[r+1,c-1]==0 and ib[r+1,c-2]==0) )):
ibo[r,c]= ibo[r,c+1]= ibo[r,c+2]= ibo[r,c-1]= ibo[r,c-2]=0 #删除横线桥
c+=2
for c in range(9,w-9):
for r in range(9,h-9):
if( (ib[r,c]==255 and ib[r+1,c]==255 and ib[r+2,c]==255 and ib[r-1,c]==255 and ib[r-2,c]==255) \
and ( ib[r+3,c]==255 and ib[r+4,c]==255 and ib[r-3,c]==255 and ib[r-4,c]==255 ) \
and ( (ib[r,c+1]==0 and ib[r+1,c+1]==0 and ib[r+2,c+1]==0 and ib[r-1,c+1]==0 and ib[r-2,c+1]==0) \
or (ib[r,c-1]==0 and ib[r+1,c-1]==0 and ib[r+2,c-1]==0 and ib[r-1,c-1]==0 and ib[r-2,c-1]==0) )):
ibo[r,c]= ibo[r+1,c]= ibo[r+2,c]= ibo[r-1,c]= ibo[r-2,c]=0#删除竖线桥
r+=2
print("del_bridge_line iterations step %g/%g ok." % (n+1,iterations))
ib=ibo.copy()
return ibo
#主函数
def main_call(_filename,_kernel_size=2,_gblur_count=1,_erode_count=8, _del_brdige_count=4,_open_count=3,_is_show_dbg_img=True):
#这里可以把图像换成111,也能正常计数,有较好的普适性,但上一个简单算法就不能正常计数222.png这个图像
img_src=cv2.imread(_filename,cv2.IMREAD_COLOR) #读入一张RGB图片,忽略ALPHA通道
if(_is_show_dbg_img):cv2.imshow("draw00",img_src)#绘制原图
KS=_kernel_size
img_gray = cv2.cvtColor(img_src,cv2.COLOR_BGR2GRAY) #将图片变为灰度图片来分析
histb = cv2.calcHist([img_gray], [0], None, [256], [0,255])
if(_is_show_dbg_img):plt.plot(histb)#绘制一下灰度直方图,用于辅助观察分析
'''
img_gray = cv2.equalizeHist(img_gray)
cv2.imshow("draw01",img_gray)#绘制均衡化之后的图像
histb2 = cv2.calcHist([img_gray], [0], None, [256], [0,255])
plt.plot(histb2)#绘制一下灰度直方图,用于辅助观察分析
'''
gblur_count=_gblur_count #可调节的参数->高斯滤波次数
img_blur=img_gray
for n in range(gblur_count):
img_blur = cv2.GaussianBlur(img_blur,(3,3),0)# 高斯滤波(低通,去掉高频分量,图像平滑)
# 可以按需多做一次或N次
img_inverse=255-img_blur #0~255反相,为了适应后面的腐蚀算法
if(_is_show_dbg_img):cv2.imshow("draw11",img_inverse)
#ret, img_thresh = cv2.threshold(img_inverse, 240,250, cv2.THRESH_BINARY) #固定阈值处理成二值化图像
ret, img_thresh = cv2.threshold(img_inverse, 0, 255, cv2.THRESH_BINARY + cv2.THRESH_OTSU)
#上面用大律法,自动从直方图的波谷选一个阈值做二值切分
#但它不是通吃法,比如第三张红细胞样图,只有用固定阀值才能正确计数
if(_is_show_dbg_img):cv2.imshow("draw22",img_thresh)
print("开始进行孔洞填充.....")
img_fill=fill_food(img_thresh)#孔洞填充
if(_is_show_dbg_img):cv2.imshow("draw33",img_fill)
#下面为了去除细胞之间的粘连,以免把两个细胞计算成一个
kernel_std=np.ones((KS,KS),np.uint8) #进行腐蚀操作,kernel=初值为1的2*2数组
#old_ibo=cv2.erode(img_fill,kernel=kernel_std,iterations=10)#这个老算法没有先处理边界,效果不好看
#cv2.imshow("draw44_old",old_ibo)
print(img_fill.shape)
#下面为了在四边上做好卷积而做的padding
img_padding=cv2.copyMakeBorder(img_fill, top=2, bottom=2, left=2, right=2, borderType= cv2.BORDER_CONSTANT, value=0 )
erode_count=_erode_count #可调节的参数->首发腐蚀次数
print("开始进行%g次CV2卷积腐蚀....."%erode_count)
#注意:必要至少5次以上的连续腐蚀,才能形成中间连接宽度为>=5的细胞粘连桥接直线,与clear_bridge_line函数的固定长度相匹配
img_erode=cv2.erode(img_padding,kernel=kernel_std,iterations=erode_count) #腐蚀:卷积核沿着图像滑动,如果与卷积核对应的原图像的所有像素值都是1,那么中心元素就保持原来的像素值,否则就变为零。
if(_is_show_dbg_img):cv2.imshow("draw44_new",img_erode)
print(img_erode.shape)
nh,nw=img_erode.shape[:2]
img_no_padding=img_erode[2:nh-2,2:nw-2] #这里是padding还原
print(img_no_padding.shape)
del_brdige_count=_del_brdige_count #可调节的参数->删除细胞间的残连直线次数
print("请稍等,因为没有用多线程/CPU加速/GPU加速.....")
print("开始进行%g次细胞粘连桥接直线删除:"%del_brdige_count)
img_del_brdige=clear_bridge_line(img_no_padding,del_brdige_count)
if(_is_show_dbg_img):cv2.imshow('draw44_del_bridge_line_over', img_del_brdige)
#下面再来几次开运算操作,提高图像视觉圆滑效果(注意,迭代次数过多,或kernel面积过大,都可能会把一些小面积细胞弄消失)
open_count=_open_count #可调节的参数->最后开运算次数
kernel_open = np.ones((KS,KS), np.uint8)
print("开始进行%g次形态学开运算....."%open_count)
image_opening = cv2.morphologyEx(img_del_brdige, cv2.MORPH_OPEN, kernel_open,iterations=open_count)
if(_is_show_dbg_img):cv2.imshow("draw55",image_opening)
h,w=image_opening.shape[:2]
max_area_lmit=h*w/4
min_area_lmit=2
#加宽边界,为了多打字
img_padding=cv2.copyMakeBorder(image_opening, top=20, bottom=10, left=10, right=60, borderType= cv2.BORDER_CONSTANT, value=0 )
contours,hirearchy=cv2.findContours(img_padding, cv2.RETR_TREE, cv2.CHAIN_APPROX_SIMPLE)# 找出块轮廓
#对连通域面积进行比较
contours_out=[] #建立空list,放减去最小面积的数
for i in contours:
if (cv2.contourArea(i)>min_area_lmit and cv2.contourArea(i)
# 排除面积太小或太大的块轮廓 而且排除 "点数不足2"的离散点块
# 这里还可以利用面积和点数/密度,对细胞进行初分类,CV提供了丰富的块属性值供我们读取
contours_out.append(i)
#下面生成彩色结构的灰度图像,为的是在上面画彩色标注
color_of_gray_img=cv2.cvtColor(img_padding,cv2.COLOR_GRAY2BGR)
cv2.drawContours(color_of_gray_img,contours_out,-1,(50,50,250),2) #用红色线,描绘块轮廓
#求连通域重心 以及 在重心坐标点描绘数字
total_num=len(contours_out)
for i,j in zip(contours_out,range(total_num)):
M = cv2.moments(i)
cX=int(M["m10"]/M["m00"])
cY=int(M["m01"]/M["m00"])
cv2.putText(color_of_gray_img, str(j+1), (cX+10, cY), 1,1, (50, 250, 50), 1) #在中心坐标点上描绘数字
strout="Count=%d"%total_num #输出计算细包个数
#print(strout)
cv2.putText(color_of_gray_img, strout, (2, 12), 1,1, (250, 150, 150), 1)
#展示最终结果图片
if(_is_show_dbg_img):
cv2.imshow("draw66",color_of_gray_img)
#改变显示位置
cv2.moveWindow("draw00",0,0)
cv2.moveWindow("draw11",50,50)
cv2.moveWindow("draw22",100,100)
cv2.moveWindow("draw33",150,150)
cv2.moveWindow("draw44_new",200,200)
cv2.moveWindow("draw44_del_bridge_line_over",250,250)
cv2.moveWindow("draw55",300,300)
cv2.moveWindow("draw66",350,350)
return total_num
#可调节参数区
_gblur_count=1 #图像预处理时的高斯滤波次数
_kernel_size=2 #腐蚀与膨胀的核大小,根据图像中细胞的大小来调节
_erode_count=8 #腐蚀次数,少了细胞会连在一起,多个计算作一个;但腐蚀次数多了细胞会小到消失从而不能计入总数
_del_brdige_count=4 #删除细胞间的连接线的次数,多了速度慢(这个是最卡的),少了也会是细胞连在起,是否启用要看调试图上,是否有细胞连线存在
_open_count=3 #后期开运算次数,少了细胞会连在一起计算成一个;多了细胞会小到消失从而不能计入总数
_is_show_dbg_img=True #是否显示中间步骤调试图像
#可调节参数区
#主函数调用:
total_num=main_call("333.png",_kernel_size,_gblur_count,_erode_count,_del_brdige_count,_open_count,_is_show_dbg_img)
print("zhuwei->图像处理完成")
print("zhuwei->细胞计数(Count) = %g" % total_num )
cv2.waitKey()
#cv2.destroyWindow()
#-----全代码结束------#
注意:代码我是肯定全跑通了的,并测试了十张以上不同风格的细胞图像,都是微调参数而已,其中一张必要做固定阀值,如果运行报错,多半是代码中含隐形的ASCII编码,一个你要先做好对齐,一个就是想办法删除那些隐形编码(看起来像空格和TAB),然后再运行就OK了.
上图就是经过6次以上腐蚀后,形成的细胞桥接直线,用像素模板法clear_bridge_line清除,以保证正确的细胞计数
因为过多的腐蚀会让一些小细胞消失.
三.从实验到产品的过渡:
上面的代码也只能说勉强能应用到实践中,到了这一步,我们离真正的产品化都还有很长的距离:
首先就是解决算法的速度优化和调用接口及移植的问题.然后才是将深度学习网络模型,比如简单的alexnet或复杂的yolov3引入,解决细胞的分类识别,解决团块的分类识别(两个粘在一起算一个类,三个在一起又算一个类,四个都又一类,如此类推,这样才能更准确地进行计数)