基于R语言的随机森林算法运用
有关数据挖掘中的分类算法有很多,如贝叶斯判别法、Fisher判别法、决策树、支持向量机和随机森林等,本文将对随机森林做一个介绍,并使用R语言实现该算法的应用。
随机森林算法的实质是基于决策树的分类器集成算法,其中每一棵树都依赖于一个随机向量,随机森林的所有向量都是独立同分布的。随机森林就是对数据集的列变量和行观测进行随机化,生成多个分类数,最终将分类树结果进行汇总。
随机森林相比于神经网络,降低了运算量的同时也提高了预测精度,而且该算法对多元共线性不敏感以及对缺失数据和非平衡数据比较稳健,可以很好地适应多达几千个解释变量数据集。
随机森林的组成
随机森林是由多个CART分类决策树构成,在构建决策树过程中,不进行任何剪枝动作,通过随机挑选观测(行)和变量(列)形成每一棵树。对于分类模型,随机森林将根据投票法为待分类样本进行分类;对于预测模型,随机森林将使用单棵树的简单平均值来预测样本的Y值。
随机森林的估计过程
1)指定m值,即随机产生m个变量用于节点上的二叉树,二叉树变量的选择仍然满足节点不纯度最小原则;
2)应用Bootstrap自助法在原数据集中有放回地随机抽取k个样本集,组成k棵决策树,而对于未被抽取的样本用于单棵决策树的预测;
3)根据k个决策树组成的随机森林对待分类样本进行分类或预测,分类的原则是投票法,预测的原则是简单平均。
随机森林性能因素
1)每棵树生长越茂盛,组成森林的分类性能越好;
2)每棵树之间的相关性越差,或树之间是独立的,则森林的分类性能越好。
主要函数
R语言中的randomForest包可以实现随机森林算法的应用,该包中主要涉及5个重要函数,关于这5个函数的语法和参数请见下方:
1)randomForest()函数用于构建随机森林模型
randomForest(formula, data=NULL, ..., subset, na.action=na.fail)
randomForest(x, y=NULL, xtest=NULL, ytest=NULL, ntree=500,
mtry=if (!is.null(y) && !is.factor(y))
max(floor(ncol(x)/3), 1) else floor(sqrt(ncol(x))),
replace=TRUE, classwt=NULL, cutoff, strata,
sampsize = if (replace) nrow(x) else ceiling(.632*nrow(x)),
nodesize = if (!is.null(y) && !is.factor(y)) 5 else 1,
maxnodes = NULL,
importance=FALSE, localImp=FALSE, nPerm=1,
proximity, oob.prox=proximity,
norm.votes=TRUE, do.trace=FALSE,
keep.forest=!is.null(y) && is.null(xtest), corr.bias=FALSE,
keep.inbag=FALSE, ...)
formula指定模型的公式形式,类似于y~x1+x2+x3...;
data指定分析的数据集;
subset以向量的形式确定样本数据集;
na.action指定数据集中缺失值的处理方法,默认为na.fail,即不允许出现缺失值,也可以指定为na.omit,即删除缺失样本;
x指定模型的解释变量,可以是矩阵,也可以是数据框;y指定模型的因变量,可以是离散的因子,也可以是连续的数值,分别对应于随机森林的分类模型和预测模型。这里需要说明的是,如果不指定y值,则随机森林将是一个无监督的模型;
xtest和ytest用于预测的测试集;
ntree指定随机森林所包含的决策树数目,默认为500;
mtry指定节点中用于二叉树的变量个数,默认情况下数据集变量个数的二次方根(分类模型)或三分之一(预测模型)。一般是需要进行人为的逐次挑选,确定最佳的m值;
replace指定Bootstrap随机抽样的方式,默认为有放回的抽样;
classwt指定分类水平的权重,对于回归模型,该参数无效;
strata为因子向量,用于分层抽样;
sampsize用于指定样本容量,一般与参数strata联合使用,指定分层抽样中层的样本量;
nodesize指定决策树节点的最小个数,默认情况下,判别模型为1,回归模型为5;
maxnodes指定决策树节点的最大个数;
importance逻辑参数,是否计算各个变量在模型中的重要性,默认不计算,该参数主要结合importance()函数使用;
proximity逻辑参数,是否计算模型的临近矩阵,主要结合MDSplot()函数使用;
oob.prox是否基于OOB数据计算临近矩阵;
norm.votes显示投票格式,默认以百分比的形式展示投票结果,也可以采用绝对数的形式;
do.trace是否输出更详细的随机森林模型运行过程,默认不输出;
keep.forest是否保留模型的输出对象,对于给定xtest值后,默认将不保留算法的运算结果。
2)importance()函数用于计算模型变量的重要性
importance(x, type=NULL, class=NULL, scale=TRUE, ...)
x为randomForest对象;
type可以是1,也可以是2,用于判别计算变量重要性的方法,1表示使用精度平均较少值作为度量标准;2表示采用节点不纯度的平均减少值最为度量标准。值越大说明变量的重要性越强;
scale默认对变量的重要性值进行标准化。
3)MDSplot()函数用于实现随机森林的可视化
MDSplot(rf, fac, k=2, palette=NULL, pch=20, ...)
rf为randomForest对象,需要说明的是,在构建随机森林模型时必须指定计算临近矩阵,即设置proximity参数为TRUE;
fac指定随机森林模型中所使用到的因子向量(因变量);
palette指定所绘图形中各个类别的颜色;
pch指定所绘图形中各个类别形状;
还可以通过R自带的plot函数绘制随机森林决策树的数目与模型误差的折线图
4)rfImpute()函数可为存在缺失值的数据集进行插补(随机森林法),得到最优的样本拟合值
rfImpute(x, y, iter=5, ntree=300, ...)
rfImpute(x, data, ..., subset)
x为存在缺失值的数据集;
y为因变量,不可以存在缺失情况;
iter指定插值过程中迭代次数;
ntree指定每次迭代生成的随机森林中决策树数量;
subset以向量的形式指定样本集。
5)treesize()函数用于计算随机森林中每棵树的节点个数
treesize(x, terminal=TRUE)
x为randomForest对象;
terminal指定计算节点数目的方式,默认只计算每棵树的根节点,设置为FALSE时将计算所有节点(根节点+叶节点)。
一般treesize()函数生成的结果用于绘制直方图,方面查看随机森林中树的节点分布情况。
应用
在随机森林算法的函数randomForest()中有两个非常重要的参数,而这两个参数又将影响模型的准确性,它们分别是mtry和ntree。一般对mtry的选择是逐一尝试,直到找到比较理想的值,ntree的选择可通过图形大致判断模型内误差稳定时的值。
下面使用坦泰尼克号乘客是否存活数据应用到随机森林算法中,看看模型的准确性如何。
library(randomForest)
#读取训练样本和测试样本
train <- read.table(file = 'C:\\Users\\admin\\Desktop\\train.csv', head = TRUE, sep = ',')
test <- read.table(file = 'C:\\Users\\admin\\Desktop\\test.csv', head = TRUE, sep = ',')
#数据结构信息
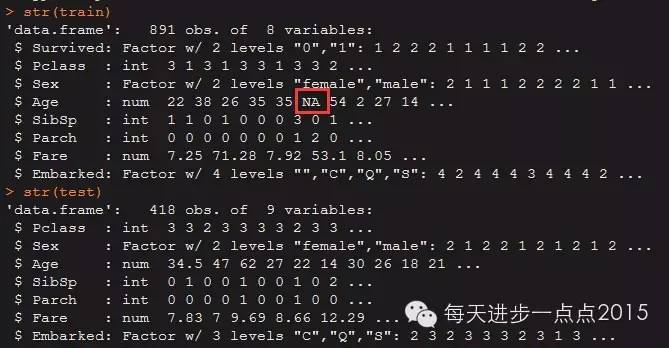
str(train)
str(test)
发现train数据集中存在缺失值。
#数据类型变换
train$Survived <- as.factor(train$Survived)
#使用rfImpute()函数补齐缺失值
train_impute <- rfImpute(Survived~., data = train)
#选取随机森林mtry值
n <- length(names(train))
set.seed(1234)
for (i in 1:(n-1)){
model <- randomForest(Survived~., data = train_impute, mtry = i)
err <- mean(model$err.rate)
print(err)
}
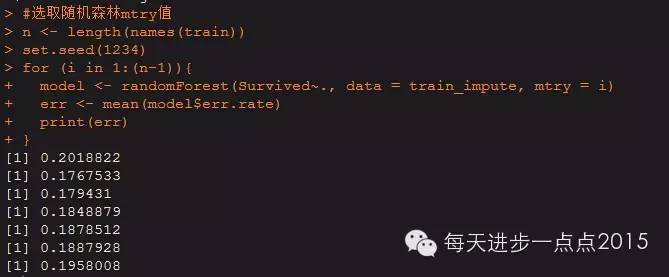
发现当mtry=2时,模型内平均误差最小,故确定参数mtry=2。
#选取ntree值
set.seed(1234)
model <- randomForest(Survived~., data = train_impute, mtry = 2, ntree=1000)
plot(model)
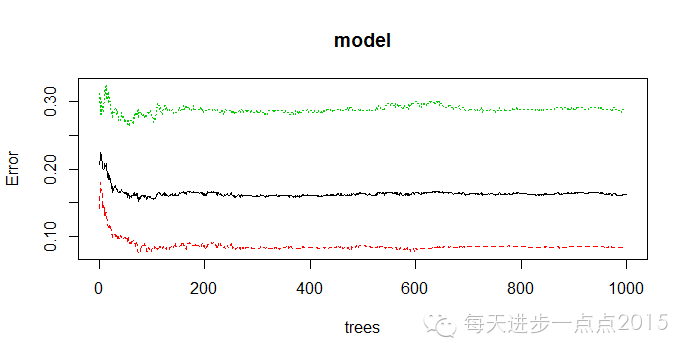
发现ntree在400左右时,模型内误差基本稳定,故取ntree=400。
set.seed(1234)
fit <- randomForest(Survived~., data = train_impute, mtry = 2, ntree=400, importance = TRUE)
fit
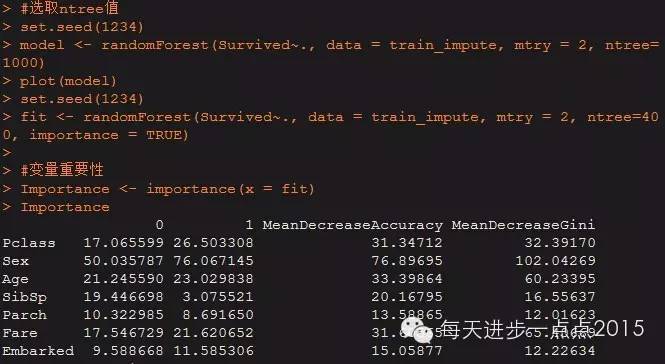
#变量重要性
Importance <- importance(x = fit)
Importance
varImpPlot(fit)
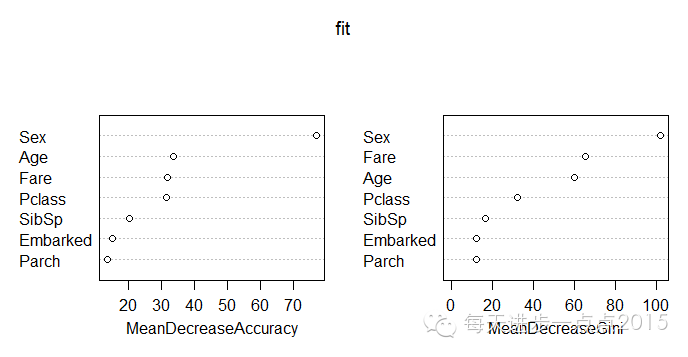
从返回的数据和图形可知,模型中乘客的性别字段最为重要,接下来是年龄(票价)和票价(年龄)。
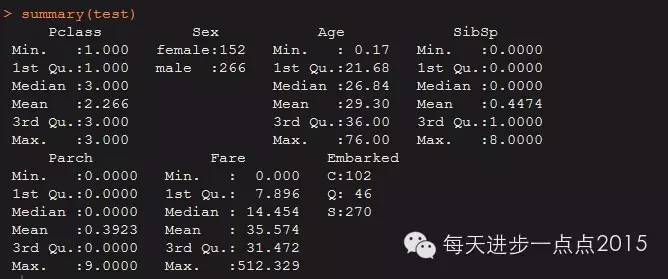
对待测样本进行预测,发现待测样本中存在缺失值,这里使用多重插补法将缺失值补齐。
library(mice)
Imput <- mice(data = test, m = 10)
Age <- data.frame(Age = apply(Imput$imp$Age,1,mean))
Fare <- data.frame(Fare = apply(Imput$imp$Fare,1,mean))
#添加行标号
test$Id <- row.names(test)
Age$Id <- row.names(Age)
Fare$Id <- row.names(Fare)
#替换缺失值
test[test$Id %in% Age$Id,'Age'] <- Age$Age
test[test$Id %in% Fare$Id,'Fare'] <- Fare$Fare
summary(test)
#原数据集模型的准确率分析
pred1 <- predict(object = fit, newdata = train_impute)
Freq1 <- table(pred1,train_impute$Survived)
Freq1
sum(diag(F))/sum(F)
#新数据集的预测情况
pred2 <- predict(object = fit, newdata = test)
这里报错,报错的原因是用于建立模型的数据集和test数据集不一致。test数据集中缺少Survived变量,我在想,难道对于新的数据集(没有因变量)随机森林就无法预测了吗?还请阅读者给予帮助和建议。
参考资料:
http://www.36dsj.com/archives/21036
数据挖掘:R语言实战