GraPhlAn教程中文版——超炫物种树进化树绘制
文章目录
- GraPhlAn教程中文版
- 概述Overview
- 介绍Introduction
- 安装Installation
- 方法1. Bioconda快速安装
- 方法2. Mercurial下载
- 方法3. 直接下载压缩包
- 脚本Scripts
- 分步示例Step-by-step example
- 0绘图 Default plot (step 0)
- 1全局选项 Global options (step 1)
- 2节点选项 Node options (step 2)
- 3标签选项 Label options (step 3)
- 4外部选项 External options (step 4)
- 其它示例Other examples
- 人类微生物组计划Human Microbiome Project
- 肠道微生物组 Gut microbiome
- 菌株水平分析 PhyloPhlAn
- 炎症性肠病生物地理 IBD biogeography
- 完整参数列表 Complete list of parameters
- 支持 Support
- 附录
- 附录1. 颜色
- 附录2. 形状选择
- Reference
- 猜你喜欢
- 写在后面
GraPhlAn教程中文版
GraPhlAn tutorial原文链接:https://bitbucket.org/nsegata/graphlan
GraPhlAn是一个软件工具,可用于生成分类树和系统发育树的高质量圆形表示形式。 它着重于系统发育和分类学驱动的研究的简洁、整合、信息丰度信,输出结果图片可直接用于高水平文章发表使用。
概述Overview
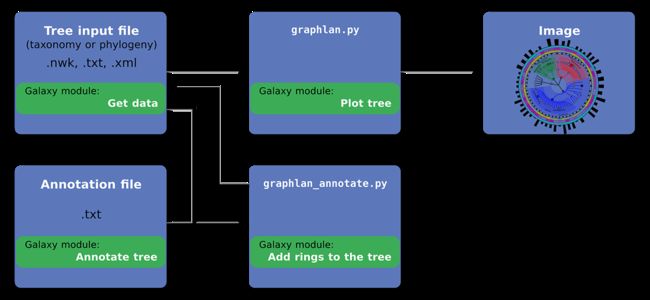
下图报告了GraPhlAn结构的简要概述:
介绍Introduction
请按照本教程学习如何使用GraPhlAn。
安装Installation
GraPhlAn保存于bitbucket网站,可以通过以下两种方法获取:
方法1. Bioconda快速安装
conda install graphlan
方法2. Mercurial下载
在Linux系统中需要安装mercurial,如Ubuntu中sudo apt install mercurial,才可以使用hg命令
hg clone ssh://[email protected]/nsegata/graphlan
或者
hg clone https://[email protected]/nsegata/graphlan
方法3. 直接下载压缩包
详者注:其也可以不安装Mercurial直接下载。在 https://bitbucket.org/nsegata/graphlan/downloads/ 页面有软件打包的下载链接:
wget -c https://bitbucket.org/nsegata/graphlan/downloads/graphlan_commit_6ca8735.zip
# 下载失败请在windows上访问此链接下载
unzip graphlan_commit_6ca8735.zip
mv graphlan_commit_6ca8735 graphlan
这将在graphlan子文件夹中本地下载GraPhlAn相关数据。 然后,您必须将此子文件夹放入系统路径,以便可以从系统中的任何位置使用GraPhlAn:
export PATH=`pwd`/graphlan/:$PATH
将以上行添加到bash配置文件中将使添加路径永久生效。 对于Windows或MacOS系统,应遵循类似的步骤。
脚本Scripts
GraPhlAn主要包括两个脚本:
- graphlan_annotate.py
- graphlan.py
第一个脚本 (graphlan_annotate.py) 用于在输入树文件中添加注释信息。
graphlan_annotate.py --annot annotation_file.txt input_file.txt new_input_file_name.xml
注释文件可以多次添加至输入树文件。
第二个脚本(graphlan.py)用于绘图,支持 png, pdf, ps, eps, svg 等格式。可使用–dpi设置分辨率 (默认值为 72),使用–size调节图片尺寸 (默认为7.0英寸)。
graphlan.py new_input_file_name.xml image_name.png --dpi 150 --size 7
graphlan.py new_input_file_name.xml image_name.svg --dpi 150 --size 7
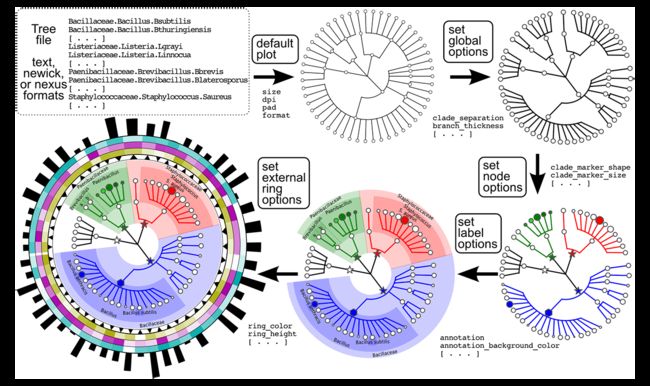
分步示例Step-by-step example
本分步教程基于您可以在examples文件夹(快速链接)内找到的guide示例。
指南文件夹包含以下文件:
- guide.txt
- step0.sh
- annot_0.txt
- step1.sh
- annot_1.txt
- step2.sh
- annot_2.txt
- step3.sh
- annot_3.txt
- step4.sh
如果需要更多帮助,可以查看每个文件.txt或.sh。 我们决定为每个步骤行添加一个注释(#comment),以指定您将要使用以下编写的自定义内容。
我们下面进入示例目录
cd graphlan/examples/guide
0绘图 Default plot (step 0)
当你执行如下命令
./step0.sh
你可以看到下图:
step0.sh执行了如下两个绘图命令:
# 以guide.txt为输入文件,输出位图step_0.png,指定分辨率300和大小宽3.5英寸
graphlan.py guide.txt step_0.png --dpi 300 --size 3.5
# 绘制svg矢量,方便AI编辑
graphlan.py guide.txt step_0.svg --dpi 300 --size 3.5
这是GraPhlAn的最基本输出(树骨架),可以采用注释进一步美化。
我们看到这是一个三层的物种分类树,输入文件是什么格式呢?
head guide.txt
是以点(.)为分隔的物种层级注释文件,我们常用的是7级注释。本文的示例是采用了科、属、种三级注释。
Bacillaceae.Anoxybacillus.Aflavithermus
Bacillaceae.Bacillus.Bamyloliquefaciens
Bacillaceae.Bacillus.Banthracis
Bacillaceae.Bacillus.Batrophaeus
Bacillaceae.Bacillus.Bcellulosilyticus
Bacillaceae.Bacillus.Bcereus
Bacillaceae.Bacillus.Bclausii
Bacillaceae.Bacillus.Bcoagulans
Bacillaceae.Bacillus.Bcoahuilensis
Bacillaceae.Bacillus.Bhalodurans
1全局选项 Global options (step 1)
运行下行命令
./step1.sh
你可以看到下图:
和上图相比,是不是明显线条更粗了,也更美观些。
step1.sh脚本执行以下命令:
# 在guide.txt树上添加annot_0.txt的注释,输出整合文件为guide_1.xml
graphlan_annotate.py --annot annot_0.txt guide.txt guide_1.xml
# 绘制位图png
graphlan.py guide_1.xml step_1.png --dpi 300 --size 3.5
graphlan.py guide_1.xml step_1.svg --dpi 300 --size 3.5
第一个命令将annot_0.txt中的设置绑定到guide.txt树,从而生成一个新树:guide_1.xml。后面的命令仅生成两个图像:.png和.svg。
annot_0.txt包含以下定制参数:
clade_separation 0.5
branch_thickness 1.5
branch_bracket_depth 0.8
branch_bracket_width 0.25
clade_marker_size 40
clade_marker_edge_color #555555
clade_marker_edge_width 1.2
它们的意思分别是:
- clade_separation 指定分支之间的分数间隔,该间隔与子树之间的分支距离成比例。该选项可用于在视觉上分离更多相互分支深的分支。默认值为0.0。本次设为0.5,上图各分枝类间的距离大于内部枝间距离,有各明显的聚类感觉,也方便为标签预留空间。
- branch_thickness 设置连接分类单元的线的全局粗细。默认值为0.75。本次设为1.5,线宽比默认加粗了一倍。
- branch_bracket_depth 设置分支托架的相对位置,该分支托架是子分类单元分支所起源的径向段。默认值为1(官网标注为0.25是错误的),分枝从节点延伸的长度,范围0-1,大于1会超过上一节点显示错误,这里0.8比默认要好,层级关系更明确;
- branch_bracket_width 设置相对于最分开的子根的位置的分支括号的宽度。默认值为0.25(官网标注为1是错误的)。即分枝矩形上边的宽,1为矩形,0为三角形;本次使用0.8为梯形分枝比较美观,画面留白较少而且有成簇感。
- clade_marker_size:设置代表树内进化枝根的标记的大小。默认值为20.0。节点圈的大小。本次使用40比默认大一倍,最后可根据图形整体大小调整优化。
- clade_marker_edge_color 设置标记边框的颜色。默认值为**#000000(即黑色),节点圈的颜色,这里使用#555555(灰色)与枝分开**。
- clade_marker_edge_width 设置进化枝标记的边框粗细。默认值为0.5,使用1.2加粗边框些。
2节点选项 Node options (step 2)
执行时
./step2.sh
您将获得以下树:
step2.sh脚本执行以下命令:
graphlan_annotate.py --annot annot_1.txt guide_1.xml guide_2.xml
graphlan.py guide_2.xml step_2.png --dpi 300 --size 3.5
graphlan.py guide_2.xml step_2.svg --dpi 300 --size 3.5
第一个命令将annot_1.txt文件与上一步生成的树:guide_1.xml 绑定在一起,将结果保存在guide_2.xml中。后两个命令将生成两种格式的输出图像。
annot_1.txt包含以下自定义:
Bacillus clade_marker_color b
Bacillus clade_marker_size 120
Bacillus clade_marker_shape h
这些规范适用于规则“ GRAPHICAL TREE OPTIONS(图型树选项)”,并具有以下语法:
[clade_name{+|*|^}] graphical_tree_option graphical_tree_option_value
如果省略了进化枝名称,则该选项将应用于所有进化枝。可以使用包含树根所有名称的完整标签或仅使用最后一级来指定进化枝(如果最后一级名称不是唯一的,则该命令会影响多个匹配进化枝)。
(可选)在进化枝名称的末尾,可以添加以下字符之一:+,*,^。其中
- *表示指定的进化枝及其所有后代均受该属性影响;
- +表示指定分支及其所有终端节点均受到影响;
- ^表示指定进化枝的所有(唯一)终端节点均受到影响。
annot_1.txt中使用的graphic_tree_option是:
- clade_marker_size 是表示树内进化枝根的标记的大小。默认值为20.0
- clade_marker_color 指定表示树内进化枝根的标记的填充色。默认值为 #FFFFFF(即白色),b代表blue蓝色
- clade_marker_shape 提供进化枝标记的形状。有关更多信息,请参见软件包中
readme.txt文件中的“ MARKER SHAPES”表,或见本文附录。默认值为’o’(即圆圈), h代表六角形
此外官网中还有些参数没説,但很常用
- clade_marker_label 设置节点内标签(文字),一般显示个1-2位数字非常好看
- clade_marker_edge_color 设置节点边颜色
- clade_marker_font_color 节点标签文字颜色,默认黑色
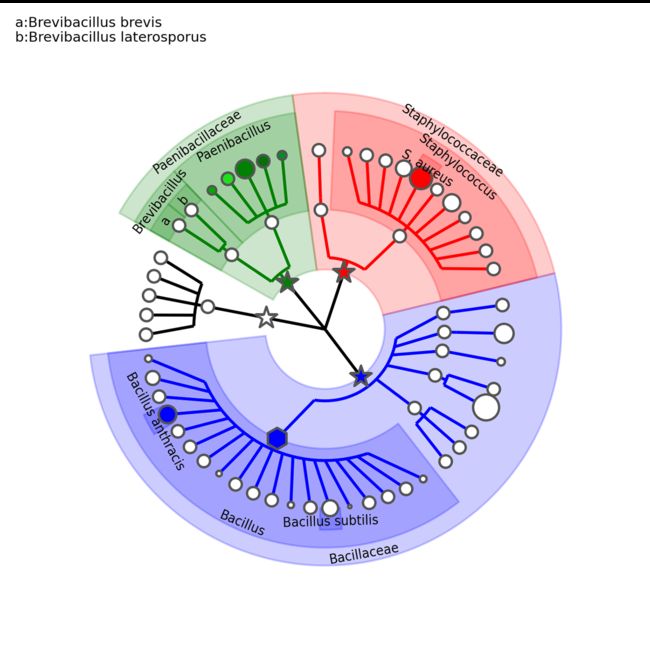
3标签选项 Label options (step 3)
执行时
./step3.sh
您将获得以下树:
step3.sh脚本执行以下命令:
graphlan_annotate.py --annot annot_2.txt guide_2.xml guide_3.xml
graphlan.py guide_3.xml step_3.png --dpi 300 --size 3.5
graphlan.py guide_3.xml step_3.svg --dpi 300 --size 3.5
第一个命令将annot_2.txt中的设置绑定到guide_2.xml树,从而生成一个新树:guide_3.xml。后面的命令仅生成两个图像:.png和.svg。
annot_2.txt包含以下定制内容:
# 注释文本和颜色
Bacillus annotation Bacillus
Bacillus annotation_background_color b
# 注释文本和缩写、颜色
Bbrevis annotation a:Brevibacillus brevis
Bbrevis annotation_background_color g
Blaterosporus annotation b:Brevibacillus laterosporus
Blaterosporus annotation_background_color g
这些规范适用于规则“ANNOTATION OPTIONS(注释选项)”,语法如下:
[clade_name] annotation_option graphical_tree_option_value
我们注释将阴影/背景色突出显示分支和相应的子树。注释可以是彩色的,它们的透明度(alpha)通道可以进行整体调节,并具有与之关联的标签。
具体而言,annot_2.txt中使用的graphical_tree_option_value为:
注释指定要与注释关联并显示的标签。这可以采用几种格式:
- str(不包含’:'的字符串)指定要完全(唯一)显示在阴影上的字符串
- key:str(应为短字符)将显示在注释底纹上,而完整的key:string标签将报告为外部图例(a/b对应完整名称的图例)
- *:str 将自动生成一个缩写并将其用作前一个key:str的情况
-
- 进化枝的名称(特别是最后一个分类学级别)将在上面的第一种情况下用作str
- : 代表上述前两种情况的组合,自动将全称变为缩写
annotation_background_color 定义注释背景的颜色。默认为灰色
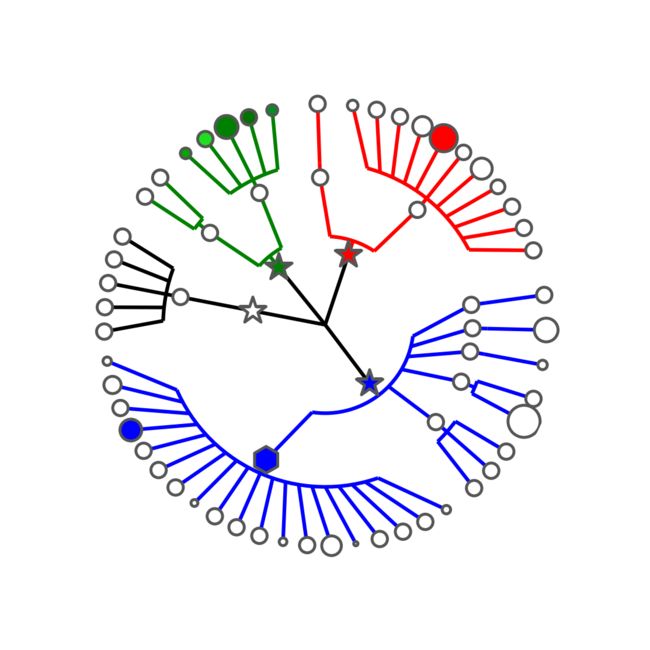
4外部选项 External options (step 4)
执行以下命令:
./step4.sh
您将获得以下树:
step4.sh脚本执行以下命令:
graphlan_annotate.py --annot annot_3.txt guide_3.xml guide_4.xml
graphlan.py guide_4.xml step_4.png --dpi 300 --size 3.5 --pad 0.0
graphlan.py guide_4.xml step_4.svg --dpi 300 --size 3.5 --pad 0.0
第一个命令将annot_3.txt中的设置绑定到guide_3.xml树,从而生成一个新树:guide_4.xml。后面的命令仅生成两个图像:.png和.svg。
annot_3.txt包含以下内容:
ring_internal_separator_thickness 1 0.5
[..]
ring_width 1 0.5
ring_height 1 0.75
[..]
Bacillaceae.Anoxybacillus.Aflavithermus ring_shape 1 v
[..]
Bacillaceae.Anoxybacillus.Aflavithermus ring_alpha 2 0.423462136478
[..]
Bacillaceae.Anoxybacillus.Aflavithermus ring_color 2 #AAAA00
[..]
Bacillaceae.Anoxybacillus.Aflavithermus ring_height 5 2.48900854747
[..]
Bacillaceae.Bacillus.Bsubtilis clade_marker_label 65
[..]
Bacillaceae.Bacillus.Banthracis clade_marker_font_color w
Bacillaceae.Bacillus.Banthracis clade_marker_edge_color b
[..]
这些规范适用于规则“RING OPTIONS(环选项)”。
我们称其为树本身外部的图形元素,可以看作是“圆形热图”,“圆形条形图”,甚至更多(类似于指示符元素)。这些“环”直接链接到内部树,因为这些环的每个部分都对应于一个树叶(并且还可能对应于内部节点)。可以为同一张图像指定多个环,每个环必须具有一个渐进的关联编号(级别“ 1”是最内部的环)。
环的一般语法为:
[clade_name] ring_option ring_level ring_option_value
如果clade_name不存在或如果*,则ring选项将应用于ring_level中的所有环扇区。 ring_level是必须始终指定的整数**。
**
在上面的示例中,ring_options在这里使用:
- ring_color 表示环段的颜色。默认为黑色
- ring_width 指定环段的宽度,该宽度是可用于特定进化枝的总圆形宽度的一部分。默认值为1.0
- ring_height 设置圆形线段的高度。如果未指定,则对级别中的所有圆环段应用相同的默认高度(*树的0.1 大小),否则高度等于级别中的最大高度值。默认值是同一级别中环的最高高度,如果未指定高度,则为0.1
- ring_alpha 设置透明度值。 0.0表示完全透明(因此不可见),1.0表示完全不透明。默认值为1.0
- ring_shape 指定环的形状。矩形的默认值为R,这意味着将使用整个可用区域。当前的替代项是v或^,表示三角形(具有相反方向),可以用作突出特定进化枝的指向箭头。默认为R
一些其他的环选项涉及非类特定的方面,例如环本身的标签或环之间的图形分隔。这些选项在没有树名的情况下以以下树列格式指定:
global_ring_option ring_level global_ring_option_value
- ring_internal_separator_thickness 设置分隔不同环级别的圆线的粗细。这是指每个环两侧最内部的部分。默认值为0.0,表示不存在
最后一行适用于规则“GRAPHICAL TREE OPTIONS”:
- clade_marker_edge_color 指定标记边框的颜色。默认值为#000000(即黑色)
- clade_marker_label 指定要插入指定进化枝的标签
- clade_marker_font_color 指定特定类标记标签的字体颜色。默认值为’k’,即黑色
其它示例Other examples
在这里,我们要显示其他示例(在程序的example示例文件夹中),以显示GraPhlAn提供的灵活性和几种自定义可能性。
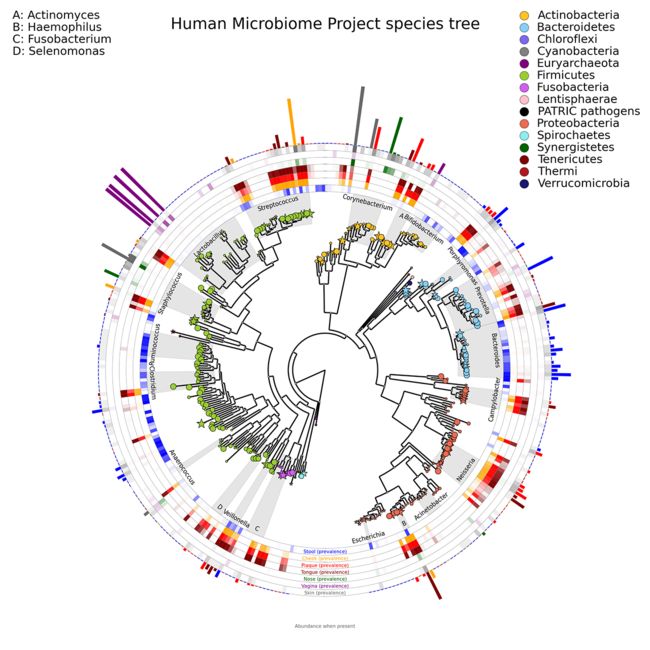
人类微生物组计划Human Microbiome Project
在人类微生物组中丰富的生物的系统树上使用环形热图和条形图。 颜色强度对应于每个身体部位的物种流行度; 圆外侧的条形高度与分类单元的丰度成正比(参考文献见 http://www.sciencedirect.com/science/article/pii/S016895251200145X)。
下图数据位于example中的HMP_tree文件夹。
详者注:上图中结点加形状、大小、颜色;外圈加热图、柱状图我们前面都有学过。
本次使用的是进化树,它和物种注释层级文件一样使用即可。关于进化树的GraPhlAn实战,请参阅我们之前的教程 GraPhlAn进化树。
本图中的文字标签,需要在树中存在对应的枝节点标签。
我们查看一下注释文件头
cd examples/HMP_tree
head annot.txt
看到此表头中一些新的参数
title Human Microbiome Project species tree
title_font_size 33
total_plotted_degrees 340
annotation_background_alpha 0.1
start_rotation 270
branch_bracket_width 0.0
class_legend_font_size 24
annotation_legend_font_size 24
annotation_background_separation -0.03
annotation_background_offset 0.0
annotation_background_width 0.07
branch_thickness 2.5
ring_label 1 Stool (prevalence)
ring_label_color 1 #0000FF
- title 图表标题
- title_font_size 标题字号
- total_plotted_degrees 图形面积,最大360占整个圆,小于360则形成开口扇形,此处340度留20度给环形作标题区域
- annotation_background_alpha 注释背景的透明度,即有颜色,又让文字显示清楚
- start_rotation 开始角度,可转动图形,调整开口僧
- class_legend_font_size 类图例文字大小
- annotation_legend_font_size 注释图例文字大小
- annotation_background_separation 注释背景分开
- annotation_background_offset 注释背景偏移量
- annotation_background_width 注释背景宽度
- ring_label 设置环形的标题
- ring_label_color 设置环形的标题颜色
肠道微生物组 Gut microbiome
特种分类树,比较HMP和MetaHIT的肠道微生物组。 特别是:颜色与生物标记进化枝的效应大小直接相关; 节点的大小反映了它们的相对丰度(参考文献 http://www.nature.com/nmeth/journal/v9/n8/full/nmeth.2066.html)。
下图是example文件夹中存在的gut_microbiome文件夹的参考。
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-20xy3xlM-1582818813975)(http://210.75.224.110/Note/LiuYongXin/200227graphlan/gut_microbiome.png)]
这是一个典型的LEfSe分析的默认物种树配色和样式,详见。
- LEfSe分析简介
- 简化美化LEfSe分析结果图
菌株水平分析 PhyloPhlAn
带有分类注释的高分辨率微生物生命树。 比例表示总分支长度的标准化分数。 灰色标记表示具有至少10个基因组的属的最低共同祖先。 外部条长度代表每个基因组中包含的400种蛋白质的分数。 红色的外部三角形表示我们的方法预测的基因组在分类学上被误贴标签并且可以放心地替换。 蓝色三角形表示有问题的标签已经过精致处理,但仍不属于完全一致的类别; 绿色三角形表示我们有信心完善了其不完整分类标签的基因组; 黑色三角形表示来自IMG-GEBA的566个基因组已被新放置到树中(参考文献 https://www.nature.com/articles/ncomms3304)。
下图数据在example文件夹中的PhyloPhlAn文件夹。
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-bMABbV6w-1582818813976)(http://210.75.224.110/Note/LiuYongXin/200227graphlan/ppa_tol.png)]
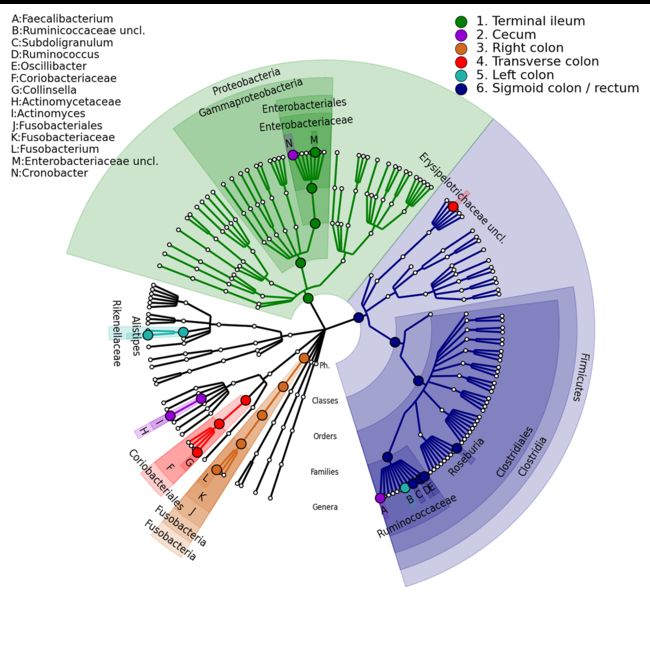
炎症性肠病生物地理 IBD biogeography
显示炎症性肠病生物地理学的分类树。 具体来说,以下身体部位具有不同的颜色:回肠末端,盲肠,右结肠,横结肠,左结肠和乙状结肠/直肠(参考 http://genomebiology.com/2012/13/9/R79)。
下图参考了example文件夹中的IBD_biogeography文件夹。
完整参数列表 Complete list of parameters
有关GraPhlAn提供的可能配置的详细列表,请查看软件解压文件夹中的readme.txt文件。
GraPhlAn与其他工具的集成
我们开发了一个名为export2graphlan.py的自动转换脚本,该脚本允许从LEfSe,MetaPhlAn2和HUMAnN输入和/或输出文件自动转换为GraPhlAn。 输入文件也可以BIOM 2.0格式给出。
该转换脚本的目的是支持生物学家,通过自动为GraPhlAn编写树和注释文件来帮助他们。 export2graphlan在GraPhlAn中作为子目录存在。 可以在此处找到一些说明如何使用它的示例:export2graphlan教程:http://210.75.224.110/Note/LiuYongXin/200227graphlan/export2graphlan%20-%20tutorial
支持 Support
我们通过Google网上论坛 https://groups.google.com/forum/#!forum/graphlan-users 为GraPhlAn用户提供支持,您也可以将任何问题通过电子邮件发送至 [email protected]。
英文版更新于 2018-06-02
中文版更新于 2020-02-27
附录
关于GraPhlAn的使用,请参阅我们之前的实战教程
- GraPhlAn进化树
- 物种树Cladogram
- LEfSe分析简介
- 简化美化LEfSe分析结果图
附录1. 颜色
颜色有三种设置方法
- 颜色英文名称
blue, green, red, cyan, magenta, yellow, black, white
- 单个字母的缩写
‘b’ (blue蓝), ‘g’ (green绿), ‘r’ (red红), ‘c’ (cyan青), ‘m’ (magenta洋红), ‘y’ (yellow黄), ‘k’ (black黑), ‘w’ (white白)
-
RGB模式颜色
#rrggbb, 例如 #FF0000 代表红色
附录2. 形状选择
- ‘.’ : 点 point marker
- ‘,’ : pixel marker
- ‘o’ : 圈 circle marker
- ‘v’ : 下三角 triangle_down marker
- ‘^’ : triangle_up marker
- ‘<’ : triangle_left marker
- ‘>’ : triangle_right marker
- ‘1’ : tri_down marker
- ‘2’ : tri_up marker
- ‘3’ : tri_left marker
- ‘4’ : tri_right marker
- ‘s’ : square marker
- ‘R’ : 矩阵 rectangle marker
- ‘p’ : pentagon marker
- ‘*’ : star marker
- ‘h’ : hexagon1 marker
- ‘H’ : hexagon2 marker
- ‘+’ : plus marker
- ‘x’ : x marker
- ‘D’ : diamond marker
- ‘d’ : thin_diamond marker
- ‘|’ : vline marker
- ‘_’ : hline marker
Reference
http://huttenhower.sph.harvard.edu/graphlan
Asnicar, Francesco, George Weingart, Timothy L. Tickle, Curtis Huttenhower, and Nicola Segata. 2015. ‘Compact graphical representation of phylogenetic data and metadata with GraPhlAn’, PeerJ, 3: e1029.
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA