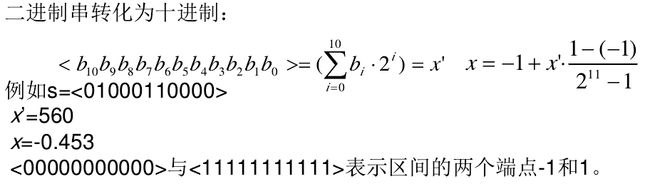遗传算法详解及Java实现
1. 遗传算法的起源
20世纪60年代中期,美国密西根大学的John Holland提出了位串编码技术,这种编码既适合于变异又适合杂交操作,并且他强调将杂交作为主要的遗传操作。遗传算法的通用编码技术及简单有效的遗传操作为其广泛的应用和成功奠定了基础。
2. 遗传算法的目的
解决经典数学方法无法有效地求出最优解的复杂的、大规模的难题。
3. 遗传算法的思想
遗传算法通常使用二进制编码来仿照基因编码,初代种群产生之后,按照适者生存和优胜劣汰的原理,逐代(generation)演化产生出越来越好的近似解,在每一代,根据问题域中个体的适应度(fitness)大小选择个体,并借助于自然遗传学的遗传算子(genetic operators)进行组合交叉(crossover)和变异(mutation),产生出代表新的解集的种群。
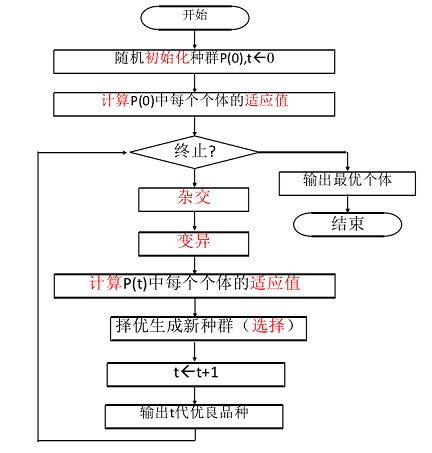
4. 遗传算法的步骤
(1) 用固定长度的染色体表示问题变量域,选择染色体种群数量为N,交叉概率为C,突变概率为M
(2) 定义适应性函数来衡量问题域上单个染色体的性能或适应性。适应性函数是在繁殖过程中选择配对染色体的基础。
(3) 随机产生一个大小为N的染色体的种群。
(4) 计算每个染色体的适应性。
(5) 在当前种群中选择一对染色体。双亲染色体被选择的概率和其适应性有关。适应性高的染色体被选中的概率高于适应性低的染色体。
(6) 通过执行遗传操作——交叉和突变产生一对后代染色体。
(7) 将后代染色体放入新种群中。
(8) 重复步骤5,直到新染色体种群的大小等于初始种群的大小N为止。
(9) 用新(后代)染色体种群取代初始(双亲)染色体种群。
(10) 回到步骤4,重复这个过程直到满足终止条件为止。
5. 算法思路:
(1)变量作为实数,可以视为演化算法的表现型形式。从表现型到基因型的映射称为编码。我们这里采用二进制编码,将某个变量值代表的个体表示为一个{0,1}二进制串。串长取决于求解的精度。
(2)用遗传算法解决函数优化问题,先随机产生0和1填充10个46位基因数字串来创建染色体的初始种群,再通过自然选择,交叉,变异等步骤得出下一代种群。
(3)迭代,再次执行步骤2直到满足需求或达到迭代次数。
6.遗传算法解决函数优化问题
(1)问题描述
(2)编码与解码
确定求解精度到6位小数,由于区间长度为6,必须将区间[0,6]分为 6 × 1 0 6 6× 10^6 6×106
等份。因为由 2 22 < 6 × 1 0 6 < 2 23 2^{22} <6×10^6 <2^{23} 222<6×106<223 得,使用23位的二进制数来表示变量,故单个染色体需46位基因(以表示x和y两个变量)
Java实现代码:
/**
* 将染色体转换成x,y变量的值
*/
private double[] calculatefitnessvalue(String str) {
//二进制数前23位为x的二进制字符串,后23位为y的二进制字符串
int a = Integer.parseInt(str.substring(0, 23), 2);
int b = Integer.parseInt(str.substring(23, 46), 2);
double x = a * (6.0 - 0) / (Math.pow(2, 23) - 1); //x的基因
double y = b * (6.0 - 0) / (Math.pow(2, 23) - 1); //y的基因
//需优化的函数
double fitness = 3 - Math.sin(2 * x) * Math.sin(2 * x)
- Math.sin(2 * y) * Math.sin(2 * y);
double[] returns = { x, y, fitness };
return returns;
}
(3)轮盘选择
基本思想:个体被选中的概率与其适应度值成正比,即按由个体适应度值所决定的某个规则选择将进入下一代的个体。(详细解析)
Java实现代码:
/**
* 轮盘选择
* 计算群体上每个个体的适应度值;
* 按由个体适应度值所决定的某个规则选择将进入下一代的个体;
*/
private void select() {
double evals[] = new double[ChrNum]; // 所有染色体适应值
double p[] = new double[ChrNum]; // 各染色体选择概率
double q[] = new double[ChrNum]; // 累计概率
double F = 0; // 累计适应值总和
for (int i = 0; i < ChrNum; i++) {
evals[i] = calculatefitnessvalue(ipop[i])[2];
if (evals[i] < bestfitness){ // 记录下种群中的最小值,即最优解
bestfitness = evals[i];
bestgenerations = generation;
beststr = ipop[i];
}
F = F + evals[i]; // 所有染色体适应值总和
}
//划分区间,打造选择所用的轮盘
for (int i = 0; i < ChrNum; i++) {
p[i] = evals[i] / F;
if (i == 0)
q[i] = p[i];
else {
q[i] = q[i - 1] + p[i];
}
}
for (int i = 0; i < ChrNum; i++) {
double r = Math.random();
if (r <= q[0]) {
ipop[i] = ipop[0];
} else {
for (int j = 1; j < ChrNum; j++) {
if (r < q[j]) {
ipop[i] = ipop[j];
break; //确定区间后跳出循环
}
}
}
}
}
(4)组合交叉(杂交)
单点杂交:设定杂交概率与杂交个体,按照断裂点进行染色体杂交
示例:
杂交前:a=<0101|0000>, b=<0111|1111>
杂交后:a=<0101|1111>, b=<0111|0000>
Java实现代码:
/**
* 交叉操作 交叉率为60%,平均为60%的染色体进行交叉
*/
private void cross() {
String temp1, temp2;
for (int i = 0; i < ChrNum; i++) {
if (Math.random() < 0.60) {
int pos = (int)(Math.random()*GENE)+1; //pos位点前后二进制串交叉
temp1 = ipop[i].substring(0, pos) + ipop[(i + 1) % ChrNum].substring(pos);
temp2 = ipop[(i + 1) % ChrNum].substring(0, pos) + ipop[i].substring(pos);
ipop[i] = temp1;
ipop[(i + 1) % ChrNum] = temp2;
}
}
}
(5)变异
基本思想:根据变异概率选择变异位点,将二进制位改变
Java实现代码:
/**
* 基因突变操作 1%基因变异
*/
private void mutation() {
for (int i = 0; i < 4; i++) {
int num = (int) (Math.random() * GENE * ChrNum + 1);
int chromosomeNum = (int) (num / GENE) + 1; // 染色体号
int mutationNum = num - (chromosomeNum - 1) * GENE; // 基因号
if (mutationNum == 0)
mutationNum = 1;
chromosomeNum = chromosomeNum - 1;
if (chromosomeNum >= ChrNum)
chromosomeNum = 9;
String temp;
String a; //记录变异位点变异后的编码
if (ipop[chromosomeNum].charAt(mutationNum - 1) == '0') { //当变异位点为0时
a = "1";
} else {
a = "0";
}
//当变异位点在首、中段和尾时的突变情况
if (mutationNum == 1) {
temp = a + ipop[chromosomeNum].substring(mutationNum);
} else {
if (mutationNum != GENE) {
temp = ipop[chromosomeNum].substring(0, mutationNum -1) + a
+ ipop[chromosomeNum].substring(mutationNum);
} else {
temp = ipop[chromosomeNum].substring(0, mutationNum - 1) + a;
}
}
//记录下变异后的染色体
ipop[chromosomeNum] = temp;
}
}
(6)运行结果
public static void main(String args[]) {
GA Tryer = new GA();
Tryer.ipop = Tryer.initPop(); //产生初始种群
String str = "";
//迭代次数
for (int i = 0; i < 100000; i++) {
Tryer.select();
Tryer.cross();
Tryer.mutation();
Tryer.generation = i;
}
double[] x = Tryer.calculatefitnessvalue(Tryer.beststr);
str = "最小值" + Tryer.bestfitness + '\n' + "第"
+ Tryer.bestgenerations + "个染色体:<" + Tryer.beststr + ">" + '\n'
+ "x=" + x[0] + '\n' + "y=" + x[1];
System.out.println(str);
}
完整代码:Github
参考资料:
- 《人工智能——智能系统指南》