PDB文件说明
转载自 科研路上的小曹
蛋白质数据库Protein Data Bank(PDB)是一个包含蛋白质、核酸等生物大分子的结构数据的数据库,网址是http://www.rcsb.org。PDB可以经由网络免费访问,是结构生物学研究中的重要资源。为了确保PDB资料的完备与权威,各个主要的科学杂志、基金组织会要求科学家将自己的研究成果提交给PDB。PDB数据库存储结构数据的文件是PDB文件,每一个蛋白质或核酸都对应着一个编号,即PDBID, 文件的扩展名为.pdb。PDB文件可以由各种3D结构显示软件打开,比如pymol,Swiss-PDB viewer,VMD等。PDB文件里面的信息是有严格的格式的。各行数据,如标识,原子名,原子序号,残基名称,残基序号等,不仅要按照严格的顺序书写,而且各项所占的空符串长度,及其所处的各行的位置都是严格规定。今天为大家介绍一下PDB文件中信息的格式。
PDB文件中的记录类型:
一.标题部分
HEADER: 分子类, 公布日期, ID号OBSLTE: 注明此ID号已废弃, 改用新ID号TITLE: 说明实验方法类型CAVEAT: 可能的错误警告COMPND: 化合物分子组成SOURCE: 化合物来源KEYWDS: 关键词EXPDTA: 测定结构所用的实验方法AUTHOR: 结构测定者REVDAT: 修订日期及相关内容SPRSDE: 已撤销或更改的相关记录JRNL: 发表坐标的期刊REMARKREMARK 1: 有关文献REMARK 2: 最大分辨率REMARK 3: 用到的程序和统计方法. 记述结构优化的方法和相关统计数据.REMARK 4-999: 其他信息
二. 一级结构
DBREF: 其他序列库的有关记录SEQADV: PDB与其他记录的出入SEQRES: 残基序列MODRES: 对标准残基的修饰
三. 杂因子
HET: 非标准残基HETATM: 非标准残基的名称HETSNY: 非标准残基的同义字FORMOL: 非标准残基的化学式
四. 二级结构
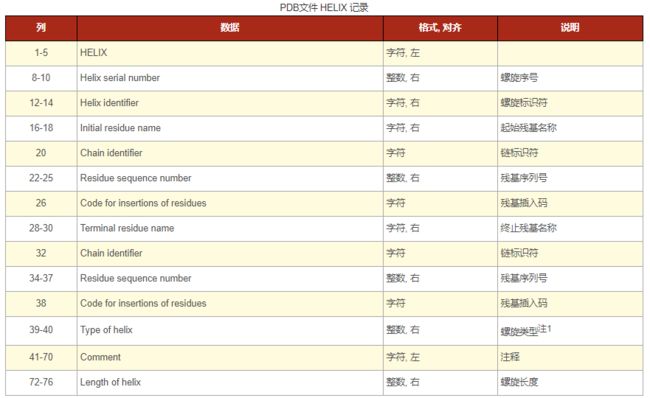
HELIX: 螺旋. 标识螺旋的位置和类型(右手α螺旋等), 每个螺旋一条记录.SHEET: 片层. 标识每个片层的位置, 类型(sense, 如反平行等), 相对于模型中每个束的片层(如果存在的话)中前一束的说明, 每个片层一条记录.TURN: 转角
五. 连接注释
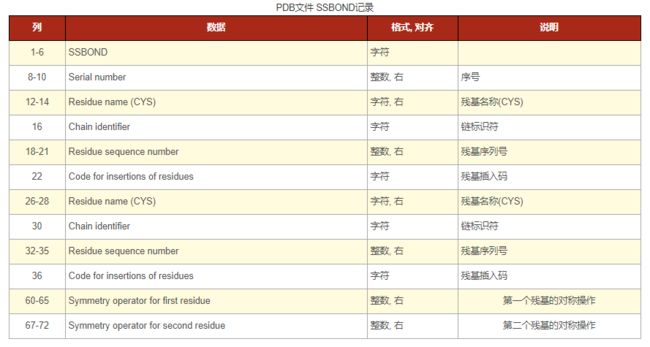
SSBOND: 二硫键. 定义半胱氨酸CYS残基之间的二硫键LINK: 残基间化学键HYDBND: 氢键SLTBRG: 盐桥CISPEP: 顺式残基
六. 晶胞特征及坐标变换
CRYST1: 晶胞参数(NMR除外). 记述晶胞结构参数(a, b, c, α, β, γ, 空间群)以及Z值(单位结构中的聚合链数).ORIGXn: 直角-PDB坐标SCALEn: 直角-晶体分数坐标(n=1, 2, 3, NMR除外). 说明数据中直角坐标向晶体分数坐标的变换因子.MTRIXn: 非晶相对称TVECT: 平移矢量
七. 坐标部分
MODEL: 多亚基时显示亚基号 当一个PDB文件中包含多个结构时(例: NMR结构解析), 该记录出现在各个模型的第一行.MODEL记录行的第11-14列上记入模型序号. 序号从1开始顺序记入, 在11-14列中从右起写. 比如说有30个模型, 则第1至9号模型, 该行的7-13列空白, 在14列上记入1-9的数字; 第10-30号模型, 该行的7-12列空白, 13-14列上记入10-30的数字.ATOM: 标准残基的原子. 记述标准残基(氨基酸以及核酸)中各原子的原子名称, 残基名称, 直角坐标(单位埃), 占有率, 温度因子等信息.SIGATM: 标准差ANISOU: 各向异性SIGUIJ: 各种温度因素导致的标准差TER: 残基链的末端. 表示残基链的结束. 在每个聚合链的末端都必须有TER记录, 但因序列无序造成的链中断处不需要该记录. 例如, 一个血红蛋白分子包含四个亚链. 彼此之间并不相连.TER标识了每条链的结束, 以防显示时这条链与下一条相连.HETATM: 非标准残基的原子. 记述非标准残基(标准氨基酸以及核酸以外的化合物, 包括抑制剂, 辅因子, 离子, 溶剂)中各原子的原子名称, 残基名称, 直角坐标(单位埃), 占有率, 温度因子等信息. 与ATOM记录的唯一区别在于HETATM残基默认情况下不会与其他残基相连. 注意, 水分子也应放在此记录中.ENDMDL: 亚基结束. 与MODEL记录成对出现, 记述在各模型的链末端的TER记录之后.
八. 连接信息部分
-
CONECT: 原子间的连接信息
九. 簿记
MASTER: 版权拥有者END: 文件结束. 标志PDB文件的结束, 必需记录.
一些记录类型的说明
PDB文件里面的每个记录都有着严格的格式. 每个记录中的字段, 如标识, 原子名称, 原子序号, 残基名称, 残基序号等, 不仅要按照严格的顺序书写, 而且每个字段所占的字符串长度, 及其所处的位置都是严格规定好的. 这些记录中, 通常最关心的是原子记录, 其详细说明可参考PDB原子记录官方文档.
一些老的PDB文件可能不完全遵循新格式. 对大多数用户而言, 最值得注意的区别在于ATOM和HETATM记录中的温度因子字段. 下文的例子中没有使用这些字段. 此外, 有些字段常常留空, 例如, 如当原子没有可替换位置时, 可替位置标识符就会留空.
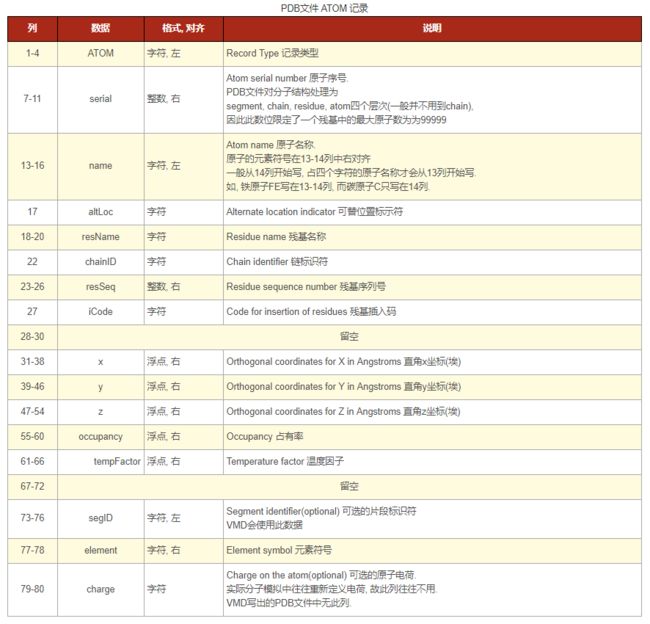
ATOM记录
HETATM记录
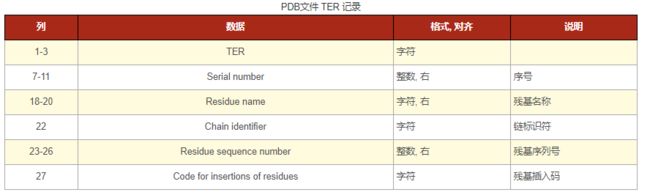
TER记录
SSBOND记录
HELIX记录
注1: 螺旋类型有如下几种:
1: Right-handed alpha (default) 右手α螺旋(默认)2: Right-handed omega 右手ω螺旋3: Right-handed pi 右手π螺旋4: Right-handed gamma 右手γ螺旋5: Right-handed 3/10 右手3/10螺旋7: Left-handed omega 右手ω螺旋6: Left-handed alpha 右手α螺旋8: Left-handed gamma 右手γ螺旋9: 2/7 ribbon/helix 2/7带状螺旋10: Polyproline 聚脯氨酸
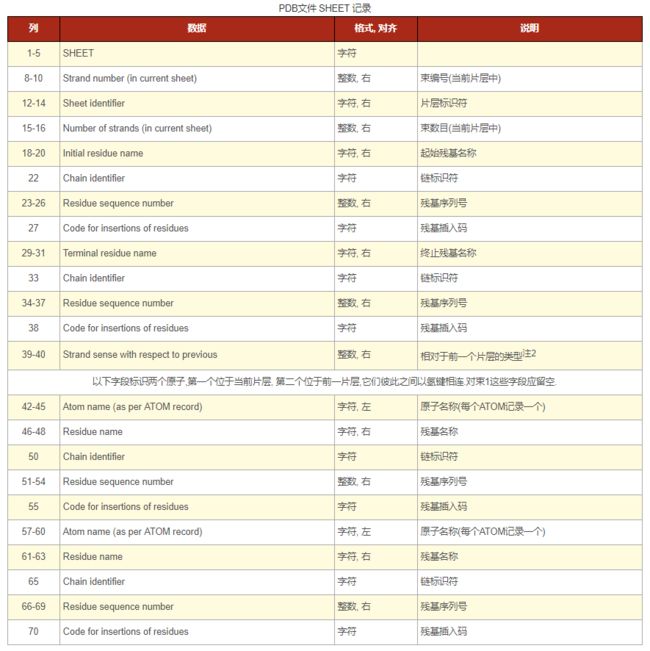
SHEET记录
注2: 类型标识:
1: 平行-1反平行0: 用于束1
格式说明
对于熟悉FORTRAN程序语言的用户, 下面是格式说明
ATOM或HETATM:Format ( A6,I5,1X,A4,A1,A3,1X,A1,I4,A1,3X,3F8.3,2F6.2,6X,A4,A2,A2 )SSBOND:Format ( A6,1X,I3,1X,A3,1X,A1,1X,I4,A1,3X,A3,1X,A1,1X,I4,A1,23X,2I3,1X,2I3 )HELIX:Format ( A6,1X,I3,1X,A3,2(1X,A3,1X,A1,1X,I4,A1),I2,A30,1X,I5 )SHEET:Format ( A6,1X,I3,1X,A3,I2,2(1X,A3,1X,A1,I4,A1),I2,2(1X,A4,A3,1X,A1,I4,A1) )
在FORTRAN语言的输入/输出格式中, X表示输入/输出空格; An表示输入/输的字符串占n位, 左对齐; In表示输入/输的整数占n位, 左对齐; Fm.n表示输入/输的浮点数占m位, 其中小数点后的数字占n位. 这些格式前面的整数则表示重复次数, 如23X表示23个空格, 3F8.3表示F8,3格式重复三次.
如果你使用其他程序语言, 可根据上面的格式说明转换为相应的形式.
PDB文件示例
单链蛋白
胰升血糖素(Glucagon)是一个小蛋白, 29个残基处于单条链中. 第一个残基是终端为氨的氨基酸HIS, 接着的是SER和GLU残基. 坐标部分开头如下:
">12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 1 N HIS 1 49.668 24.248 10.436 1.00 25.00
ATOM 2 CA HIS 1 50.197 25.578 10.784 1.00 16.00
ATOM 3 C HIS 1 49.169 26.701 10.917 1.00 16.00
ATOM 4 O HIS 1 48.241 26.524 11.749 1.00 16.00
ATOM 5 CB HIS 1 51.312 26.048 9.843 1.00 16.00
ATOM 6 CG HIS 1 50.958 26.068 8.340 1.00 16.00
ATOM 7 ND1 HIS 1 49.636 26.144 7.860 1.00 16.00
ATOM 8 CD2 HIS 1 51.797 26.043 7.286 1.00 16.00
ATOM 9 CE1 HIS 1 49.691 26.152 6.454 1.00 17.00
ATOM 10 NE2 HIS 1 51.046 26.090 6.098 1.00 17.00
ATOM 11 N SER 2 49.788 27.850 10.784 1.00 16.00
ATOM 12 CA SER 2 49.138 29.147 10.620 1.00 15.00
ATOM 13 C SER 2 47.713 29.006 10.110 1.00 15.00
ATOM 14 O SER 2 46.740 29.251 10.864 1.00 15.00
ATOM 15 CB SER 2 49.875 29.930 9.569 1.00 16.00
ATOM 16 OG SER 2 49.145 31.057 9.176 1.00 19.00
ATOM 17 N GLN 3 47.620 28.367 8.973 1.00 15.00
ATOM 18 CA GLN 3 46.287 28.193 8.308 1.00 14.00
ATOM 19 C GLN 3 45.406 27.172 8.963 1.00 14.00
注意到, 每一行(记录)都以记录类型ATOM开始, 记录中的下一项是原子序号.
原子名称是ATOM记录中的第三项, 它的前一或二个字符包含原子类型的元素符号. 所有以C开始的原子名称都代表碳原子, 同理, N代表氮原子, O代表氧原子. 原子名称的下一字符为远程标识符, 表示离氨基碳原子的远近, 含义如下
A: αB: βG: γD: δE: εZ: ζH: η
如果需要, 原子名称的最后一个字符可以代表分支标识符.
ATOM记录的下一数据字段为残基类型. 注意, 每一 记录都包含残基类型. 在上面的例子中, 链中的第一个残基为HIS, 第二个为SER.
ATOM记录的下一数据字段为残基的序列号. 注意到, 残基从HIS变为SER后, 残基序列号从1变为2. 两个相同的残基可能相邻, 因此残基编号对于区分它们非常重要.
ATOM记录的下三个数据字段分别为原子的X, Y, Z坐标. 后面接着的数据字段是占有率. 最后的数据字段是温度因子(也称B值).
胰升血糖素的PDB文件以这种方式继续下去, 直至最后一个残基
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 239 N THR 29 3.391 19.940 12.762 1.00 21.00
ATOM 240 CA THR 29 2.014 19.761 13.283 1.00 21.00
ATOM 241 C THR 29 .826 19.943 12.332 1.00 23.00
ATOM 242 O THR 29 .932 19.600 11.133 1.00 30.00
ATOM 243 CB THR 29 1.845 20.667 14.505 1.00 21.00
ATOM 244 OG1 THR 29 1.214 21.893 14.153 1.00 21.00
ATOM 245 CG2 THR 29 3.180 20.968 15.185 1.00 21.00
ATOM 246 OXT THR 29 -.317 20.109 12.824 1.00 25.00
TER 247 THR 29
注意, 这一残基包含额外的氧原子OXT, 它处于末端羰基上. TER记录终止了氨基酸链.
双链蛋白
更复杂的一个蛋白, 胎血红蛋白(fetal hemoglobin), 包含两条残基酸链(α和γ), 以及两个血红素基团. 这个蛋白坐标部分的前10行内容如下:
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 1 N VAL A 1 6.280 17.225 4.929 1.00 0.00
ATOM 2 CA VAL A 1 6.948 18.508 4.671 1.00 0.00
ATOM 3 C VAL A 1 8.436 18.338 4.977 1.00 0.00
ATOM 4 O VAL A 1 8.813 17.657 5.941 1.00 0.00
ATOM 5 CB VAL A 1 6.317 19.598 5.527 1.00 0.00
ATOM 6 CG1 VAL A 1 6.959 20.999 5.376 1.00 0.00
ATOM 7 CG2 VAL A 1 4.819 19.636 5.383 1.00 0.00
ATOM 8 N LEU A 2 9.259 18.958 4.152 1.00 0.00
ATOM 9 CA LEU A 2 10.715 18.872 4.330 1.00 0.00
ATOM 10 C LEU A 2 11.156 20.058 5.187 1.00 0.00
数据文件与上面胰升血糖素的基本一样, 除了第五个数据字段包含单个字符的链标识符A, 它标识血红蛋白分子的α链. 而在胰升血糖素的例子中, 这一字段为空. 在链A的终止处, 出现血红素基团的记录
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 1058 N ARG A 141 -6.576 12.834 -10.275 1.00 0.00
ATOM 1059 CA ARG A 141 -8.044 12.831 -10.214 1.00 0.00
ATOM 1060 C ARG A 141 -8.186 14.096 -9.365 1.00 0.00
ATOM 1061 O ARG A 141 -7.591 15.139 -9.671 1.00 0.00
ATOM 1062 CB ARG A 141 -8.579 11.531 -9.580 1.00 0.00
ATOM 1063 CG ARG A 141 -8.386 11.441 -8.054 1.00 0.00
ATOM 1064 CD ARG A 141 -8.727 10.045 -7.568 1.00 0.00
ATOM 1065 NE ARG A 141 -9.095 10.056 -6.143 1.00 0.00
ATOM 1066 CZ ARG A 141 -9.268 8.931 -5.414 1.00 0.00
ATOM 1067 NH1 ARG A 141 -8.602 8.795 -4.282 1.00 0.00
ATOM 1068 NH2 ARG A 141 -10.097 7.962 -5.830 1.00 0.00
ATOM 1069 OXT ARG A 141 -8.973 13.984 -8.310 1.00 0.00
TER 1070 ARG A 141
HETATM 1071 FE HEM A 1 8.133 8.321 -15.014 1.00 0.00
HETATM 1072 CHA HEM A 1 8.863 8.752 -18.417 1.00 0.00
HETATM 1073 CHB HEM A 1 10.362 10.946 -14.389 1.00 0.00
HETATM 1074 CHC HEM A 1 8.482 7.374 -11.743 1.00 0.00
HETATM 1075 CHD HEM A 1 6.982 5.180 -15.773 1.00 0.00
HETATM 1076 N A HEM A 1 9.452 9.545 -16.178 1.00 0.00
α链中最后一个残基为ARG, 额外的氧原子OXT同样出现在末端羰基基团中. TER记录标识了多肽链的结束. 在多肽链的结束处使用TER记录非常重要, 这样, 才不至于将一条链的终结处与另一条链的起始处相连.
上面的例子中, TER记录是正确的, 并且应该存在. 但是, 即便没有TER记录标识, 分子链仍然应该在某处终止, 因为HETATM残基不会与其他残基相连, 或互相相连. 作为单个残基的血红素基团由HETATM记录组成.
在α链血红素基团的结束处, γ链开始出现:
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
HETATM 1109 CAD HEM A 1 7.582 6.731 -20.480 1.00 0.00
HETATM 1110 CBD HEM A 1 8.992 6.848 -20.968 1.00 0.00
HETATM 1111 CGD HEM A 1 8.998 6.529 -22.465 1.00 0.00
HETATM 1112 O1D HEM A 1 9.693 5.683 -22.895 1.00 0.00
HETATM 1113 O2D HEM A 1 8.276 7.153 -23.229 1.00 0.00
ATOM 1114 C ACE G 0 7.896 -18.462 -1.908 1.00 0.00
ATOM 1115 O ACE G 0 7.246 -18.839 -.922 1.00 0.00
ATOM 1116 CH3 ACE G 0 9.415 -18.301 -1.832 1.00 0.00
ATOM 1117 N GLY G 1 7.354 -18.174 -3.077 1.00 0.00
ATOM 1118 CA GLY G 1 5.904 -18.282 -3.283 1.00 0.00
ATOM 1119 C GLY G 1 7.139 -19.112 -2.930 1.00 0.00
ATOM 1120 O GLY G 1 7.026 -20.248 -2.448 1.00 0.00
ATOM 1121 N HIS G 2 8.300 -18.533 -3.176 1.00 0.00
ATOM 1122 CA HIS G 2 9.565 -19.224 -2.889 1.00 0.00
这里, 新链的开始隐含着TER记录存在. 新链的标识符为G. 整个文件以与前面相同的模式继续下去, 到整条γ链及其血红素结束.
数据字段中的空格非常关键. 如果没有提供数据, 相应的字段应该留空. 例如, 仅包含单条氨基酸链的蛋白没有链标识符, 因此, 22列应该留空.
对于上面的例子, 看起来PDB格式依赖于 残基 的概念. 残基的规则总结如下:
所有处于单个残基内的原子都必须具有唯一的名称. 例如, 残基
VAL可能只有一个名称为CA的原子. 其他残基可能也含有CA原子, 但VAL中出现的CA不能超过一个.残基名称最大长度为三个字符, 并且能唯一地标识残基类型. 因此, 文件中具有给定名称的所有残基都具有相同的残基类型, 相同的结构. 每个特定残基在PDB文件中出现时都应具有相同的原子和连接性.
PDB格式文件中的常见错误
如果一个PDB文件无法正常展示, 在其成百上千行数据中找到错误位置有时很困难. 这里给出PDB文件中一些最常见的错误.
程序创建的PDB文件
虚假的超长键
由程序创建的PDB文件中, 常见的一种错误会导致在本来不该相连的残基间显示出非常长的键. 这种错误来自于缺少了分子链结束处的TER记录. 根据PDB标准, TER记录标识了分子链的结束. 文件中如果缺失了TER记录, 应该插入它们. 或者, 作为替代方法, 对每条链使用不同的链标识符.
显示超长键的第二个常见原因是不正确地使用ATOM记录, 而不使用HETATM记录. HETATM记录应该用于那些不形成链的化合物, 如水或血红素. 许多程序创建的PDB文件没有正确地使用HETATM记录. 在这种情况下, ATOM记录的开头 6 列应改为HETATM, 这样, 其余列的排列仍然正确.
未正确排列的原子名称
PDB记录中未正确排列的原子名称可能导致问题. ATOM和HETATM记录中的原子名称由下列内容组成: 元素符号(如C), 右 对齐在13-14列中; 远程标识字符(如A), 左 对齐在15-16列中. 许多程序只是简单地从第13列开始将整个原子名称左对齐. 在下面血红蛋白的一部分文件中可以清楚地看到区别:
正确的
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
HETATM 976 FE HEM 1 12.763 34.157 9.102 1.00 0.00
HETATM 977 CHA HEM 1 16.124 33.461 10.405 1.00 0.00
HETATM 978 CHB HEM 1 11.350 32.580 12.046 1.00 0.00
HETATM 979 CHC HEM 1 9.326 34.709 7.887 1.00 0.00
HETATM 980 CHD HEM 1 14.138 35.379 6.119 1.00 0.00
错误的
">12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
HETATM 976 FE HEM 1 12.763 34.157 9.102 1.00 0.00
HETATM 977 CHA HEM 1 16.124 33.461 10.405 1.00 0.00
HETATM 978 CHB HEM 1 11.350 32.580 12.046 1.00 0.00
HETATM 979 CHC HEM 1 9.326 34.709 7.887 1.00 0.00
HETATM 980 CHD HEM 1 14.138 35.379 6.119 1.00 0.00
手动创建的PDB文件
重复的原子名称
在手动创建的PDB文件中, 一个可能的编辑错误是, 对于一个给定残基中的所有原子没有指定唯一的名称. 在下面的例子中, 残基VAL中有两个原子具有名称CA.
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 1 N VAL A 1 6.280 17.225 4.929 1.00 0.00
ATOM 2 CA VAL A 1 6.948 18.508 4.671 1.00 0.00
ATOM 3 C VAL A 1 8.436 18.338 4.977 1.00 0.00
ATOM 4 O VAL A 1 8.813 17.657 5.941 1.00 0.00
ATOM 5 CA VAL A 1 6.317 19.598 5.527 1.00 0.00
ATOM 6 CG1 VAL A 1 6.959 20.999 5.376 1.00 0.00
ATOM 7 CG2 VAL A 1 4.819 19.636 5.383 1.00 0.00
ATOM 8 N LEU A 2 9.259 18.958 4.152 1.00 0.00
ATOM 9 CA LEU A 2 10.715 18.872 4.330 1.00 0.00
ATOM 10 C LEU A 2 11.156 20.058 5.187 1.00 0.00
取决于所用的可视化程序, 可能无法正确显示残基的连接, 或者只有当标记残基才会给出缺少CB原子的错误.
序列之外的残基
在下面的例子中, 出现于文件中的第二个残基(SER)被错误地编号为残基5. 许多可视化程序会显示残基5与残基1和3相连, 但只有当初确实需要这样时才正确. 如果残基5被假定出现在残基4和残基6之间, 它就应该出现在那里.
>12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 1 C HIS 1 49.169 26.701 10.917 1.00 16.00
ATOM 2 CA HIS 1 50.197 25.578 10.784 1.00 16.00
ATOM 3 CB HIS 1 51.312 26.048 9.843 1.00 16.00
ATOM 4 CD2 HIS 1 51.797 26.043 7.286 1.00 16.00
ATOM 5 CE1 HIS 1 49.691 26.152 6.454 1.00 17.00
ATOM 6 CG HIS 1 50.958 26.068 8.340 1.00 16.00
ATOM 7 N HIS 1 49.668 24.248 10.436 1.00 25.00
ATOM 8 ND1 HIS 1 49.636 26.144 7.860 1.00 16.00
ATOM 9 NE2 HIS 1 51.046 26.090 6.098 1.00 17.00
ATOM 10 O HIS 1 48.241 26.524 11.749 1.00 16.00
ATOM 11 C SER 5 47.713 29.006 10.110 1.00 15.00
ATOM 12 CA SER 5 49.138 29.147 10.620 1.00 15.00
ATOM 13 CB SER 5 49.875 29.930 9.569 1.00 16.00
ATOM 14 N SER 5 49.788 27.850 10.784 1.00 16.00
ATOM 15 O SER 5 46.740 29.251 10.864 1.00 15.00
ATOM 16 OG SER 5 49.145 31.057 9.176 1.00 19.00
ATOM 17 C GLN 3 45.406 27.172 8.963 1.00 14.00
ATOM 18 CA GLN 3 46.287 28.193 8.308 1.00 14.00
输入错误
有时字母l和数字1被互相替换了. 取决于这种错误在文件中出现的位置, 导致的问题也不一样. 错误放置的原子可能预示着错误出现在坐标字段中. 确定这种错误的一种方式是, 使用大写字母表示文件中的数据, 然后使用文本编辑器查找所有的小写字母l.
氢原子约定
PDB文件中的氢原子约定如下:
出现在
ATOM记录中的氢原子, 处于特定残基所有其他原子的后面.每个氢原子的名称根据与它相连原子的名称来确定: 名称的第一个位置(13列)为可选的数字, 当有两个或多个氢原子与同一个原子相连时才使用; 第二个位置(14列)为元素符号
H; 接下来的两列包含与氢原子相连原子的远程和分支标识符(1或2个字符).
示例如下
">12345678901234567890123456789012345678901234567890123456789012345678901234567890
----+----1----+----2----+----3----+----4----+----5----+----6----+----6----+----8
ATOM 1 N VAL 1 -13.090 1.966 9.741 1.00 0.00
ATOM 2 CA VAL 1 -12.852 3.121 8.892 1.00 0.00
ATOM 3 C VAL 1 -13.047 4.399 9.711 1.00 0.00
ATOM 4 O VAL 1 -12.143 5.228 9.800 1.00 0.00
ATOM 5 CB VAL 1 -13.753 3.058 7.658 1.00 0.00
ATOM 6 CG1 VAL 1 -13.930 4.446 7.036 1.00 0.00
ATOM 7 CG2 VAL 1 -13.208 2.063 6.631 1.00 0.00
ATOM 8 H VAL 1 -13.919 1.449 9.527 1.00 0.00
ATOM 9 HA VAL 1 -11.816 3.075 8.557 1.00 0.00
ATOM 10 HB VAL 1 -14.734 2.707 7.977 1.00 0.00
ATOM 11 1HG1 VAL 1 -13.951 4.357 5.950 1.00 0.00
ATOM 12 2HG1 VAL 1 -14.866 4.883 7.384 1.00 0.00
ATOM 13 3HG1 VAL 1 -13.098 5.085 7.333 1.00 0.00
ATOM 14 1HG2 VAL 1 -12.623 1.298 7.142 1.00 0.00
ATOM 15 2HG2 VAL 1 -14.039 1.594 6.104 1.00 0.00
ATOM 16 3HG2 VAL 1 -12.575 2.588 5.917 1.00 0.00
在上面的例子中
所有氢原子都出现在残基的其他原子之后
9号原子
HA与2号原子CA相连. 这两个原子的远程标识符A相同.有三个氢原子与
CG1相连. 它们具有相同的远程标识符, 分支标识符, 但13列中含有区分数字, 因此每个氢原子都具有唯一的名称.当只有一个氢原子与给定原子相连时, 不需要使用数字作为氢原子名称的前缀.
氨基酸残基与核酸缩写
一些概念说明
温度因子 B-factor
The B-factor (or temperature factor) is an indicator of thermal motion about an atom. However, it should be pointed out that the B-factor is a mix of real thermal displacement, static disorder (multiple but defined conformations) and dynamic disorder (no defined conformation), and all the overlap between these definitions.
B因子也叫温度因子, 一般在晶体测定的pdb中都有, 是晶体学中的一个重要参数. 晶体学中结构因子可以表达为坐标x, y, z与Bj因子的函数. 物理学上对于Bj的表征有很多理论模型, 最成功的是由Debye和Waller提出的. 将固体内振荡的量子本质计算在内后, 他们将Bj表征为绝对温度T和其他各基本参数的函数. 由此可见, Bj与原子的质量等基本性质有关, 也与实验温度有关.
B因子体现了晶体中原子电子密度的”模糊度”(diffusion), 这个”模糊度”实际上反映了蛋白质分子在晶体中的构象状态. B因子越高, “模糊度”越大, 相应部位的构象就越不稳定. 在晶体学数据中, B因子一般是以原子为单位给出的, 我们可以换算成相应残基的B因子, 从而分析残基的构象稳定性. 另外, 计算出的B因子中实际上包含了实验中的很多因素, 如晶体结构测定的实验误差等, 精度高的晶体结构数据提供较可靠的B因子数据.
此外, 另外温度因子还和占有率相关, 如果本身结构解析过程中占有率低, 也会导致温度因子升高. 这个时候只能说是X-ray收集数据的时候这个地方的信号比较弱, 而和结构本身的构象如何, 没有关系.
PDB中的晶体学数据是以原子为单位的, 它所给出的B因子是相对于每个原子的. 统计中, 首先将原子的B因子换算成残基的B因子, 即把每个残基所有原子的B因子取平均值. 由于蛋白质分子表面残基的运动性比较大, B因子相对较高, 所以在统计中除去了这部分残基, 具体方法是将数据中B因子高的残基去掉10%, 对剩下的残基进行统计, 计算平均值.
温度因子做图后可以体现蛋白某些部位的活动性和柔韧性. 它也可以由计算rmsf得到. 在GROMACS中, g_rmsf可以将rmsf换算成B因子输出至pdb. 与晶体测定结构中的B因子相比较, 如果呈较好的相关, 可以说明模拟的过程是正常, 合理的. 但pdb中的B因子都是原子的, 一般是比较残基间的, 可以转换一下.
R-factor
In overview, the R-factor is a measure of how well a particular model structure fits the observed electron density. Or simply, “a measure of agreement between the crystallographic model and the original X-ray diffraction data”.
参考资料
PDB文件的格式
PDB文件详解
有关原子坐标文件
WOLFRAM 语言 IMPORT/EXPORT 格式 PDB
教你读懂蛋白的PDB文件
PDB 文件格式
什么叫HETATM
温度因子(B因子)专题
Introduction to Protein Data Bank Format
Biopython PDB模块
来源:https://jerkwin.github.io/2015/06/05/PDB%E6%96%87%E4%BB%B6%E6%A0%BC%E5%BC%8F%E8%AF%B4%E6%98%8E/
本文来自陈泽庸科学网博客。
链接地址:http://blog.sciencenet.cn/blog-3196388-1110691.html