一、数据准备
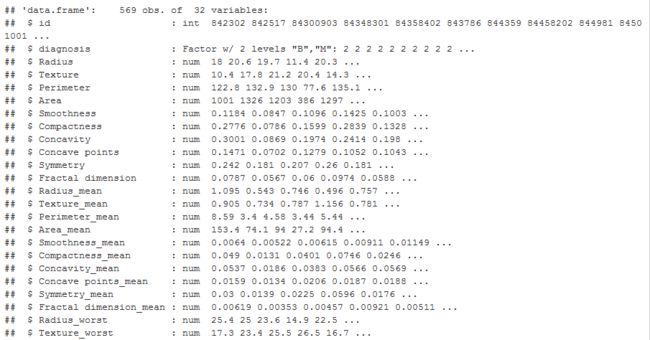
乳腺癌数据包括569例细胞活检案例,每个案例有32个特征。
第一个特征是识别号码,
第二个特征是癌症诊断结果(癌症诊断结果用编码“M”表示恶性,用编码“B”表示良性),
其他30个特征是数值型的实验室测量结果。(其他30个数值型测量结果由数字化细胞核的10个不同特征的均值、标准差和最差值(即最大值)构成)。这些特征包括:
Radius(半径)、Texture(质地)、Perimeter(周长)、Area(面积)、Smoothness(光滑度)、Compactness(致密性)、Concavity(凹度)、Concave points(凹点)、Symmetry(对称性)、Fractal dimension(分形维数)
url<-'http://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/wdbc.data'
#原数据的列名为v1,v2,不好理解,因此header=F
wdbc<-read.csv(url,header = F)
二、数据预处理
#根据数据描述,对每一列重命名
wdbc.name<-c("Radius","Texture","Perimeter","Area","Smoothness","Compactness","Concavity","Concave points","Symmetry","Fractal dimension")
wdbc.name<-c(wdbc.name,paste(wdbc.name,"_mean",sep=""),paste(wdbc.name,"_worst",sep=""))
names(wdbc)<-c("id","diagnosis",wdbc.name)
最终的数据为:
str(wdbc)
attach(wdbc)
table(diagnosis)
可以得到有357个为良性,有212个为恶性 因为id列没有意义,去掉id列
wdbc<-wdbc[-1]
并将目标属性编码因子化B良性M恶性
diagnosis<-factor(diagnosis,levels=c("B","M"),labels=c("Benign","Malignant"))
diagnosis
并计算各自占比
round四舍五入round(x,digits=n)
prop.table得到边缘概率prop.table(x,margin=null)
round(prop.table(table(diagnosis))*100,digits=1)
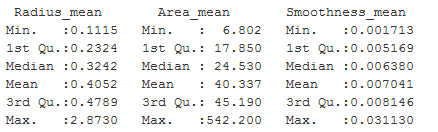
通过summary详细地观察3个特征:可以看出不同特征的度量值差别大
summary(wdbc[c("Radius_mean","Area_mean","Smoothness_mean")])
kNN的距离计算在很大程度上依赖于输入特征的测量尺度。由于光滑度的范围是0.05~0.16,且面积的范围是143.5~2501.0,所以在距离计算中,面积的影响比光滑度的影响大很多,这可能潜在地导致我们的分类器出现问题,所以我们应用min-max标准化方法将特征值重新调整到一个标准范围内,对数据通过归一化来进行无量纲处理
即显然数据需要转换,转换函数为:
normalize<-function(x){
return((x-min(x))/(max(x)-min(x)))
}
我们并不需要对这30个数值变量逐个进行min-max标准化,这里可以使用R中的一个函数来自动完成此过程
lapply()函数接受一个列表作为输入参数,然后把一个具体函数应用到每一个列表元素。因为数据框是一个含有等长度向量的列表,所以我们可以使用lapply()函数将normalize()函数应用到数据框中的每一个特征。最后一个步骤是,应用函数as.data.frame()把lapply()返回的列表转换成一个数据框。过程如下所示:
wdbc_n<-as.data.frame(lapply(wdbc[2:31],normalize))
这里使用的后缀_n是一个提示,即wdbc中的值已经被min-max标准化了。
为了确认转换是否正确应用,让我们来看看其中一个变量的汇总统计量:
summary(Area_mean)
正如预期的那样,area_mean变量的原始范围是143.5~2501.0,而现在的范围是0~1。
三、切分数据集
接下来需要切分数据集,实际需要构造training、validation、test,其中validation用来校正提高模型准确性,为简单起见,我们只用train和test
第一种方法:由于我们没有新病人的数据,所以使用前469条记录作为训练数据集,剩下的100条记录用来模拟新的病人
wdbc_train<-wdbc_n[1:469,]
wdbc_test<-wdbc_n[470:569,]
#存储目标变量标签
wdbc_train_labels<-wdbc[1:469,1]
wdbc_test_labels<-wdbc[470:569,1]
mal_rate<-table(wdbc_train_labels)
round(mal_rate[2]/sum(mal_rate),digits=2)
第二种方法:如果样本中的恶性肿瘤大部分分布在1,则将469作为训练集就有很大问题,此时采用随机取样
set.seed(1234)
ratio<-sample(1:dim(wdbc_n)[1],469,replace=F)
wdbc_train<-wdbc_n[ratio,]
wdbc_test<-wdbc_n[-ratio,]
wdbc_train_labels<-wdbc[ratio,1]
wdbc_test_labels<-wdbc[-ratio,1]
mal_rate<-table(wdbc_train_labels)
round(mal_rate[2]/sum(mal_rate),digits=2)
第三种方法:直接利用"caret"包中的crateDataPartition函数可自动分区
library(caret)
set.seed(1234)
ratio<-createDataPartition(y=diagnosis,p=0.8,list=FALSE)
wdbc_train<-wdbc_n[ratio,]
wdbc_test<-wdbc_n[-ratio,]
wdbc_train_labels<-wdbc[ratio,1]
wdbc_test_labels<-wdbc[-ratio,1]
mal_rate<-table(wdbc_train_labels)
round(mal_rate[2]/sum(mal_rate),digits=2)
四、构建模型
构建模型,class包中的knn函数,由于训练数据集含有469个实例,所以我们可能尝试k = 21,它是一个大约等于469的平方根的奇数。根据二分类的结果,使用奇数将消除各个类票数相等这一情况发生的可能性。
函数knn()返回一个因子向量,为测试数据集中的每一个案例返回一个预测标签,我们将该因子向量命名为wdbc_test_pred。
现在,我们可以使用knn()函数对测试数据进行分类:
library(class)
wdbc_test_pred<-knn(train=wdbc_train,test=wdbc_test,cl=wdbc_train_labels,k=21)
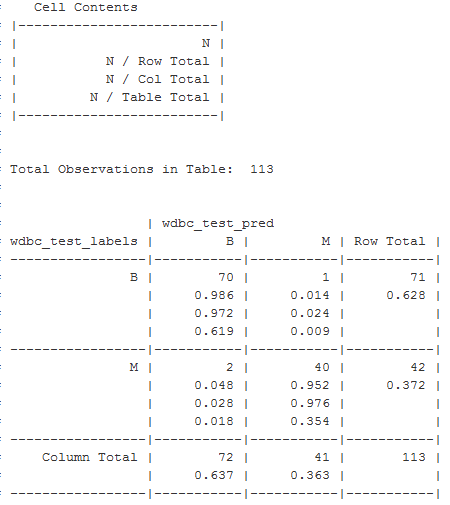
该过程的下一步就是评估wdbc_test_pred向量中预测的分类与wdbc_test_labels向量中已知值的匹配程度如何。为了做到这一点,我们可以使用gmodels添加包中的CrossTable()函数,它在第2章中介绍过。如果你还没有安装该添加包,可以使用install.packages("gmodels")命令进行安装。
在使用library(gmodels)命令载入该添加包后,可以创建一个用来标识两个向量之间一致性的交叉表。指定参数prop.chisq = FALSE,将从输出中去除不需要的卡方(chi-square)值,如下所示:
library(gmodels)
CrossTable(x=wdbc_test_labels,y=wdbc_test_pred,prop.chisq=FALSE)
行为真实结果,列为预测结果,对角框的数字越小,模型越好
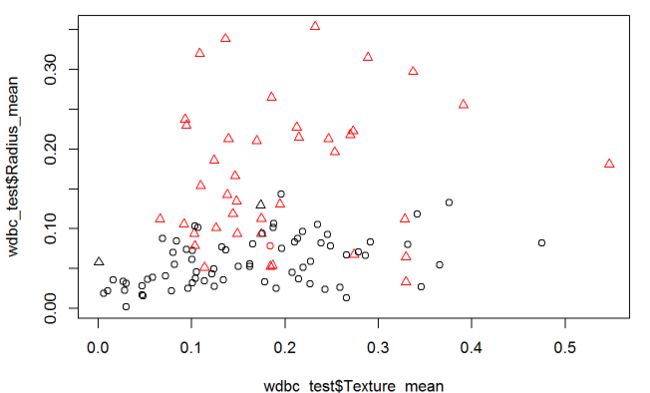
选取两个变量作为横纵坐标进行画图,观察实际类别与预测的分类结果。
plot(wdbc_test$Texture_mean,wdbc_test$Radius_mean,col=wdbc_test_pred,pch=as.integer(wdbc_test_labels))
颜色代表分类后得到的结果,形状代表真实的类别。从检测结果和图上都可以看出,分类结果基本与真实结果一致。