Milvus 赋能 AI 药物研发
新药研发领域长期以来都以耗时长、成本高、风险大、回报率低而著称,一款新药的平均研发成本已经高达 26 亿美元,而平均耗时需要十年。尽管付出了如此高昂的研发成本和漫长的研发周期,却依然无法保证所研发的药物能够顺利通过全部临床实验而投放市场。即便是难度较低的仿制药研发,其研发的进程也是十分缓慢。
伴随深度学习等一系列 AI 技术的不断发展,将 AI 技术与药物研发相结合,可以很大程度上减少新药研发时间、降低新药研发成本,也可以加速仿制药的研发和入市,毫无疑问人工智能和机器学习将开创一个更快速、更低价、更高效的药物研发时代。
Zilliz 公司联手全球顶尖制药研发企业共同开发了 MolSearch 化合物分子结构分析软件,为 AI 药物研发探索出了一个新的技术突破点。
| Milvus 向量搜索引擎
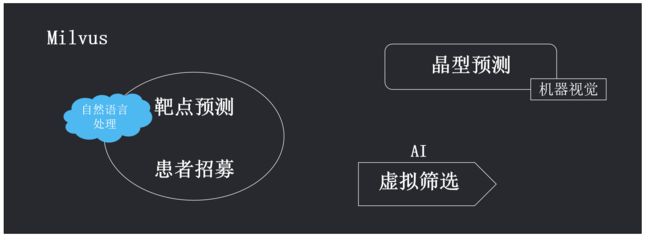
Milvus 作为一款开源的特征向量相似度搜索引擎,凭借其强大的非结构化数据处理能力已经广泛应用于 AI 技术的重点领域如:机器视觉(图片视频处理)、自然语言处理、语音识别等。
随着 AI 技术与药物研发领域的深度结合,Milvus 在这一领域也有着广阔的应用前景。 比如新药研发过程药物晶型预测可以结合 Milvus 在图像识别的应用,有效的预测出合适的药物晶型;靶点筛选和患者招募过程可以抽象为对文本语义分析问题,可以结合 Milvus 在自然语言处理的应用,快速分析有关药物研发的文本数据等。
虚拟药物筛选是新药研发过程中的一个关键步骤,通过模拟药物筛选的过程,预测化合物可能的活性,对比较有可能成为药物的化合物进行针对性的实体筛选,极大的降低了药物研发的成本。这过程在传统的方案受限于算法和算力,在对千万级别的化合物分子进行相似性、子结构、超结构等分析时,耗时在分钟级别,而集成了 Milvus 的方案能够对十亿级的化学式数据进行秒级分析,这一技术突破能够极大的提升新药研发的效率。
Milvus 能够广泛的应用在药物研发的各个阶段,通过将成熟的 AI 模型结合 Milvus 向量搜索引擎,一定会为药物研发领域带来更多颠覆性的技术突破。
| MolSearch 虚拟药物筛选工具
MolSearch 是基于 Milvus 向量相似度检索引擎研发的一款开源化合物分析软件,前端设计参考了开源软件 MolView[1],具体搭建和功能介绍可以参考 https://github.com/zilliztech/MolSearch。
药物化学专家通常根据骨架跃迁对分子模块进行优化,在此基础上设计出新药结构供后续筛选。我们共同研发 MolSearch 的初衷,是为了加速对海量化合物的虚拟筛选。虚拟筛选是新药研发中必不可少也是十分重要的一步,它的结果很大程度决定了后期小白鼠实验以及临床试验能否成功,虚拟筛选时化合物底库数量越多,筛选的准确率越高,那么新药研发成功的可能性也就越高。
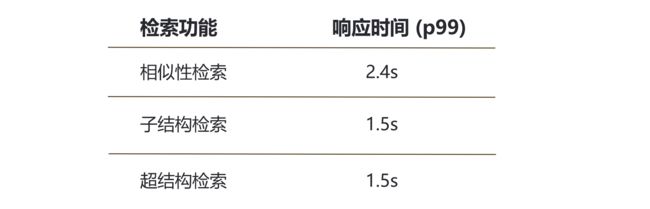
MolSearch 系统中分子检索功能通过集成 Milvus 作为核心检索引擎,实现了十亿级的化学分子结构进行秒级分析的能力,这也是 MolSearch 在药物研发领域的一个重要技术突破。目前在 MolSearch 中集成了8.2亿 zinc 开放化学式分子式数据集[2],通过将化学式转换为 2048 位的化学指纹(特征向量)在 Milvus 中进行高性能向量计算而实现对分子结构的相似性、子结构和超结构检索,其端到端的检索性能如下:
表中的 p 表示百分比,响应时间(p99)表示99%的检索能在多少时间完成。
| 系统概览
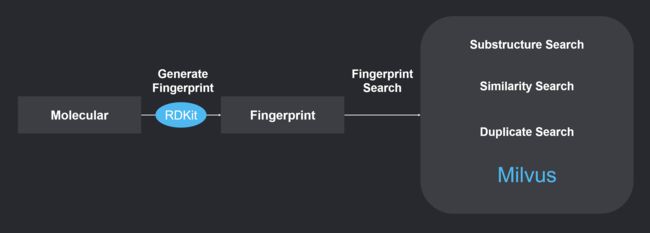
MolSearch 系统中运用的虚拟化合物筛选技术,首先通过 RDKit 工具[3]将化合物分子的化学式转换为化学式指纹 (Chemical Fingerprint),就是一组特征向量,然后通过计算这些向量之间的距离来分析化合物分子之间的相似性。
1. 化学指纹生成
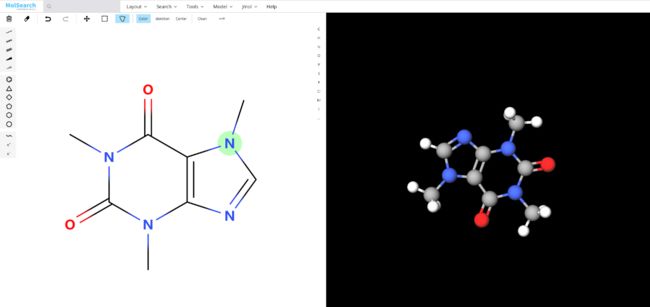
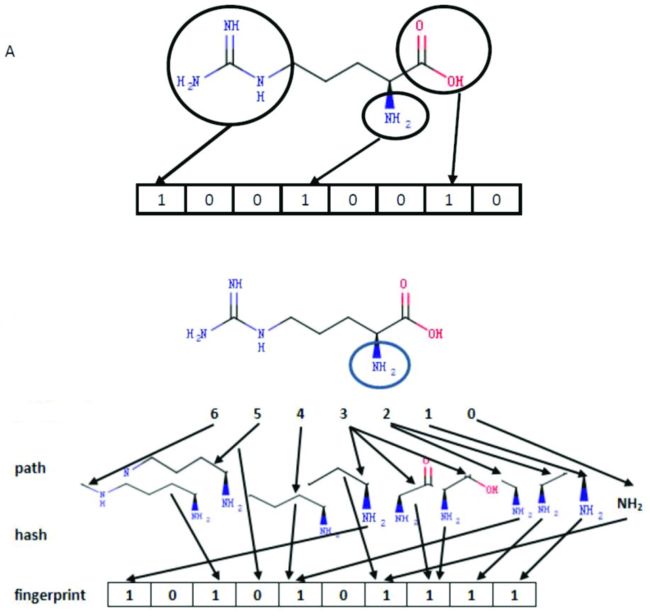
化学指纹通常用来做结构检索和相似度检索,如下图所示,指纹是 (1/0) 位表示的有序列表,每一位代表化学结构中例如指定元素,分子片段等的存在。
MolSearch 系统中利用 RDKit 工具生成 RDKit fingerprint,该算法分析从一个原子开始直至到达指定数量键的路径(path,通常为线性)上所有的分子片段,然后对每一个路径进行哈希(hash)产生指纹(fingerprint),如下图中展示了从NH2(已圈出)开始一直到 6 个长度的所有路径,然后将每个路径散列为二进制位。
上图仅展示了从一个单个起始原子出发的片段和比特位,对于完整的指纹,将对分子中的每个原子进行重复该过程。此类指纹适用于任意一个分子,并可以指定 fpSize 调整其向量维度,最终生成的 vectors 可以导入 Milvus 并检索。
fromrdkitimportChemmols=Chem.MolFromSmiles(smiles)fp=Chem.RDKFingerprint(mols,fpSize=VECTOR_DIMENSION)bit_fp=DataStructs.BitVectToFPSText(fp)vectors=bytes.fromhex(hex_fp)
2. 化合物检索
通过将生成的向量导入 Milvus 建立化合物库,根据不同的计算方式可以实现对化合物的相似度检索、子结构检索和超结构检索。
from milvus import *milvus = Milvus()milvus.insert(collection_name=MILVUS_TABLE, records=vectors)milvus.search(collection_name=MILVUS_TABLE, query_records=query_list, top_k=topk, params={})
-
相似度检索
用于寻找与输入的参考分子比较相似的分子。
-
子结构检索
检测一个分子结构是否为另一个分子的子结构。
-
超结构检索
检测一个分子结构是否为另一个分子的超结构。
3. 化学指纹计算
Milvus 支持各种常用的相似度计算指标,包括欧氏距离、内积、汉明距离和杰卡德距离等。针对二值型数据 MolSearch 系统选择 Jaccard/Substructure/Superstructure 距离计算相似度。
根据以上参数,化学式指纹计算可以描述为:
| 总结
Milvus 凭借其先进的软、硬件算法,能够为各类 AI 应用提供企业级的稳定、高性能向量检索支持。MolSearch 系统正式充分发挥了 Milvus 这一特性,实现了对海量分子式的高性能分析能力,颠覆了传统的虚拟药物筛选方案,实现了技术突破。我们相信 Milvus 必将会在药物研发的其他各个领域获得更广阔的应用前景,期待与 AI 药物研发领域的有志同仁携手共建 Milvus 这一 AI 数据处理平台。
| 引用
-
http://molview.org/
-
Sterling and Irwin, J. Chem. Inf. Model, 2015, https://pubs.acs.org/doi/abs/10.1021/acs.jcim.5b00559
-
Landrum, G. 2010. “RDKit.” Q2. https://www.rdkit.org/
| 欢迎加入 Milvus 社区
github.com/milvus-io/milvus | 源码
milvus.io | 官网
milvusio.slack.com | Slack 社区
zhihu.com/org/zilliz-11/columns | 知乎
zilliz.blog.csdn.net | CSDN 博客
space.bilibili.com/478166626 | Bilibili
![]()