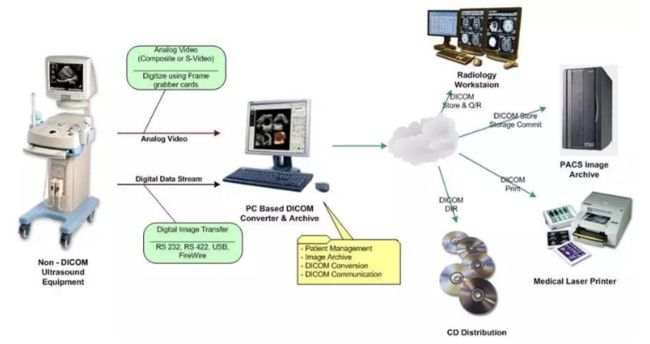
DICOM(Digital Imaging and Communications in Medicine)即医学数字成像和通信,是医学图像和相关信息的国际标准(ISO 12052)。它定义了质量能满足临床需要的可用于数据交换的医学图像格式,可用于处理、存储、打印和传输医学影像信息。DICOM可以便捷地交换于两个满足DICOM格式协议的工作站之间。目前该协议标准不仅广泛应用于大型医院,而且已成为小型诊所和牙科诊所医生办公室的标准影像阅读格式。
DICOM被广泛应用于放射医疗、心血管成像以及放射诊疗诊断设备(X射线,CT,核磁共振,超声等),并且在眼科和牙科等其它医学领域得到越来越深入广泛的应用。在数以万计的在用医学成像设备中,DICOM是部署最为广泛的医疗信息标准之一。当前大约有百亿级符合DICOM标准的医学图像用于临床使用。
目前,越来越多的DICOM应用程序和分析软件被运用于临床医学,促使越来越多的编程语言开发出支持DICOM API的框架。今天就让我来介绍一下Python语言下支持的DICOM模块,以及如何完成基本DICOM信息分析和处理的编程方法。
[Pydicom]
Pydicom是一个处理DICOM文件的纯Python软件包。它可以通过非常容易的“Pythonic”的方式来提取和修改DICOM数据,修改后的数据还会借此生成新的DICOM文件。作为一个纯Python包,Pydicom可以在Python解释器下任何平台运行,除了必须预先安装Numpy模块外,几乎无需其它任何配置要求。其局限性之一是无法处理压缩像素图像(如JPEG),其次是无法处理分帧动画图像(如造影电影)。
[SimpleITK]
Insight Segmentation and Registration Toolkit (ITK)是一个开源、跨平台的框架,可以提供给开发者增强功能的图像分析和处理套件。其中最为著名的就是SimpleITK,是一个简化版的、构建于ITK最顶层的模块。SimpleITK旨在易化图像处理流程和方法。
[PIL]
Python Image Library (PIL) 是在Python环境下不可缺少的图像处理模块,支持多种格式,并提供强大的图形与图像处理功能,而且API却非常简单易用。
[OpenCV]
OpenCV的全称是:Open Source Computer Vision Library。OpenCV是一个基于BSD许可(开源)发行的跨平台计算机视觉库,可以运行在Linux、Windows和Mac OS操作系统上。它轻量级而且高效——由一系列 C 函数和少量 C++ 类构成,同时提供了Python、Ruby、MATLAB等语言的接口,实现了图像处理和计算机视觉方面的很多通用算法。
下面就让我以实际Python代码来演示如何编程处理心血管冠脉造影DICOM图像信息。
1. 导入主要框架:SimpleITK、pydicom、PIL、cv2和numpy
import SimpleITK as sitk
from PIL import Image
import dicom
import numpy as np
import cv2
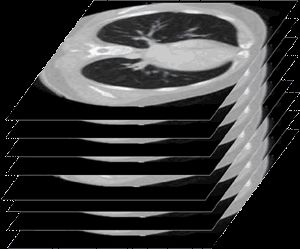
2. 应用SimpleITK框架来读取DICOM文件的矩阵信息。如果DICOM图像是三维螺旋CT图像,则帧参数则代表CT扫描层数;而如果是造影动态电影图像,则帧参数就是15帧/秒的电影图像帧数。
def loadFile(filename):
ds = sitk.ReadImage(filename)
img_array = sitk.GetArrayFromImage(ds)
frame_num, width, height = img_array.shape
return img_array, frame_num, width, height
3. 应用pydicom来提取患者信息。
def loadFileInformation(filename):
information = {}
ds = dicom.read_file(filename)
information['PatientID'] = ds.PatientID
information['PatientName'] = ds.PatientName
information['PatientBirthDate'] = ds.PatientBirthDate
information['PatientSex'] = ds.PatientSex
information['StudyID'] = ds.StudyID
information['StudyDate'] = ds.StudyDate
information['StudyTime'] = ds.StudyTime
information['InstitutionName'] = ds.InstitutionName
information['Manufacturer'] = ds.Manufacturer
information['NumberOfFrames'] = ds.NumberOfFrames
return information
4. 应用PIL来检查图像是否被提取。
def showImage(img_array, frame_num = 0):
img_bitmap = Image.fromarray(img_array[frame_num])
return img_bitmap
5. 采用CLAHE (Contrast Limited Adaptive Histogram Equalization)技术来优化图像。
def limitedEqualize(img_array, limit = 4.0):
img_array_list = []
for img in img_array:
clahe = cv2.createCLAHE(clipLimit = limit, tileGridSize = (8,8))
img_array_list.append(clahe.apply(img))
img_array_limited_equalized = np.array(img_array_list)
return img_array_limited_equalized
这一步对于整个图像处理起到很重要的作用,可以根据不同的原始DICOM图像的窗位和窗宽来进行动态调整,以达到最佳的识别效果。
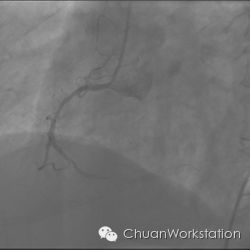
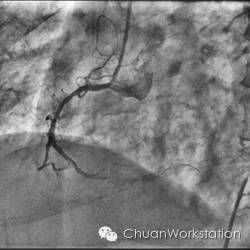
原始图像:
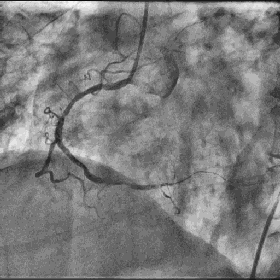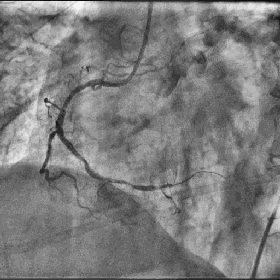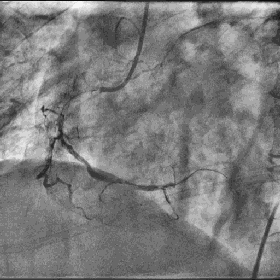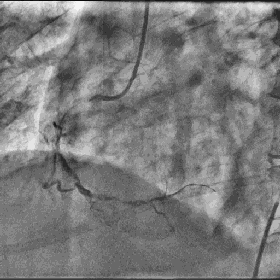
经过自动窗位窗宽调节,生成:
最后应用OpenCV的Python框架cv2把每帧图像组合在一起,生成通用视频格式。
def writeVideo(img_array):
frame_num, width, height = img_array.shape
filename_output = filename.split('.')[0] + '.avi'
video = cv2.VideoWriter(filename_output, -1, 16, (width, height))
for img in img_array:
video.write(img)
video.release()
参考:http://www.wtoutiao.com/p/14fY4tu.html
(转载请注明出处!)