Cell Host综述-建立因果关系:合成菌群在植物菌群研究中的机会
本文“宏基因组”公众号原创。编译作者:小钢蛋,编辑:metagenome
Establishing Causality: Opportunities of Synthetic Communities for Plant Microbiome Research
建立因果关系:合成菌群在植物菌群研究中的机会
Cell Host and Microbe [IF:17.872] 2017-08-09 Perspective
DOI: https://doi.org/10.1016/j.chom.2017.07.004
第一作者:Julia A Vorholt;
通讯作者:Julia A Vorholt;
其它作者:Christine Vogel,Charlotte I Carlström,Daniel B Müller
热心肠日报
Cell子刊:合成菌群在植物菌群研究中的机会
① 讨论解决菌群原位互作的遗传复杂性的还原论方法,其目标是解析参与菌群组装、动力学、快速恢复/耐受、建立菌群与植物表型之间的因果关系的生物学机制;
② 利用自下而上的方法选定构成合成菌群的混合菌株,在实验上易于操作,可检验无菌系统的靶向操作的假说,产生可转化为农业应用的知识;
③ 菌群、宿主和环境参数的修饰,可动态、空间维度上定量评估宿主和细菌的特征。
主编评语:这是Cell Host & Microbe发出的关于如何利用混合不同菌株的“合成菌群”以促进植物菌群研究的观点文章,立足于植物,但对其他菌群,包括人类肠道菌群的研究也有启发意义。
摘要
近年来,随着植物微生物组学的研究更加深入,更加强调了微生物群落对植物生长以及健康等方面的重要作用。植物微生物组学的研究目的在于发现植物与微生物之间,微生物与微生物之间互作的分子机制,从而更加深入理解微生态系统乃至整个生态系统的功能。在这篇综述中,作者讨论了微生物在原生态位相互作用模式太复杂,很难在实验条件下进行研究这一重要问题,以及解决这一问题的简化方法。在人为可控的条件下进行人工模式群落的合成,就可以在动态层面上对植物和微生物作用特点进行定量评估。这篇综述总结了这一新兴领域的研究进展,以及现在相关研究的机遇与挑战。并在多个层面上探究植物与微生物互作的重要功能,进一步强调了微生物组学在生物学领域的重要地位。
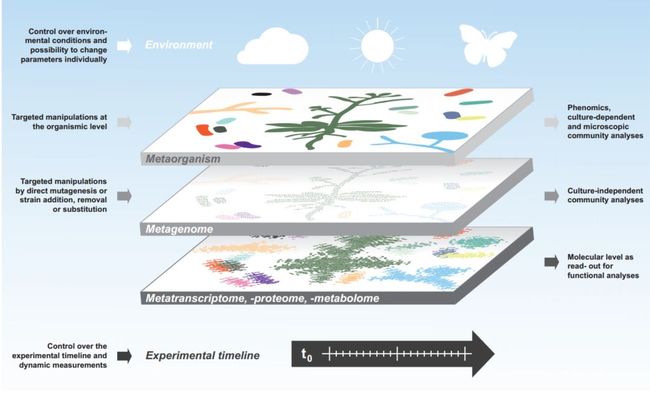
图1 植物微生物组学的研究层面
图一图注:
1.最上层代表生物层面,表示植物,微生物以及与植物相关的微环境。
2.中间层代表宏基因组层面,包括植物以及所有微生物的组学信息。
3.最下层是生物有机体的分子层面,包括宏转录组,宏蛋白组,宏代谢组方面的信息。
实验条件下可控的研究也应该从多个层面进行展开,并且需要考虑生物以及非生物的环境因素。
1.植物微生物组
随着二代测序技术的快速发展,不同宿主的微生物群落研究已经进入了新纪元。已经有多个研究关注不同群落里中相对丰度较高的物种。例如,在人体肠道中丰度最高的细菌为厚壁菌门和拟杆菌门,而在拟南芥中则为变形菌门,放线菌门,拟杆菌门和厚壁菌门。Muller等在2016的一篇综述中说明有些微生物的出现只是由于随机事件,而核心的微生物群落应该是在不同时空条件以及不同健康植物有机体之间,都能保持一定的稳定性和重复性。最近的一些研究发现,微生物群落的组装过程不是随机的,而是每年都按照相似的模式进行组装。
植物相关微生物组是与植物的表型息息相关的。已经有研究实例证明,植物相关微生物组在保护植物免受病原侵害,大量元素的获取,耐干旱与耐盐碱,植物花期等方面都有主导作用。由于这些表型都与植物健康相关,所以Theis等提出了这样的假设:植物已经进化出富集和保持其相关微生物组的功能,并能调控其相关微生物组的行为,并且植物和相关微生物组存在着共进化。
植物自身免疫系统不止在于帮助植物抵抗病原,也与有益微生物的感应相关,因此免疫系统也是形成植物相关微生物组的一个初步筛选方式。反过来,微生物对植物的的免疫系统也存在调节作用。一些植物激素,例如水杨酸,茉莉酸,乙烯都可以作为调节微生物群落的效应因子。但是以往的研究仅仅关注了少数特定的病原,以及有限的有益微生物,例如荧光假单胞菌。因此,今后的研究很有必要进行延伸,考虑不同的植物表型,例如植物营养获取,以及整个微生物群落。
Leach等2017年在Cell的一篇综述中提出phytobiome的概念:即研究植物系统时,不止考虑植物及其相关的微生物,还要考虑病毒,食草动物,环境因子等因素。
图2 植物微生物组研究中人工重复群落的概念
图二图注:人工合成/重组群落的菌株来自于植物组织材料。
基于不同的科学问题,通过由细节到整体的考察,进行人工群落中微生物的选择,并接种于特定基因型的植物上,生长在实验可控的条件下。总而言之,人工合成群落的方法可以帮助人们更加深入的理解微生物组的功能,并且可以为研发新型农业生物制剂提供理论基础。
2.构建人工群落的目的与挑战
目前关于微生物组的研究很大一部分都停留在高通量测序以及数据的关联性分析的水平上,这就导致关于微生物群落的组装,动态过程,抗逆性产生的原因等方面的研究还不清晰。所以,要深入解决微生物组的问题,就需要设计一个条件在实验室条件下可控的微生物模式群落。可控因素可以包括:某种特定的微生物是否存在,是否对某种菌或对植物进行基因改造,植物生长条件如土壤类型,湿度等。当这些条件达到可控时,就可以找出植物的基因型,微生物的基因型,以及环境因子的之间的因果关系。
为了清楚的解释科学问题,人工群落需要依据实验目的进行严格的设计。设计时需要定量考虑群落的组成及成分的相对丰度,全部的基因组信息,在转录或蛋白表达水平上的基因表达信息,和次级代谢产物以及不同尺度上的环境因素。
人工设计植物相关微生物组可以考虑一些已经有全基因组信息的植物,例如拟南芥,水稻,玉米,大豆等。当然,人们还是希望可以获得一个模式的微生物群落可以用于各个系统中。实验条件下的植物微生物组系统一方面可以揭示微生物群落间的相互作用关系,也可以找出在群落中起关键作用的核心物种。例如 van der Voort et al. 在2016年在抑制病原真菌Rhizoctonia solani的土壤中找出了起关键作用的细菌群落。
图3 低、高复杂性人工群落的优势和局限性:
图三图注:
a. 低复杂性的群落(由1-10个微生物组成):具有更高的可重复性和操作性,但是有可能缺少自然群落中的关键物种或功能物种。
b. 高复杂性的群落(由多于100个微生物组成):可以模拟自然环境,维持群落的多样性,仍具有较高的可操作性。
c. 生态学分析:最大化的还原自然的微生物多样性,可以进行相关性分析,但是可能无法进行实验证明。
3.菌株获得以及无菌植物种植
高物种多样性的菌株获得是构建人工群落的基础。如何保证选择的菌株能够更大程度上的代表自然环境是个关键问题,在实际操作中,我们可以用扩增子测序得到的样品中OTU作为一定的参考,宏基因组测序也可以提供一定的物种信息。尽管有许多生信分析的手段,但想构建一个能够代表自然环境真实微生物群落还是要尽可能的去分离和培养物种多样性高的微生物类群。使用多种培养基,包括模拟植物的培养基,以及一些特殊的寡营养的培养基,梯度改变营养物质的浓度,可以增加可培养微生物的种类。微流控技术的发展使微生物共培养成为可能,这样就可以获得一些共生以及互生物种。
更重要的是菌株的获得应该来自不同植物组织,例如叶,根,花,种子,也要来自不同的植物品种,还要来自不同的植物生长条件,比如不同的气候。目前为止,研究者对微生物菌株的分离,培养时只关注细菌,然而为了更加科学的说明问题,微生物的获得应该包括真菌,卵菌,对于某些特定植物可能还要包括古菌,这样才能全面反映植物相关微生物的多样性。
获得更多的菌株不仅可以与自然微生物群落更加相似,进行合成微生物组学时大量的菌株还可以筛选新的天然产物。研究微生物与微生物之间的作用还可以探究不同微生物的生态位分化,而微生物与植物的互作还可以帮我们理解微生物协助宿主植物抗病,促进植物生长等方面机制。此外,我们在获得可培养的微生物菌株时最好对其中的新物种进行基因组测序,以帮助我们进行功能的评估。除了考虑单菌的基因组,微生物群落的宏基因组信息对于研究基因与表型的关系也十分重要。
为了更好的研究植物与微生物组的互作,需要一个更加完善的无菌系统。已经有一些模式的无菌系统用于研究实验条件下植物的培养和植物与微生物的互作,这些系统包括含琼脂培养基的组培瓶(Magenta boxes),和多孔板/细菌培养板,以及含有土壤有机质的黏土,或者高压蒸汽处理的土壤。为了更好的模拟自然环境,还有尽可能多的考虑自然条件,例如光强度,紫外线辐射,湿度,植物密度,使这些条件尽可能与自然环境相似。
4.微生物组的设计
综合性的合成群落应该包括多种组分。低复杂性的群落有利于研究其中每种组分所起的作用,但是,这样的群落可能会导致一些自然环境中存在的网络中节点微生物的缺失。群落的复杂度提高可以更大程度的模拟自然环境,虽然高复杂性的群落也可能会缺少一些关键元素,但也可以相对说明问题。为了包括更多的组分,可以用环境样品16S,18S rRNA基因测序结果作为参考,虽然把可检测的OTU转化为可培养的菌株还存在一些问题。比如与植物密切相关的Pseudomonas属的细菌,由于植物基因型的不同,这个属内同一物种的不同菌株有的可能对植物具有有益作用然而有的却对植物起着有害作用。所以我们不能仅仅依靠这些物种的系统分类地位就对其生理功能进行定性,我们还需要对属于同一分类单元的不同菌株进行单独的功能验证。不同于依赖系统分类地位进行人工菌群重建,研究者还可以依靠相应菌株的功能基因组信息来筛选菌株,例如在人类肠道微生物研究中发现,没有一个类群的微生物能在所有的肠道样品中找到,说明肠道微生物组的功能不仅仅是被物种定义的而是被共享的基因和功能决定的。其它潜在人工功能菌群的筛选还包括是否促进植物生长,是否增强植物营养获得以及帮助宿主抵御病菌侵染等。
在构建人工微生物群落时需要注意这些问题:
这些菌株之间是否相互拮抗?
这些菌株是否能在液体或者固体培养基上生长?
用于实验的菌株生长周期多长?所有菌株是否按等比例混合,如果是应该怎么精确测定?
这些菌株应该如何接种到植物上?
这些问题都将导致结果大不相同,所以为了保证实验的可重复性,必须所有技术手段都要保证精确和一致。
图4 植物微生物组学研究中群落重建实验考察的因素
图四图注:
基于系统发生关系:例如这些群落是否在不同地区,不同宿主基因型的条件下都稳定存在
基于OTU聚类
基于相互作用网络:例如网络分析中的节点微生物
基于功能:例如考虑对宿主植物有益的功能
5.分析和解读植物微生物群落的功能
深入理解植物微生物组需要多层面的分析手段,包括:相对丰度和绝对丰度;空间分布格局以及分子分析手段。在一个低复杂度的微生物群落中,可以通过平板计数的方法进行人工微生物群落定量,但这种方法不适用于高复杂度的微生物群落。通过16S 扩增子测序可以揭示微生物的相对丰度,但无法得知每种菌的绝对丰度。对于16S基因拷贝数的问题,可以通过已经测序的细菌物种的全基因组信息进行校正。此外作者例举了一些对人工群落数据分析的方法,例如扩增子测序,宏基因组测序,基于荧光单细胞分选来分析动态过程,定量PCR等。
全基因组关联分析已经广泛用于植物基因型和表型的关联研究,未来宏基因组关联分析将建立揭示特定菌株与植物表型关联的桥梁。微生物生态模型对于我们理解微生物群落的动态变化非常重要。然而建立微生物群落结构的数学模型来预测微生物功能仍然面临巨大的挑战。微生物与微生物以及宿主之间的互作关系可以用许多方法进行预测,包括关联分析,本地相似度分析,贝叶斯网络分析等。共存网络分析能用于连续生态结构预测,包括代谢互作,群落抗性和稳定性以及鉴定核心物种和核心基因。
6.用于植物有益表型的微生物组工程
土著微生物群落对宿主植物有着重要的影响:例如营养状况,生物量的积累以及对生物或非生物胁迫的抗性。因此,可以通过选择特定的微生物群落来人工重建微生物群落,以获得具有优良特性和服务于植物健康及生长的微生物群落。虽然作为在实验室条件下进行的微生物群落的选择才刚刚发展,但它基于宿主植物表型的自然选择,已经在几千年前的原始社会的农业生产中就已经开始运用了。最重要的原则是基于植物表型的选择。Swenson等人首选运用这一原则,依据植物生物量和pH来筛选植物微生物组工程菌。最近Panke-Busse等人发现此方法选择的微生物可影响植物开花时间和发育。
我们需要使用尽可能多的微生物培养方法和手段来获得更多的微生物类群。尽管我们在分离培养时可能遗漏了植物微生物组的某些特定微生物类群,通过我们可以通过高通量测序对人工合成的微生物群落进行重新测序来帮助我们揭示与植物表型改善的微生物作用机制。人工微生物群落构建实验可以精确跟踪微生物群落结构的变化,为揭示微生物组基因型与植物表型之间潜在的因果关系提供新的科学手段。
7.人工微生物群落构建的初步探究
目前使用人工合成群落研究植物微生物组仍处于起步阶段。然而,这个领域将会由于大量微生物基因组的测序以及大量微生物菌株的获得而快速发展。Bodenhausen建立了由七个菌株组成的简单合成群落,用于研究拟南芥叶际微生物群落的组成是如何随拟南芥基因型的变化而变化。人工合成微生物群落也被用于揭示自然界中复杂的微生物与植物互作关系,例如,Lebeis等人通过构建由38个不同细菌构成的微生物群落来研究植物免疫对适当的根微生物群建立的影响。他发现拟南芥水杨酸信号的传导能显著影响拟南芥根微生物群的组装。此外他们还发现在一个由35种细菌构成的微生物群落中,在磷饥饿条件下下,人工菌群增强了PSR,PHR1和PSR转录调节因子的活性,并且PHR1不仅激活了这些条件下磷酸盐胁迫反应的基因,而且直接抑制了植物免疫基因的表达,从而在植物营养和免疫之间建立了分子联系。
牛犇等人通过构建由7个不同物种构成的微生物群落实验表明,E. cloacae在这个简化的微生物群落组装中至关重要。在没有E. cloacae的情况下Curtobacterium pusillum物种丰度增加,而其他物种则在群落中消失,这表明E. cloacae是这7个物种构成的简化微生物组中的核心微生物。此外,作者证明这7个物种构成的简化微生物组比单独的每个物种构成的微生物群落能更好地抑制Fusarium verticillioides病原菌。
Hu等人研究发现在自然土壤中生长的番茄根际加入8中不同假单胞菌群落,比加入单种假单胞菌物种抑制细菌青枯雷尔氏菌(Ralstonia solanacearum)的效果更好。作者推测这可能是由于高多样性的假单胞菌群落加剧对营养物质的竞争来抑制病原菌对宿主的侵染。总之,这些人工微生物菌群的构建研究证明了人工微生物菌群用于研究微生物与微生物互作以及与宿主互作的潜力,并且为研究者提供了将人工菌群与环境因子联系起来的第一途径。
结论
由于其生物体系的复杂性,目前对影响微生物群落的形成因素的理解还不深入。基于人工微生物群落的研究,可用于揭示微生物互作的机理,以及决定植物相关微生物群落结构与功能的因素。人工重建的微生物群落并不是要完全替代环境方面的研究,而是通过一个实验条件下可控的系统,来研究某些实验现象更深入的机制。当实验条件下可以深入理解某些机制后,也就具有了应用于温室和田间试验的潜力。人工模式微生物群落为研究植物的一些表型产生的原因确立了基础,它可以在多个层面上进行植物微生物组的研究。还可以补充进行对某些特定微生物和植物的基因修饰,来研究不同组分之间的相互作用。在作物育种方面,也可以培育一些可以筛选特定有益微生物群体的品种,或者对植物增加与微生物互作的基因。最后,这项研究不仅可以帮助我们理解机理,还可以将这些知识应用到农业中,解决现在植物微生物组应用方面所遇到的问题,例如生防菌株的易变性。而且,使用合成群落作为生物制剂的新策略可以下一代农业技术的发展方向。
Reference
Vorholt, J. A., Vogel, C., Carlström, C. I., & Mueller, D. B. (2017). Establishing causality: opportunities of synthetic communities for plant microbiome research. Cell Host & Microbe, 22(2), 142-155.
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外2600+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]() 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读