GA遗传算法
GA理论介绍
遗传算法(Genetic Algorithm)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法。(百度解释)
遗传算法的核心就是“物竞天择”。”物竞天择“通俗来说就是淘汰不适合生存的,留下对环境适应性强的物种。
就好比有一群非常神秘的物种(有些个体很普通不会飞,有些个体天生就有一个小翅膀,有些个体天生就有大翅膀),但是他们都有一个共同的喜好,就是喜欢冒险,就喜欢往悬崖峭壁跳。这时候不会飞的个体存活率就相当低,许多都死亡了,有小翅膀的个体存活率较高一些,只有少许死亡,当然拥有大翅膀的个体,只有个别死亡。这时候存活下来的个体,再次回到峭壁上,繁衍后代,遗传前代的基因,或者有些会发生变异。然后这个群体继续往悬崖跳,一直循环,最后经过几百代以后就有可能全部存活下来。最终适应环境。
遗传算法就是通过不断地迭代找到全局最优解。
在使用梯度下降解决问题时,只能找到U形函数的全局最优解,但是一旦出现类似于下图这样的函数,梯度下降就只能找到局部最优解。
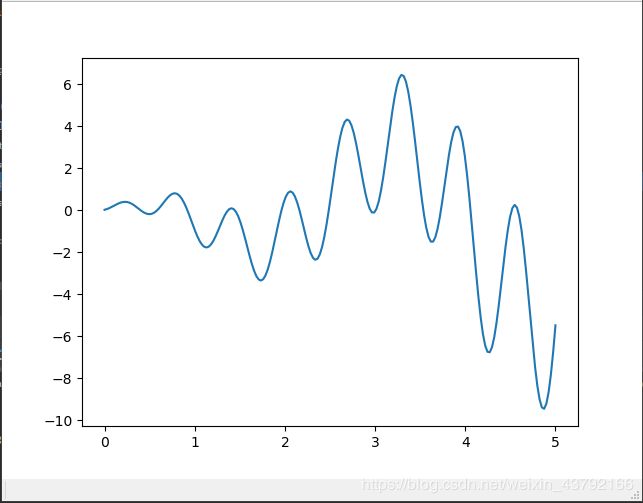
因此就需要通过遗传算法找全局最优解。
现在生成一百个个体随机放到上面的函数图中,如图
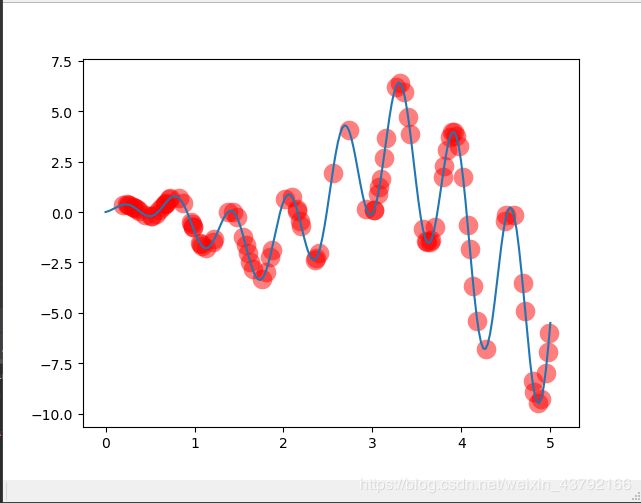
让函数值作为每一个个体在整个环境中的适应能力用英文fitness表示,也就是fitness越高,适应能力越强,现在的目的就是培养一群适应能力强的个体。因此就需要优秀的父代进行交配生出可能带有更优秀的子代。
假如现在群体只有四个,1号,2号,3号,4号,分别的fitness为1, 2, 3, 4.很明显得分越高代表基因越好就需要多交配。怎么才能做到多交配,就需要概率来控制。将fitness全部加起来就是1+2+3+4=10,每一号的概率就是0.1, 0.2, 0.3, 0.4,我现在就按照这个概率随机生成编号,那么与4号交配的概率就大了。
交配完子代个体的基因还会有坑进行变异。
然后按照上面的步骤不断地迭代就可以产生优秀的群体。
import numpy as np
import matplotlib.pyplot as plt
DNA_SIZE = 10 # DNA 长度用[0101010101]表示,交配就是交换部分基因(最为x轴输入)
POP_SIZE = 100 # population size
CROSS_RATE = 0.8 # mating probability (DNA crossover)这个个体有0.8的几率能够找到配偶进行交配
MUTATION_RATE = 0.003 # mutation probability产生基因突变的概率
N_GENERATIONS = 200#迭代次数
X_BOUND = [0, 5] # x upper and lower bounds x轴的取值范围
定义需要求最优解的函数,就是上面的图
def F(x): return np.sin(10*x)*x + np.cos(2*x)*x # to find the maximum of this function
find non-zero fitness for selection
获得函数的值的大小就是fitness,为何return这样的形式?目的就是保证算概率的时候不会出现负数,1e-3是保证不会返回0,以至于概率不为0.
def get_fitness(pred): return pred + 1e-3 - np.min(pred)
convert binary DNA to decimal and normalize it to a range(0, 5)
基因是二进制,转化为十进制,并且控制在0到5 区间上
def translateDNA(pop): return pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) / float(2**DNA_SIZE-1) * X_BOUND[1]
根据概率选择待交配的群体
def select(pop, fitness): # nature selection wrt pop's fitness
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True,
p=fitness/fitness.sum())
return pop[idx]
0.8的概率获得交配权,获得交配权,就需要交换基因,产生新的的个体
def crossover(parent, pop): # mating process (genes crossover)
if np.random.rand() < CROSS_RATE:
i_ = np.random.randint(0, POP_SIZE, size=1) # select another individual from pop
cross_points = np.random.randint(0, 2, size=DNA_SIZE).astype(np.bool) # choose crossover points
parent[cross_points] = pop[i_, cross_points] # mating and produce one child
return parent
基因突变,每个基因有0.003的概率突变0变为1,1变为0
def mutate(child):
for point in range(DNA_SIZE):
if np.random.rand() < MUTATION_RATE:
child[point] = 1 if child[point] == 0 else 0
return child
产生群体
pop = np.random.randint(2, size=(POP_SIZE, DNA_SIZE)) # initialize the pop DNA
画出最原始的图
plt.ion() # something about plotting
x = np.linspace(*X_BOUND, 200)
plt.plot(x, F(x))
迭代产生很多代子代
for _ in range(N_GENERATIONS):
F_values = F(translateDNA(pop)) # compute function value by extracting DNA
# something about plotting
if 'sca' in globals(): sca.remove()
sca = plt.scatter(translateDNA(pop), F_values, s=200, lw=0, c='red', alpha=0.5); plt.pause(0.05)
# GA part (evolution)
fitness = get_fitness(F_values)
print("Most fitted DNA: ", pop[np.argmax(fitness), :])
pop = select(pop, fitness)
pop_copy = pop.copy()
for parent in pop:
child = crossover(parent, pop_copy)
child = mutate(child)
parent[:] = child # parent is replaced by its child
plt.ioff(); plt.show()