PCA降维及SVD
PCA降维
1.相关背景
我们在实际工作中经常需要分析不同组呈现来的成千上百个指标的数据,这些指标之间经常有一些相关性指标,比如厘米和英尺,这样的指标我们只要保留一个就可以,还有一些隐藏的高度相关的特征,以通过降维方法来进行数据预处理。
2. 数据降维
主成分分析(Principal Component Analysis,简称PCA)是一种常用的降维方法,属于无监督学习。所谓降维就是将数据指标从高维度减到低维度,因为低维度的数据有如下优点:
1) 更容易进行数据分析和数据可视化
2)更容易进行数据存储
3)降低算法的运行成本
3.PCA原理
样本点分布在正交属性空间中,我们如何找到一个超平面(直线的高维推广)对所有样本点最合适的表达?
- 1.最近重构性:样本点到这个超平面的距离足够近(类似线性回归)
- 2.最大可分性:样本点到这个超平面的投影尽可能分开(方差最大化)
以上两种方式得到的主成分分析的推导是等价的,下面从”最大可分析“进行推导PCA的过程。
3.1 向量的表示及基变换
3.1.1 向量的內积
a ⃗ ⋅ b ⃗ = ∣ a ⃗ ∣ ∣ b ⃗ ∣ c o s α ( α 为 两 个 向 量 的 夹 角 ) \vec a\cdot\vec b = |\vec a||\vec b|cos\alpha(\alpha为两个向量的夹角) a⋅b=∣a∣∣b∣cosα(α为两个向量的夹角)。
几何意义:a向量在b向量上的投影数乘b向量的模,也等于b向量在a向量上的投影数乘a向量的模,投影有正负,取决于两个向量的夹角是否大于90度,如果大于90度则为负。
根据几何意义,假设将 b ⃗ \vec b b的模设为1,那么 a ⃗ ⋅ b ⃗ = ∣ a ⃗ ∣ c o s α \vec a\cdot\vec b = |\vec a|cos\alpha a⋅b=∣a∣cosα,那么此时代表的几何意义就是向量a在向量b上的投影。接下来引入”基”
3.1.2 基
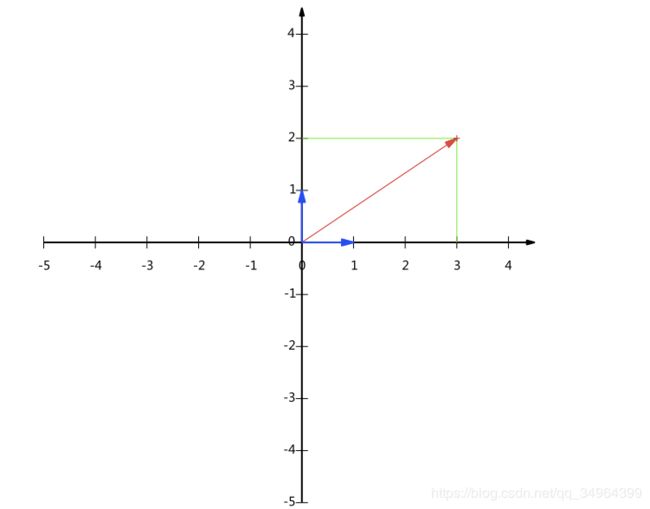
(3,2)可以表示为向量,实际表示线性组合: x ( 1 , 0 ) T + y ( 0 , 1 ) T x(1,0)^T + y(0,1)^T x(1,0)T+y(0,1)T。 x = ( 3 , 2 ) ⋅ ( 1 , 0 ) T = 3 , y = ( 3 , 2 ) ⋅ ( 0 , 1 ) T = 2 x = (3,2)·(1,0)^T=3, y = (3,2)·(0,1)^T=2 x=(3,2)⋅(1,0)T=3,y=(3,2)⋅(0,1)T=2,实际代表的是一个向量(3,2)在(1,0)所在直线上的投影(就是我们一般称为的x轴)和向量(3,2)在(0,1)所在直线上的投影(y轴),我们把(1,0)和(0,1)叫做二维空间的一组基。
类似(1,0,0) (0,1,0)和(0,0,1)叫做三维空间的一组正交基,高维空间类似推理。
我们之所以选(1,0)和(0,1)作为“基”,是因为它们分别是x和y轴上正方向上的单位向量,这能够使得我们的二维平面上的点的坐标和向量一一对应,便于分析。
实际任何两个线性无关的向量都可以表示一组基,所谓线性无关即向量不能相互表示,对于二维来说两个向量不在一条直线上。
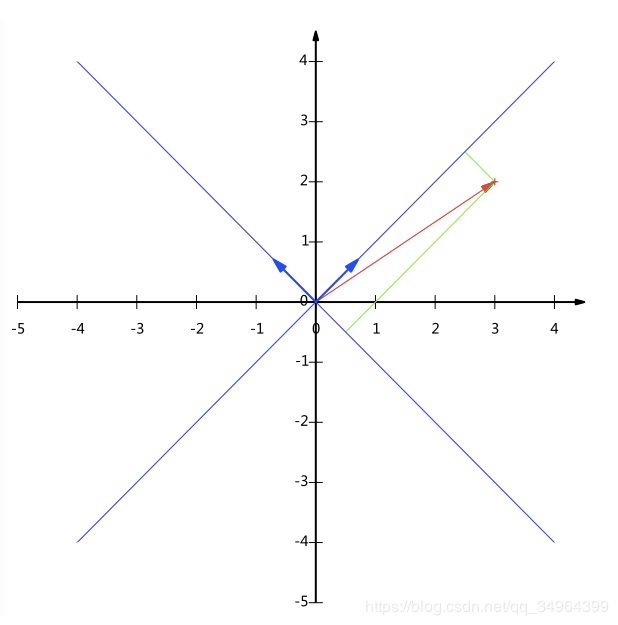
例如(1,1)和(-1,1)也可以成为一组基,但如果将基的模设为1,那么向量与基的内积就是在该基上的坐标。可以把上面的基单位化变成 ( 1 2 , 1 2 ) 和 ( − 1 2 , 1 2 ) (\frac{1}{\sqrt2},\frac{1}{\sqrt2})和(-\frac{1}{\sqrt2},\frac{1}{\sqrt2}) (21,21)和(−21,21)。
3.1.3 矩阵表示基变换
用上面的例子通过矩阵形式进行基变换。
[ 1 2 1 2 − 1 2 1 2 ] [ 3 2 ] = [ 5 2 − 1 2 ] \left[\begin{matrix}\frac{1}{\sqrt2}&\frac{1}{\sqrt2}\\-\frac{1}{\sqrt2}&\frac{1}{\sqrt2} \end{matrix}\right]\left[\begin{matrix}3\\2\end{matrix}\right] =\left[\begin{matrix}\frac{5}{\sqrt2}\\-\frac{1}{\sqrt2} \end{matrix}\right] [21−212121][32]=[25−21]
从上面的形式可以看出,等式左侧的左矩阵的每一个行代表一个基,(3,2)代表需要变换的数据点,经过矩阵变换后的数值 5 2 和 − 1 2 \frac{5}{\sqrt2}和-\frac{1}{\sqrt2} 25和−21就成了新的基的坐标。
推广到多个样本集中就是将右边矩阵的每一列列向量( a i a_i ai:第i个样本数据)变换到左边矩阵的每一列行向量( p i p_i pi)所代表的的基中,如下图所示。
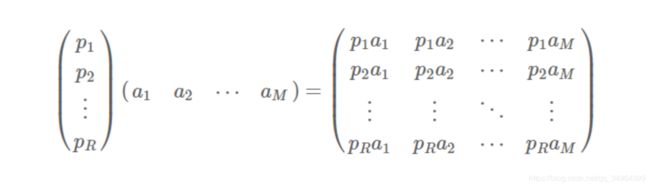
3.2 协方差矩阵及优化目标
3.2.1 方差
如果选取的基的数量小于原始样本集的维度,通过基变换就能达到降维的效果。那如何才能选取我们需要的基,我们的目标是降维后的数据B最大程度保留原有数据A的信息。更直观的说就是投影后的数据B尽可能的分散,所以可以使得数据B的各个维度的方差尽可能的大,方差公式如下:
- v a r ( X ) = c o v ( X , X ) = E [ ( X − E [ X ] ) 2 ] var(X) = cov(X,X) = E[(X-E[X])^2] var(X)=cov(X,X)=E[(X−E[X])2]
我们中心化后,均值E[X] = 0,那么公式如下 - v a r ( X ) = c o v ( X , X ) = E [ X 2 ] = 1 m ∑ i = 1 n x i 2 var(X) = cov(X,X) = E[X^2] = \frac{1}{m}\sum_{i=1}^nx_i^2 var(X)=cov(X,X)=E[X2]=m1∑i=1nxi2
3.2.2 协方差
但是如果单纯的追求使得数据B的方差最大的基方向,后续方向几乎和第一个方向重合在一起。
所以为了让两个维度表示更多的原始信息,我们需要降维后的字段之间不存在相关性,因为相关性代表两个字段之间存在着重复的信息。协方差是表示两个维度的相关性,公式如下:
- c o v ( X , Y ) = E [ ( X − E [ X ] ( Y − E [ Y ] ) cov(X,Y) = E[(X-E[X](Y-E[Y]) cov(X,Y)=E[(X−E[X](Y−E[Y])
因为中心化,E[X] = 0 - c o v ( X , Y ) = E [ ( X Y ) = 1 m ∑ i = 1 m x i y i cov(X,Y) = E[(XY) = \frac{1}{m}\sum_{i=1}^mx_iy_i cov(X,Y)=E[(XY)=m1∑i=1mxiyi
3.2.3 优化目标
综上,我们的目标变成:寻找K个单位正交基,使得原始数据映射到这组正交基上后的新数据的各个维度之间的协方差为0,各个维度的方差尽可能的大(取最大的K个方差)
这样的目标正好可以用协方差矩阵
3.2.4 协方差矩阵
任意一个mn的矩阵Z, 1 m Z Z T \frac{1}{m}ZZ^T m1ZZT 正好是协方差矩阵,可以表示我们的优化目标,为了方便,设Z为一个22的矩阵,那么
- 1 m Z Z T = ( 1 m ∑ i = 1 n x i 2 1 m ∑ i = 1 m x i y i 1 m ∑ i = 1 m x i y i ∑ i = 1 n y i 2 ) \frac{1}{m}ZZ^T=\begin{pmatrix}\frac{1}{m}\sum_{i=1}^nx_i^2 \quad \frac{1}{m}\sum_{i=1}^mx_iy_i \\\\ \frac{1}{m}\sum_{i=1}^mx_iy_i \quad \sum_{i=1}^ny_i^2 \end{pmatrix} m1ZZT=⎝⎛m1∑i=1nxi2m1∑i=1mxiyim1∑i=1mxiyi∑i=1nyi2⎠⎞
3.2.5 优化目标转成协方差矩阵
我们的优化目标是将降维后的矩阵方差最大协方差为0。设原始数据对应矩阵为X(行向量表示每个维度即特征,列向量表示每个样本的所有特征),P是一组基按行组成的矩阵,设Y = PX,则Y为X对P做基变换后的数据,设Y的协方差为D,那么:
- D = 1 m Y Y T = 1 m ( P X ) ( P X ) T = 1 m P X X T P T = P ( 1 m X X T ) P T D = \frac{1}{m}YY^T \\ \quad =\frac{1}{m}(PX)(PX)^T \\ \quad =\frac{1}{m}PXX^TP^T\\\quad=P(\frac{1}{m}XX^T)P^T D=m1YYT=m1(PX)(PX)T=m1PXXTPT=P(m1XXT)PT
= P C P T \quad=PCP^T =PCPT – 此时,发现 1 m X X T \frac{1}{m}XX^T m1XXT 正好是原始数据矩阵X的协方差矩阵,设原始矩阵的协方差矩阵为C。
这样问题就转换成了求一个矩阵P能使得原始矩阵X的协方差矩阵C对角化,且对角元素从大到小排列,取P的前K行就是需要寻找的基矩阵,用基矩阵右乘X就将X从N维降到K维上
3.2.6 协方差矩阵对角化
定理: 对 称 阵 M , 必 有 正 交 阵 E , 使 得 E − 1 M E = E T M E = Λ , 其 中 Λ 是 以 M 的 n 个 特 征 值 为 对 角 元 的 对 角 阵 。 对称阵M,必有正交阵E,使得E^{-1}ME =E^TME =\Lambda,其中\Lambda是以M的n个特征值为对角元的对角阵。 对称阵M,必有正交阵E,使得E−1ME=ETME=Λ,其中Λ是以M的n个特征值为对角元的对角阵。 M的特征向量经过施密特正交化后构成E,E的特征向量位置与 Λ 中 特 征 值 λ \Lambda中特征值\lambda Λ中特征值λ位置一一对应。
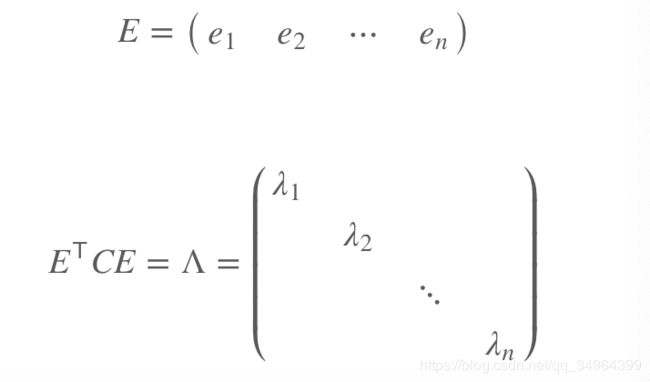
- 原始矩阵X经过中心化,协方差矩阵C的特征向量之间正交,不需要经过正交化
所以经过中心化的原始数据X构成的协方差矩阵C的特征向量按行构成的矩阵P(相当于定理中的 E T E^T ET), P C P T PCP^T PCPT 则为对角阵。如果将特征向量根据特征值 λ \lambda λ从大到小排列构成矩阵P,则对角阵 P C P T PCP^T PCPT构成的对角阵D的主对角线值为从上到下排列。最后,取P的前K行构成新矩阵Q再右乘X就将原始数据X降低到K维。
4 算法与实例
4.1 PCA算法实现流程
设有m条n个维度(特征)的数据
- 1 构建原始矩阵X (n行m列, 每 列 为 一 个 样 本 , 每 行 为 一 个 特 征 \red{每列为一个样本,每行为一个特征} 每列为一个样本,每行为一个特征)
- 2 中心化(每行减去对应行的均值,即每个特征中心化)
- 3 求协方差矩阵 C = 1 m X X T C = \frac{1}{m}XX^T C=m1XXT
- 4 求出协方差矩阵C的特征向量及特征值并标准化( 特 征 向 量 有 没 有 除 m 用 n p . l i n g . e i g / s v d 算 出 来 值 都 一 样 , 不 过 特 征 值 改 变 \red{特征向量有没有除m用np.ling.eig/svd算出来值都一样,不过特征值改变} 特征向量有没有除m用np.ling.eig/svd算出来值都一样,不过特征值改变)
- 5 按特征值大小将特征向量从上到下按行排列成矩阵P
- 6 降维(取前k行组成矩阵P1,Y1 = P1X 即为降到k维后的数据)
4.2 实例
- 2)直接中心化的一组数据X
( − 1 − 1 0 2 0 − 2 0 0 1 1 ) \begin{pmatrix}-1&-1&0&2&0 \\-2&0&0&1&1\end{pmatrix} (−1−2−10002101) - 3)求协方差矩阵
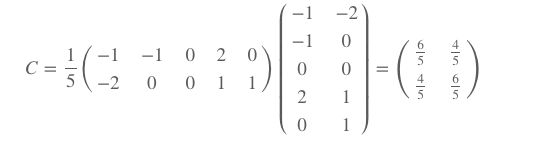
- 4)计算C的特征值及特征向量
λ 1 = 2 , λ 2 = 2 5 对 应 的 特 征 值 及 特 征 向 量 为 : c 1 = ( 1 1 ) , c 2 = ( − 1 1 ) 标 准 化 后 的 特 征 向 量 为 : c 1 = ( 1 2 1 2 ) , c 2 = ( − 1 2 1 2 ) \lambda1 = 2, \lambda2 = \frac{2}{5}\\对应的特征值及特征向量为:c_1=\begin{pmatrix}1\\1\end{pmatrix},c_2=\begin{pmatrix}-1\\1\end{pmatrix}\\ 标准化后的特征向量为:c_1=\begin{pmatrix}\frac{1}{\sqrt2}\\\frac{1}{\sqrt2}\end{pmatrix},c_2=\begin{pmatrix}-\frac{1}{\sqrt2}\\\frac{1}{\sqrt2}\end{pmatrix} λ1=2,λ2=52对应的特征值及特征向量为:c1=(11),c2=(−11)标准化后的特征向量为:c1=(2121),c2=(−2121) - 5)构建矩阵P
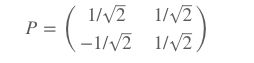
验证特征向量与C可以对角化

- 6)取P的第一行构建矩阵P1进行降维Y = P1X
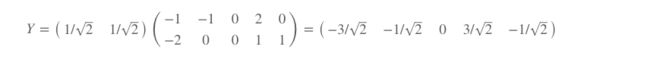
5 问题
- 为什么取特征值大的特征向量构成的矩阵Q就是我们需要的降维矩阵?
- 设特征值 λ , α \lambda, \alpha λ,α 为协方差矩阵C的一个特征值和与之对应的特征向量,那么 C α = λ α C\alpha = \lambda\alpha Cα=λα,因为等式右边为特征值与对应特征向量的乘积( λ α \lambda\alpha λα)表示在 α \alpha α向量上的拉伸, λ \lambda λ值越大伸长越大,所以等式左边矩阵C与它的特征向量的乘积( C α C\alpha Cα),也表示在 α \alpha α上的拉伸,如果 λ \lambda λ越大拉伸就越大,方差就越大,所以降维后的数据 α X \alpha X αX也就方差最大(这个所以应该是有一定关系,因为C是X的协方差矩阵)。
- D = P ( 1 m X X T ) P T D =P(\frac{1}{m}XX^T)P^T D=P(m1XXT)PT,因为D中主对角线上的值代表了方差的大小,其实也是协方差矩阵 1 m X X T \frac{1}{m}XX^T m1XXT的特征向量对应的特征值,所以取最大特征值对应的特征向量就能获得降维后的方差最大
SVD
1. 相关背景
上面PCA方法是通过对原始数据矩阵X的协方差矩阵 1 m X X T \frac{1}{m}XX^T m1XXT进行特征值分解的方法获得特征向量作为新的基,但是实际生产中可以直接对原始阵X进行svd分解获得基,因为这样更快速。
2. SVD分解
定义:一个m×n的原始数据矩阵X可以分解为 X = U Σ V T X = U \Sigma V^T X=UΣVT,其中U和V均为单位正交阵,U为左奇异矩阵,相当于 X X T XX^T XXT的特征向量按列组成的矩阵E,V为右奇异矩阵,相当于 X T X X^TX XTX的特征向量按列组成的矩阵E, Σ \Sigma Σ为对角阵,对角线上的值称为奇异值,为pca中特征值的平方根,矩阵的维度分别为 U ∈ R m × m , Σ ∈ R m × n , V ∈ R n × n U\in R^{m×m},\Sigma\in R^{m×n},V\in R^{n×n} U∈Rm×m,Σ∈Rm×n,V∈Rn×n
正常求上面的,,Σ不便于求,我们可以利用如下性质可以获得, 但 是 在 实 际 运 算 时 有 一 些 特 殊 专 门 求 解 s v d 的 算 法 进 行 计 算 \red{但是在实际运算时有一些特殊专门求解svd的算法进行计算} 但是在实际运算时有一些特殊专门求解svd的算法进行计算,并不是使用以下方法,因为以下方法还是特征值分解
X X T = U Σ V T V Σ T U T = U Σ Σ T U T XX^T = U\Sigma V^TV\Sigma ^TU^T=U\Sigma \Sigma ^TU^T XXT=UΣVTVΣTUT=UΣΣTUT,
X T X = V Σ T U T U Σ V T = V Σ T Σ V T X^TX = V\Sigma ^TU^TU\Sigma V^T=V\Sigma ^T\Sigma V^T XTX=VΣTUTUΣVT=VΣTΣVT

pca特征值分解和svd在numpy中的比较
特征值分解
X = np.mat("-1 -1 0 2 0; -2 0 0 1 1")
C = [email protected] # 没有除样本总量5,不会影响特征向量,只会影响特征值的大小
np.linalg.eig(C)
(array([10., 2.]),
matrix([[ 0.70710678, -0.70710678],
[ 0.70710678, 0.70710678]]))
svd分解
U,S,V = np.linalg.svd(X)
U,S,V
(matrix([[-0.70710678, -0.70710678],
[-0.70710678, 0.70710678]]),
array([3.16227766, 1.41421356]),
matrix([[ 0.67082039, 0.2236068 , 0. , -0.67082039, -0.2236068 ],
[-0.5 , 0.5 , 0. , -0.5 , 0.5 ],
[ 0. , 0. , 1. , 0. , 0. ],
[ 0.37894652, 0.71585306, 0. , 0.54739979, 0.21049325],
[ 0.39547381, -0.43307551, 0. , -0.01880085, 0.80974848]]))
特征向量为返回结果的matrix中的每一列,svd中的U就是我们需要的特征向量。
pca进行图像降维
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
from scipy.io import loadmat
import pandas as pd
# import seaborn as sns; sns.set()
def displayData(X):
# 实际就是把(100,400) ,每行转换成20*20 像素的小矩阵即一个数字,100行共100个数字
example_width = int(round(np.sqrt(X.shape[1])))
[m,n] = X.shape
example_height = int(n / example_width)
display_rows = int(np.floor(np.sqrt(m)))
display_cols = int(np.ceil(m / display_rows))
pad = 1
display_array = pd.DataFrame(-np.ones([pad + display_rows * (example_height + pad), pad + display_cols * \
(example_width + pad)]))
curr_ex = 0
for j in range(display_rows):
for i in range(display_cols):
max_val = max(abs(X[curr_ex,:]))
display_array.iloc[pad + j*(example_height+pad) + np.arange(example_height),\
pad + i*(example_width+pad) + np.arange(example_width)] = \
X[curr_ex,:].reshape(example_height, example_width).T / max_val # 和 matlab的reshap不同,需要加转置
curr_ex += 1
plt.imshow(display_array)
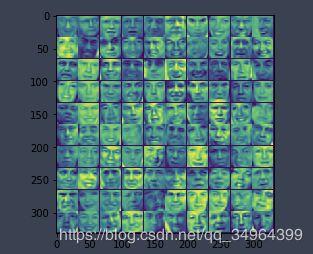
X = loadmat('ex7faces.mat')['X']
X.shape # 原始数据为5000个样本,1024个特征
(5000, 1024)
1.构建原始矩阵(行列,每列为一个样本,每行为一个特征)
2.中心化(每行减去对应行的均值,即每个特征中心化)
# 将特征数据标准化(去中心化就可以)
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler().fit(X) # 一般sklearn都是行为样本,列为特征进行标准化
norm_X = scaler.transform(X).T # 将标准化的数据转置,变成行为特征,列为样本
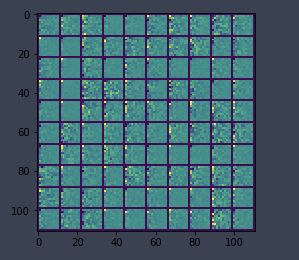
displayData(norm_X.T[:100]) # 取前100个样本进行数据展示

3 求协方差矩阵 C = 1 m X X T C = \frac{1}{m}XX^T C=m1XXT
C = (norm_X @ norm_X.T)/norm_X.shape[1]```
4.求出协方差矩阵C的特征向量及特征值并标准化
r,vec = np.linalg.eig(C)
# 或者通过svd求出特征向量 ,vec == u
u,s,v = np.linalg.svd(norm_X)
5.按特征值大小将特征向量从上到下按行排列成矩阵
6.降维(取前行组成矩阵1,1=1即为降到维后的数据)
def get_k_vec(per=0.9):
# 获取最大的k个特征和特征值
desc_r = -np.sort(-r)
total = sum(r)
# 获取最大的k个特征
for k in range(len(r)):
if desc_r[:k].sum() / total >= per:
print(k)
break
# 得到需要的索引
desc_index = np.argsort(-r)
need_index = desc_index[:k]
# 获取需要的特征向量并转置
P1 = vec[:, need_index].T
return P1
P1 = get_k_vec(0.95)
Y1 = P1 @ norm_X
# 取降维后的前100个样本的100维数据展示
displayData(Y1.T[:100,:100])
x_covered = P1.T @ Y1 # 将降维后数据通过新的基 恢复数据
displayData(x_covered.T[:100]) # 恢复数据的展示
使用sklearn降维,输入时行为样本
from sklearn.decomposition import PCA
sk_pca = PCA(n_components=1024)
Z = sk_pca.fit_transform(X)
Z.shape # (5000, 1024)
X_recover = sk_pca.inverse_transform(Z)
X_recover.shape # (5000, 1024)
displayData(X_recover[:100])