数据挖掘——谱聚类(spectral clustering)基本原理及python实现
文章目录
- 一、前言
- 二、基本原理
- (一) 无向权重图
- 1、 邻接矩阵 W
- 2、 度 D
- (二)相似矩阵/邻接矩阵 W
- 1、ϵ-邻近法
- 2、K邻近法
- 3、全连接法
- (三)拉普拉斯矩阵
- (2) 拉普拉斯矩阵的性质
- (四) 无向图切图
- 1、 子图与子图的连接权重
- 2、 切图的目标函数
- (五) 谱聚类切图
- 1、 RatioCut切图
- 2、 Ncut切图
- 三、谱聚类算法流程
- 四、python实现
- 五、sklearn库中的谱聚类使用
- 六、谱聚类算法总结
- 参考资料:
一、前言
谱聚类(spectral clustering)是广泛使用的聚类算法,比起传统的K-Means算法,谱聚类对数据分布的适应性更强,聚类效果也很优秀,同时聚类的计算量也小很多,更加难能可贵的是实现起来也不复杂。
在处理实际的聚类问题时,个人认为谱聚类是应该首先考虑的几种算法之一。
二、基本原理
谱聚类是从图论中演化出来的算法,后来在聚类中得到了广泛的应用。它的主要思想是把所有的数据看做空间中的点,这些点之间可以用边连接起来。距离较远的两个点之间的边权重值较低,而距离较近的两个点之间的边权重值较高,通过对所有数据点组成的图进行切图,让切图后不同的子图间边权重和尽可能的低,而子图内的边权重和尽可能的高,从而达到聚类的目的。
这个算法原理的确简单,但是要完全理解这个算法的话,需要对图论中的无向图,线性代数和矩阵分析都有一定的了解。下面我们就从这些需要的基础知识开始,一步步学习谱聚类。
(一) 无向权重图
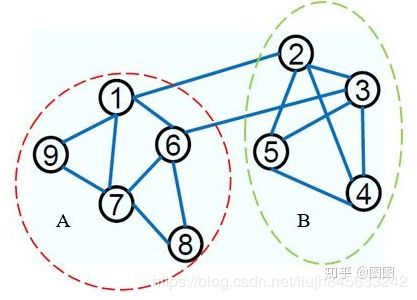
由于谱聚类是基于图论的,因此我们学习一下图的概念。对于一个图 G G G,一般用点的集合 V V V和边的集合 E E E 来描述。即为 G ( V , E ) G(V,E) G(V,E) 。其中 V V V 即为我们数据集里面所有的点 V = ( v 1 , v 2 , . . . , v n ) V=(v_{1}, v_{2}, ..., v_{n}) V=(v1,v2,...,vn) 。对于 V V V 中的任意两个点,可以有边连接,也可以没有边连接。我们定义权重 w i j w_{ij} wij为点 v i v_{i} vi 和点 v j v_{j} vj之间的权重。由于我们是无向图,所以 W i j = W j i W_{ij}=W_{ji} Wij=Wji。如下图:
1、 邻接矩阵 W
对于有边连接的两个点 v i v_{i} vi和 v j v_{j} vj , w i j > 0 w_{ij}>0 wij>0,对于没有边连接的两个点 v i v_{i} vi和 v j v_{j} vj , w i j = 0 w_{ij}=0 wij=0 .
对上面给定的图,如果我们认为连接的节点的权值是 1 1 1,没有连接的节点的权值为 0 0 0 ,则此时我们可以得到一个权值矩阵 W W W:

其中红色数字表示节点的标号,图中的每一行和每一列是对称的,他们都反映了该节点与其他节点的连接情况。
2、 度 D
定义顶点的度为该顶点与其他顶点连接权值之和:
d i = ∑ j = 1 N w i j d_{i}=\sum _{j=1}^{N} w_{ij} di=j=1∑Nwij
利用每个点度的定义,我们可以得到一个nxn的度矩阵D,它是一个对角矩阵,只有主对角线有值,对应第 i i i行的第 i i i个点的度数,定义如下:
D = ( d 1 . . . d 2 . ⋮ ⋯ ⋮ . . d n ) \mathbf {D=\begin{pmatrix} d_{1} & .& .\\ .& d_{2}&. \\ \vdots & \cdots & \vdots \\ .& .& d_{n}\\ \end{pmatrix}} D=⎝⎜⎜⎜⎛d1.⋮..d2⋯...⋮dn⎠⎟⎟⎟⎞
上面图对应的度矩阵为:

(二)相似矩阵/邻接矩阵 W
上面我们讲到了邻接矩阵 W W W,它是由任意两点之间的权重值 w i j w_{ij} wij组成的矩阵。通常我们可以自己输入权重,但是在谱聚类中,我们只有数据点的定义,并没有直接给出这个邻接矩阵,那么怎么得到这个邻接矩阵呢?
基本思想是: 距离较远的两个点之间的边权重值较低,而距离较近的两个点之间的边权重值较高,不过这仅仅是定性,我们需要定量的权重值。一般来说,我们可以通过样本点距离度量的相似矩阵 S S S来获得邻接矩阵 W W W。
构建邻接矩阵 W W W的方法有三类。ϵ-邻近法,K邻近法和全连接法。
1、ϵ-邻近法
它设置了一个距离阈值ϵ, 然后用欧氏距离 s i j s_{ij} sij 度量任意两点 x i x_{i} xi 和 x j x_{j} xj 的距离。即相似距离 s i j = ∥ x i − x j ∥ 2 2 s_{ij}=\left \| x_{i}-x_{j} \right \|_{2}^{2} sij=∥xi−xj∥22,然后根据 s i j s_{ij} sij 和 ϵ 的大小关系,来定义邻接矩阵 W W W 如下:
W i j = { 0 s i j > ϵ ϵ s i j > ϵ W_{ij}=\left\{\begin{matrix} 0 & s_{ij}>\epsilon \\ \epsilon& s_{ij}>\epsilon \end{matrix}\right. Wij={0ϵsij>ϵsij>ϵ
从上式可见,两点的权重要不就是 ϵ \epsilon ϵ, 要不就是0, 没有其他信息了。距离远近度量很不精确,因此在实际应用中,我们很少使用 ϵ \epsilon ϵ-邻近法
2、K邻近法
利用KNN算法遍历所有的样本点,取每个样本最近的 k k k个点作为近邻,只有和样本距离最近的 k k k个点之间的 w i j > 0 w_{ij}>0 wij>0。但是这种方法会造成重构之后的邻接矩阵 W W W非对称,我们后面的算法需要对称邻接矩阵。为了解决这种问题,一般采取下面两种方法之一:
第一种K 邻近法是只要一个点在另一个点的K 近邻中,则保留 S i j S_{ij} Sij
W i j = W j i = { 0 x i ∉ K N N ( x j ) a n d x j ∉ K N N ( x i ) e x p ( − ∥ x i − x j ∥ 2 σ 2 ) x i ∈ K N N ( x j ) o r x j ∈ K N N ( x i ) W_{ij}=W_{ji}=\left\{\begin{matrix} 0 & x_{i} \notin KNN(x_{j}) \quad and \quad x_{j} \notin KNN(x_{i})\\ exp(- \frac {\left \| x_{i}-x_{j} \right \|} {2\sigma ^{2}})& x_{i} \in KNN(x_{j}) \quad or \quad x_{j} \in KNN(x_{i} ) \end{matrix}\right. Wij=Wji={0exp(−2σ2∥xi−xj∥)xi∈/KNN(xj)andxj∈/KNN(xi)xi∈KNN(xj)orxj∈KNN(xi)
第二种K 邻近法是必须两个点互为K 近邻中,才能保留 S i j S_{ij} Sij
W i j = W j i = { 0 x i ∉ K N N ( x j ) o r x j ∉ K N N ( x i ) e x p ( − ∥ x i − x j ∥ 2 σ 2 ) x i ∈ K N N ( x j ) a n d x j ∈ K N N ( x i ) W_{ij}=W_{ji}=\left\{\begin{matrix} 0 & x_{i} \notin KNN(x_{j}) \quad or \quad x_{j} \notin KNN(x_{i})\\ exp(- \frac {\left \| x_{i}-x_{j} \right \|} {2\sigma ^{2}})& x_{i} \in KNN(x_{j}) \quad and \quad x_{j} \in KNN(x_{i} ) \end{matrix}\right. Wij=Wji={0exp(−2σ2∥xi−xj∥)xi∈/KNN(xj)orxj∈/KNN(xi)xi∈KNN(xj)andxj∈KNN(xi)
3、全连接法
相比前两种方法,第三种方法所有的点之间的权重值都大于0,因此称之为全连接法。
可以选择不同的核函数来定义边权重,常用的有多项式核函数,高斯核函数和Sigmoid核函数。最常用的是高斯核函数RBF,此时相似矩阵和邻接矩阵相同:
W i j = S i j = e x p ( − ∥ x i − x j ∥ 2 2 2 σ 2 ) W_{ij}=S_{ij}=exp(- \frac {\left \|x_{i}-x_{j} \right \| _{2}^{2}} {2 \sigma ^{2}}) Wij=Sij=exp(−2σ2∥xi−xj∥22)
在实际的应用中,使用第三种全连接法来建立邻接矩阵是最普遍的,而在全连接法中使用高斯径向核RBF是最普遍的。
(三)拉普拉斯矩阵
拉普拉斯矩阵(Laplacian matrix)),也称为基尔霍夫矩阵, 是表示图的一种矩阵。给定一个有n个顶点的图,其拉普拉斯矩阵被定义为:
L = D − W L=D-W L=D−W
其中 D D D为图的度矩阵, W W W为图的邻接矩阵。
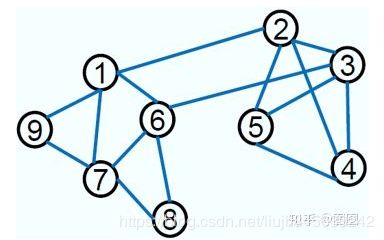
举个例子。给定一个简单的图,如下:

把此“图”转换为邻接矩阵的形式,记为: W W W

把的每一列元素加起来得到个数,然后把它们放在对角线上(其它地方都是零),组成一个 N × N N \times N N×N对角矩阵,记为度矩阵 D D D,如下图所示:

根据拉普拉斯矩阵的定义 L = D − W L=D-W L=D−W,可得拉普拉斯矩阵 L L L为:

(2) 拉普拉斯矩阵的性质
拉普拉斯矩阵 具有如下性质:
- 拉普拉斯矩阵是对称半正定矩阵,这可以由 D D D和 W W W都是对称矩阵而得;
- L 1 = 01 L 1=0 1 L1=01,即 L L L 的最小特征值是 0,相应的特征向量是 1 1 1。证明: L ∗ 1 = ( D − W ) ∗ 1 = 0 = 0 ∗ 1 L* 1 = (D -W ) * 1 = 0 = 0 * 1 L∗1=(D−W)∗1=0=0∗1。(此外,别忘了,之前特征值和特征向量的定义:若数字 λ \lambda λ 和非零向量 v ⃗ \vec {v} v 满足 A v ⃗ = λ v ⃗ A\vec{v}=\lambda \vec{v} Av=λv,则 λ \lambda λ 为的 A A A一个特征向量, v ⃗ \vec {v} v 是其对应的特征值)。
- L L L 有n个非负实特征值 0 = λ 1 ⩽ λ 2 ⩽ . . . ⩽ λ n 0=\lambda _{1} \leqslant \lambda _{2} \leqslant ... \leqslant \lambda _{n} 0=λ1⩽λ2⩽...⩽λn
- 且对于任何一个属于实向量 f ∈ R n f \in \mathbb{R}^{n} f∈Rn,有以下式子成立:
f T L f = 1 2 ∑ i , j = 1 n w i j ( f i − f j ) 2 f^{T}Lf = \frac {1} {2} \sum _{i,j=1} {n} w_{ij} (f_{i}-f_{j})^{2} fTLf=21i,j=1∑nwij(fi−fj)2
这个利用拉普拉斯矩阵的定义很容易得到如下:
f T L f = f T D f − f T W f = ∑ i = 1 n d i f i 2 − ∑ i , j = 1 n w i j f i f j = 1 2 ( ∑ i = 1 n d i f i 2 − 2 ∑ i , j = 1 n w i j f i f j + ∑ j = 1 n d j f j 2 ) = 1 2 ∑ i , j = 1 n w i j ( f i − f j ) 2 \begin {aligned} f^{T}Lf &= f^{T}Df - f^{T}Wf \\ & = \sum _{i=1} ^{n} d_{i} f_{i} ^{2} - \sum _{i,j=1} ^{n} w_{ij} f_{i} f_{j} \\ & = \frac {1} {2} ( \sum _{i=1} ^{n} d_{i} f_{i} ^{2} - 2 \sum _{i,j=1} ^{n} w_{ij} f_{i} f_{j} + \sum _{j=1} ^{n} d_{j} f_{j} ^{2}) \\ & = \frac {1} {2} \sum _{i,j=1} ^{n} w_{ij} (f_{i}-f_{j})^{2} \end {aligned} fTLf=fTDf−fTWf=i=1∑ndifi2−i,j=1∑nwijfifj=21(i=1∑ndifi2−2i,j=1∑nwijfifj+j=1∑ndjfj2)=21i,j=1∑nwij(fi−fj)2
(四) 无向图切图
1、 子图与子图的连接权重
对于无向图 G G G的切图,我们的目标是将图 G ( V , E ) G(V,E) G(V,E)切成相互没有连接的 k k k个子图,每个子图点的集合为:
A 1 , A 2 , . . . , A k , 它 们 满 足 A i ∩ A j = ∅ , 且 A 1 ∪ A 2 ∪ . . . ∪ A k = V \mathbf {A_{1}, A_{2},..., A_{k},它们满足 A_{i} \cap A_{j}=\varnothing ,且 A_{1}\cup A_{2}\cup...\cup A_{k} = V} A1,A2,...,Ak,它们满足Ai∩Aj=∅,且A1∪A2∪...∪Ak=V
定义 A A A和 B B B 是图 G G G 中两个子图,则定义子图 A A A和 B B B的切图权重为:
W ( A , B ) : = ∑ i ∈ A , j ∈ B w i j \mathbf {W(A,B):=\sum _{i \in A, j \in B} w_{ij} } W(A,B):=i∈A,j∈B∑wij
那么对于我们 k k k个子图的集合: A 1 , A 2 , . . . , A k \mathbf {A_{1}, A_{2},..., A_{k} } A1,A2,...,Ak , 我们定义切图 cut 为:
c u t ( A 1 , A 2 , . . . , A k ) = 1 2 ∑ i = 1 k W ( A i , A ˉ i ) \mathbf {cut(A_{1}, A_{2},..., A_{k} ) = \frac {1} {2} \sum _{i=1}^{k} W(A_{i}, \bar A_{i})} cut(A1,A2,...,Ak)=21i=1∑kW(Ai,Aˉi)
其中 A ˉ i \bar A_{i} Aˉi 为 A i A_{i} Ai 的补集。
2、 切图的目标函数
那么如何切图可以让子图内的点权重和高,子图间的点权重和低呢?一个自然的想法就是最小化 c u t ( A 1 , A 2 , . . . , A k ) \mathbf {cut(A_{1}, A_{2},..., A_{k} ) } cut(A1,A2,...,Ak),但是可以发现,这种极小化的切图存在问题,如下图:

我们选择一个权重最小的边缘的点,比如 C C C和 H H H之间进行cut,这样可以最小化 c u t ( A 1 , A 2 , . . . , A k ) \mathbf {cut(A_{1}, A_{2},..., A_{k} ) } cut(A1,A2,...,Ak),但是却不是最优的切图,如何避免这种切图,并且找到类似图中"Best Cut"这样的最优切图呢?接下来就来看看谱聚类使用的切图方法。
(五) 谱聚类切图
为了避免最小切图导致的切图效果不佳,我们需要对每个子图的规模做出限定,一般来说,有两种切图方式,第一种是RatioCut,第二种是Ncut。下面我们分别加以介绍。
1、 RatioCut切图
RatioCut切图为了避免上面出现的最小切图,对每个切图,不光考虑最小化 c u t ( A 1 , A 2 , . . . , A k ) \mathbf {cut(A_{1}, A_{2},..., A_{k} ) } cut(A1,A2,...,Ak) ,它还同时考虑最大化每个子图点的个数,即:
R a t i o c C u t ( A 1 , A 2 , . . . , A k ) = 1 2 ∑ i = 1 k W ( A i , A ˉ i ) ∣ A i ∣ RatiocCut(A_{1}, A_{2},..., A_{k} ) = \frac {1} {2} \sum _{i=1} ^{k} \frac {W(A_{i}, \bar A_{i})} {\left |A_{i} \right |} RatiocCut(A1,A2,...,Ak)=21i=1∑k∣Ai∣W(Ai,Aˉi)
那么怎么最小化这个RatioCut函数呢?牛人们发现,RatioCut函数可以通过如下方式表示:
我们引入指示向量 h j = h 1 , h 2 , . . . . , h k j = 1 , 2 , . . . , k h_{j}={h_{1}, h_{2}, .... , h_{k}} \quad j = 1,2,..., k hj=h1,h2,....,hkj=1,2,...,k,对于任意一个向量 h j h_{j} hj,它是一个n维向量,我们定义 h i j h_{ij} hij为:
h j i = { 0 v i ∉ A j 1 ∣ A j ∣ v i ∈ A j \mathbf {h_{ji} = \left\{\begin{matrix} 0 & v_{i}\notin A_{j}\\ \frac {1}{\sqrt {\left |A_{j}\right |}} & v_{i}\in A_{j} \end{matrix}\right.} hji={0∣Aj∣1vi∈/Ajvi∈Aj
那么我们对于 h i T L h i h_{i}^{T}Lh_{i} hiTLhi有:
h i T L h i = 1 2 ∑ m = 1 ∑ n = 1 w m n ( h i m − h i n ) 2 = 1 2 ( ∑ m ∈ A i , n ∉ A i w m n ( 1 ∣ A i ∣ − 0 ) 2 + ∑ m ∉ A i , n ∈ A i w m n ( 0 − 1 ∣ A i ∣ ) 2 ) = 1 2 ( ∑ m ∈ A i , n ∉ A i w m n 1 ∣ A i ∣ + ∑ m ∈ A i , n ∉ A i w m n 1 ∣ A i ∣ ) = 1 2 ( c u t ( A i , A ˉ i ) 1 ∣ A i ∣ + c u t ( A i , A ˉ i ) 1 ∣ A i ∣ ) = c u t ( A i , A ˉ i ) ∣ A i ∣ = R a t i o C u t ( A i , A ˉ i ) \begin{aligned} h_{i}^{T}Lh_{i} &=\frac {1}{2} \sum _{m=1} \sum _{n=1} w_{mn}(h_{im}-h_{in})^{2} \\ & = \frac {1}{2} (\sum _{m \in A_{i}, n \notin A_{i}} w_{mn} (\frac {1}{\sqrt{|A_{i}|}}-0)^{2} + \sum _{m \notin A_{i}, n \in A_{i}} w_{mn} (0-\frac {1}{\sqrt {\left | A_{i} \right |}}) ^{2}) \\ & = \frac {1}{2} (\sum _{m \in A_{i}, n \notin A_{i}} w_{mn} \frac {1}{ \left | A_{i} \right |} + \sum _{m \in A_{i}, n \notin A_{i}} w_{mn} \frac {1}{\left | A_{i} \right |} )\\ & = \frac {1} {2} (cut(A_{i}, \bar A_{i}) \frac {1} {|A_{i}|} + cut(A_{i}, \bar A_{i}) \frac {1} {|A_{i}|}) \\ & = \frac {cut(A_{i}, \bar A_{i})} {|A_{i}|} \\ & = RatioCut(A_{i}, \bar A_{i}) \end{aligned} hiTLhi=21m=1∑n=1∑wmn(him−hin)2=21(m∈Ai,n∈/Ai∑wmn(∣Ai∣1−0)2+m∈/Ai,n∈Ai∑wmn(0−∣Ai∣1)2)=21(m∈Ai,n∈/Ai∑wmn∣Ai∣1+m∈Ai,n∈/Ai∑wmn∣Ai∣1)=21(cut(Ai,Aˉi)∣Ai∣1+cut(Ai,Aˉi)∣Ai∣1)=∣Ai∣cut(Ai,Aˉi)=RatioCut(Ai,Aˉi)
上述式子列出来的是对于某一个子图 i i i,它的RatioCut对应于 h i T L h i h_{i}^{T}Lh_{i} hiTLhi, 那么对于k个子图,对应的RatioCut函数表达式为:
R a t i o C u t ( A 1 , A 2 , . . . , A k ) = ∑ i = 1 k h i T L h i = ∑ i = 1 k ( H T L H ) i i = t r ( H T L H ) RatioCut(A_{1}, A_{2},..., A_{k} ) = \sum _{i=1}^{k} h_{i}^{T}Lh_{i} = \sum _{i=1}^{k} (H^{T}LH)_{ii}= tr(H^{T}LH) RatioCut(A1,A2,...,Ak)=i=1∑khiTLhi=i=1∑k(HTLH)ii=tr(HTLH)
其中 t r ( H T L H ) tr(H^{T}LH) tr(HTLH) 为矩阵的迹。也就是说,我们的RatioCut切图,实际上就是最小化我们的 t r ( H T L H ) tr(H^{T}LH) tr(HTLH), 其中 H T H = I H^{T}H=I HTH=I,,则我们的切图优化目标为;
a r g m i n t r ( H T L H ) s . t . H T H = I \mathbf {arg min \quad tr(H^{T}LH) \quad s.t. H^{T}H=I } argmintr(HTLH)s.t.HTH=I
注意到 H H H矩阵里面的每一个指示向量都是n维的,向量中每个变量的取值为0或者 1 ∣ A i ∣ \frac {1} {\sqrt {|A_{i}|}} ∣Ai∣1,就有 2 n 2^{n} 2n种取值,有k个子图的话就有k个指示向量,共有 k 2 n k2^{n} k2n 种 H H H,因此找到满足上面优化目标的 H H H是一个NP难的问题。那么是不是就没有办法了呢?
注意观察 t r ( H T L H ) tr(H^{T}LH) tr(HTLH) 中每一个优化子目标 h i T L h i h_{i}^{T}Lh_{i} hiTLhi,其中 h h h是单位正交基, L L L是对称矩阵,此时 h i T L h i h_{i}^{T}Lh_{i} hiTLhi的最大值为 L L L 的最大特征值,最小值是 L L L 的最小特征值。如果你对主成分分析PCA很熟悉的话,这里很好理解。在PCA中,我们的目标是找到协方差矩阵(对应此处的拉普拉斯矩阵L)的最大的特征值,而在我们的谱聚类中,我们的目标是找到目标的最小的特征值,得到对应的特征向量,此时对应二分切图效果最佳。也就是说,我们这里要用到维度规约的思想来近似去解决这个NP难的问题。
对于 h i T L h i h_{i}^{T}Lh_{i} hiTLhi,目标是找到最小的 L L L的特征值,而对于 t r ( H T L H ) = ∑ i = 1 k h i T L h i tr(H^{T}LH)=\sum _{i=1}^{k} h_{i}^{T}Lh_{i} tr(HTLH)=∑i=1khiTLhi ,则我们的目标就是找到k个最小的特征值,一般来说,k远远小于n,也就是说,此时我们进行了维度规约,将维度从n降到了k,从而近似可以解决这个NP难的问题。
通过找到L的最小的k个特征值,可以得到对应的k个特征向量,这k个特征向量组成一个nxk维度的矩阵,即为我们的H。一般需要对H里的每一个特征向量做标准化,即
h i = h i ∣ h i ∣ h_{i} = \frac {h_{i}} {|h_{i}|} hi=∣hi∣hi
由于我们在使用维度规约的时候损失了少量信息,导致得到的优化后的指示向量h对应的H现在不能完全指示各样本的归属,因此一般在得到 n × k n\times k n×k 维度的矩阵H后还需要对每一行进行一次传统的聚类,比如使用K-Means聚类.
2、 Ncut切图
Ncut切图和RatioCut切图很类似,但是把Ratiocut的分母 ∣ A i ∣ |A_{i}| ∣Ai∣ 换成 v o l ( A i ) vol(A_{i}) vol(Ai) 。由于子图样本的个数多并不一定权重就大,我们切图时基于权重也更合我们的目标,因此一般来说Ncut切图优于RatioCut切图。
N C u t ( A 1 , A 2 , . . . , A k ) = 1 2 ∑ i = 1 k W ( A i , A ˉ i ) v o l ( A i ) NCut(A_{1}, A_{2},..., A_{k} ) = \frac {1} {2} \sum _{i=1} ^{k} \frac {W(A_{i}, \bar A_{i})} { vol(A_{i} )} NCut(A1,A2,...,Ak)=21i=1∑kvol(Ai)W(Ai,Aˉi)
对应的NCut切图对指示向量 h i j h_{ij} hij做了改进,注意到RatioCut切图的指示向量使用的是 1 ∣ A j ∣ \frac {1}{\sqrt {|A_{j}|}} ∣Aj∣1标示样本归属,而Ncut切图使用了子图权重 1 v o l ( A j ∣ ) \frac {1}{\sqrt {vol(A_{j}|)}} vol(Aj∣)1来标示指示向量h,定义如下:
h j i = { 0 v i ∉ A j 1 v o l ( A j ) v i ∈ A j \mathbf {h_{ji} = \left\{\begin{matrix} 0 & v_{i}\notin A_{j}\\ \frac {1}{\sqrt {vol \left (A_{j}\right )}} & v_{i}\in A_{j} \end{matrix}\right.} hji={0vol(Aj)1vi∈/Ajvi∈Aj
那么我们对于 h i T L h i h_{i}^{T}Lh_{i} hiTLhi有:
h i T L h i = 1 2 ∑ m = 1 ∑ n = 1 w m n ( h i m − h i n ) 2 = 1 2 ( ∑ m ∈ A i , n ∉ A i w m n ( 1 v o l ( A i ) − 0 ) 2 + ∑ m ∉ A i , n ∈ A i w m n ( 0 − 1 v o l ( A i ) ) 2 ) = 1 2 ( ∑ m ∈ A i , n ∉ A i w m n 1 v o l ( A i ) + ∑ m ∈ A i , n ∉ A i w m n 1 v o l ( A i ) ) = 1 2 ( c u t ( A i , A ˉ i ) 1 v o l ( A i ) + c u t ( A i , A ˉ i ) 1 v o l ( A i ) ) = c u t ( A i , A ˉ i ) v o l ( A i ) = N C u t ( A i , A ˉ i ) \begin{aligned} h_{i}^{T}Lh_{i} &=\frac {1}{2} \sum _{m=1} \sum _{n=1} w_{mn}(h_{im}-h_{in})^{2} \\ & = \frac {1}{2} (\sum _{m \in A_{i}, n \notin A_{i}} w_{mn} (\frac {1}{\sqrt{vol(A_{i})}}-0)^{2} + \sum _{m \notin A_{i}, n \in A_{i}} w_{mn} (0-\frac {1}{\sqrt {vol \left (A_{i} \right )}}) ^{2}) \\ & = \frac {1}{2} (\sum _{m \in A_{i}, n \notin A_{i}} w_{mn} \frac {1}{ vol \left( A_{i} \right)} + \sum _{m \in A_{i}, n \notin A_{i}} w_{mn} \frac {1}{vol \left ( A_{i} \right )} )\\ & = \frac {1} {2} (cut(A_{i}, \bar A_{i}) \frac {1} {vol(A_{i})} + cut(A_{i}, \bar A_{i}) \frac {1} {vol(A_{i})}) \\ & = \frac {cut(A_{i}, \bar A_{i})} {vol(A_{i})} \\ & = NCut(A_{i}, \bar A_{i}) \end{aligned} hiTLhi=21m=1∑n=1∑wmn(him−hin)2=21(m∈Ai,n∈/Ai∑wmn(vol(Ai)1−0)2+m∈/Ai,n∈Ai∑wmn(0−vol(Ai)1)2)=21(m∈Ai,n∈/Ai∑wmnvol(Ai)1+m∈Ai,n∈/Ai∑wmnvol(Ai)1)=21(cut(Ai,Aˉi)vol(Ai)1+cut(Ai,Aˉi)vol(Ai)1)=vol(Ai)cut(Ai,Aˉi)=NCut(Ai,Aˉi)
推导方式和RatioCut完全一致。也就是说,我们的优化目标仍然是
N C u t ( A 1 , A 2 , . . . , A k ) = ∑ i = 1 k h i T L h i = ∑ i = 1 k ( H T L H ) i i = t r ( H T L H ) NCut(A_{1}, A_{2},..., A_{k} ) = \sum _{i=1}^{k} h_{i}^{T}Lh_{i} = \sum _{i=1}^{k} (H^{T}LH)_{ii}= tr(H^{T}LH) NCut(A1,A2,...,Ak)=i=1∑khiTLhi=i=1∑k(HTLH)ii=tr(HTLH)
但是,此时的 H T H = I H^{T}H=I HTH=I,,而是 H T D H = I H^{T}DH=I HTDH=I, 推导如下:
h i T D h i = ∑ j = 1 n h i j 2 d j = 1 v o l ( A i ) ∑ v j ∈ A i w v j = 1 v o l ( A i ) v o l ( A i ) = I h_{i}^{T}Dh_{i} = \sum _{j=1} ^{n} h_{ij} ^{2} d_{j} = \frac {1}{vol(A_{i})} \sum _{v_{j}\in A_{i}} w_{v_{j}} = \frac {1}{vol(A_{i})} vol(A_{i}) = I hiTDhi=j=1∑nhij2dj=vol(Ai)1vj∈Ai∑wvj=vol(Ai)1vol(Ai)=I
a r g m i n t r ( H T L H ) s . t . H T D H = I \mathbf {arg min tr(H^{T}LH) \quad s.t. H^{T}DH=I } argmintr(HTLH)s.t.HTDH=I
此时我们的H中的指示向量h并不是标准正交基,所以在RatioCut里面的降维思想不能直接用。怎么办呢?其实只需要将指示向量矩阵H做一个小小的转化即可。
我们令 H = D − 1 / 2 F H=D^{-1/2}F H=D−1/2F, 则: H T L H = F T D − 1 / 2 L D − 1 / 2 F H^{T}LH=F^{T}D^{-1/2}LD^{-1/2}F HTLH=FTD−1/2LD−1/2F, H T D H = F T F = I H^{T}DH=F^{T}F=I HTDH=FTF=I,也就是说优化目标变成了:
a r g m i n t r ( F T D − 1 / 2 L D − 1 / 2 F ) s . t . F T F = I arg min tr(F^{T}D^{-1/2}LD^{-1/2}F) \quad \quad s.t. \quad F^{T}F=I argmintr(FTD−1/2LD−1/2F)s.t.FTF=I
可以发现这个式子和RatioCut基本一致,只是中间的L变成了 D − 1 / 2 L D − 1 / 2 D^{-1/2}LD^{-1/2} D−1/2LD−1/2 。这样我们就可以继续按照RatioCut的思想,求出 D − 1 / 2 L D − 1 / 2 D^{-1/2}LD^{-1/2} D−1/2LD−1/2 的最小的前k个特征值,然后求出对应的特征向量,并标准化,得到最后的特征矩阵F,最后对F进行一次传统的聚类(比如K-Means)即可。
一般来说, D − 1 / 2 L D − 1 / 2 D^{-1/2}LD^{-1/2} D−1/2LD−1/2相当于对拉普拉斯矩阵L做了一次标准化,即 L i j d i ∗ d j \frac {L_{ij}} {\sqrt {d_{i}* d_{j}}} di∗djLij
三、谱聚类算法流程
铺垫了这么久,终于可以总结下谱聚类的基本流程了。一般来说,谱聚类主要的注意点为相似矩阵的生成方式,切图的方式以及最后的聚类方法.
最常用的相似矩阵的生成方式是基于高斯核距离的全连接方式,最常用的切图方式是Ncut。而到最后常用的聚类方法为K-Means。下面以Ncut总结谱聚类算法流程。
输入: 样本集 D = ( x 1 , x 2 , . . . , x n ) D=(x_{1},x_{2},...,x_{n}) D=(x1,x2,...,xn),相似矩阵的生成方式, 降维后的维度 k 1 k_{1} k1, 聚类方法,聚类后的维度 k 2 k_{2} k2
输出: 簇划分 C ( c 1 , c 2 , . . . c k 2 ) C(c_{1},c_{2},...c_{k_{2}}) C(c1,c2,...ck2)
1)根据输入的相似矩阵的生成方式构建样本的相似矩阵 S S S
2)根据相似矩阵 S S S构建邻接矩阵 W W W,构建度矩阵 D D D
3)计算出拉普拉斯矩阵 L L L
4)构建标准化后的拉普拉斯矩阵 D − 1 / 2 L D − 1 / 2 D^{−1/2}LD^{−1/2} D−1/2LD−1/2
5)计算 D − 1 / 2 L D − 1 / 2 D^{−1/2}LD^{−1/2} D−1/2LD−1/2最小的 k 1 k_{1} k1个特征值所各自对应的特征向量 f f f
6) 将各自对应的特征向量 f f f组成的矩阵按行标准化,最终组成 n × k 1 n \times k_{1} n×k1维的特征矩阵 F F F
7)对 F F F中的每一行作为一个 k 1 k_{1} k1维的样本,共n个样本,用输入的聚类方法进行聚类,聚类维数为 k 2 k_{2} k2。
8)得到簇划分 C ( c 1 , c 2 , . . . c k 2 ) C(c_{1},c_{2},...c_{k_{2}}) C(c1,c2,...ck2)
四、python实现
import numpy as np
from sklearn.cluster import KMeans
import math
import matplotlib.pyplot as plt
def load_data(filename):
"""
载入数据
:param filename: 文件名
:return:
"""
data = np.loadtxt(filename, delimiter='\t')
return data
def distance(x1, x2):
"""
获得两个样本点之间的距离
:param x1: 样本点1
:param x2: 样本点2
:return:
"""
dist = np.sqrt(np.power(x1-x2,2).sum())
return dist
def get_dist_matrix(data):
"""
获取距离矩阵
:param data: 样本集合
:return: 距离矩阵
"""
n = len(data) #样本总数
dist_matrix = np.zeros((n, n)) # 初始化邻接矩阵为n×n的全0矩阵
for i in range(n):
for j in range(i+1, n):
dist_matrix[i][j] = dist_matrix[j][i] = distance(data[i], data[j])
return dist_matrix
def getW(data, k):
"""
获的邻接矩阵 W
:param data: 样本集合
:param k : KNN参数
:return: W
"""
n = len(data)
dist_matrix = get_dist_matrix(data)
W = np.zeros((n, n))
for idx, item in enumerate(dist_matrix):
idx_array = np.argsort(item) # 每一行距离列表进行排序,得到对应的索引列表
W[idx][idx_array[1:k+1]] = 1
transpW =np.transpose(W)
return (W+transpW)/2
def getD(W):
"""
获得度矩阵
:param W: 邻接矩阵
:return: D
"""
D = np.diag(sum(W))
return D
def getL(D,W):
"""
获得拉普拉斯矩阵
:param W: 邻接矩阵
:param D: 度矩阵
:return: L
"""
return D-W
def getEigen(L, cluster_num):
"""
获得拉普拉斯矩阵的特征矩阵
:param L:
:param cluter_num: 聚类数目
:return:
"""
eigval, eigvec = np.linalg.eig(L)
ix = np.argsort(eigval)[0:cluster_num]
return eigvec[:, ix]
def plotRes(data, clusterResult, clusterNum):
"""
结果可似化
:param data: 样本集
:param clusterResult: 聚类结果
:param clusterNum: 聚类个数
:return:
"""
n = len(data)
scatterColors = ['black', 'blue', 'green', 'yellow', 'red', 'purple', 'orange']
for i in range(clusterNum):
color = scatterColors[i % len(scatterColors)]
x1= []; y1=[]
for j in range(n):
if clusterResult[j] == i:
x1.append(data[j,0])
y1.append(data[j, 1])
plt.scatter(x1, y1, c=color, marker='+')
plt.show()
def cluster(data, cluster_num, k):
data = np.array(data)
W = getW(data, k)
D = getD(W)
L = getL(D,W)
eigvec = getEigen(L, cluster_num)
clf = KMeans(n_clusters=cluster_num)
s = clf.fit(eigvec) # 聚类
label = s.labels_
return label
if __name__ == '__main__':
cluster_num = 7
knn_k = 5
filename = '../data/Aggregation_cluster=7.txt'
data = load_data(filename=filename)
data = data[0:-1] # 最后一列为标签列
label = cluster(data, cluster_num, knn_k)
plotRes(data, label, cluster_num)
运行结果如下:
五、sklearn库中的谱聚类使用
在scikit-learn的类库中,sklearn.cluster.SpectralClustering实现了基于Ncut的谱聚类,没有实现基于RatioCut的切图聚类。同时,对于相似矩阵的建立,也只是实现了基于K邻近法和全连接法的方式,没有基于ϵϵ-邻近法的相似矩阵。最后一步的聚类方法则提供了两种,K-Means算法和 discretize算法。
对于SpectralClustering的参数,我们主要需要调参的是相似矩阵建立相关的参数和聚类类别数目,它对聚类的结果有很大的影响。当然其他的一些参数也需要理解,在必要时需要修改默认参数。
-
1)n_clusters:代表我们在对谱聚类切图时降维到的维数,同时也是最后一步聚类算法聚类到的维数。也就是说scikit-learn中的谱聚类对这两个参数统一到了一起。简化了调参的参数个数。虽然这个值是可选的,但是一般还是推荐调参选择最优参数。
-
2) affinity: 也就是我们的相似矩阵的建立方式。可以选择的方式有三类,
- 第一类是 **‘nearest_neighbors’**即K邻近法。
- 第二类是**‘precomputed’**即自定义相似矩阵。选择自定义相似矩阵时,需要自己调用set_params来自己设置相似矩阵。
- 第三类是全连接法,可以使用各种核函数来定义相似矩阵,还可以自定义核函数。最常用的是内置高斯核函数’rbf’。其他比较流行的核函数有‘linear’即线性核函数, ‘poly’即多项式核函数, ‘sigmoid’即sigmoid核函数。如果选择了这些核函数, 对应的核函数参数在后面有单独的参数需要调。自定义核函数我没有使用过,这里就不多讲了。affinity默认是高斯核’rbf’。一般来说,相似矩阵推荐使用默认的高斯核函数。
-
3) 核函数参数gamma: 如果我们在affinity参数使用了多项式核函数 ‘poly’,高斯核函数‘rbf’, 或者’sigmoid’核函数,那么我们就需要对这个参数进行调参。
- 多项式核函数中这个参数对应 K ( x , z ) = ( γ x ⋅ z + r ) d K(x,z)=(\gamma x \cdot z+r)d K(x,z)=(γx⋅z+r)d中的 γ \gamma γ。一般需要通过交叉验证选择一组合适的 γ , r , d \gamma, r , d γ,r,d
- 高斯核函数中这个参数对应 K ( x , z ) = e x p ( − γ ∣ ∣ x − z ∣ ∣ 2 ) K(x,z)=exp(− \gamma ||x−z||^{2}) K(x,z)=exp(−γ∣∣x−z∣∣2)中的 γ \gamma γ。一般需要通过交叉验证选择合适的 γ \gamma γ
- sigmoid核函数中这个参数对应 K ( x , z ) = t a n h ( γ x ⋅ z + r ) K(x,z)=tanh(\gamma x \cdot z+r) K(x,z)=tanh(γx⋅z+r)中的 γ \gamma γ。一般需要通过交叉验证选择一组合适的 γ , r \gamma, r γ,r
- γ \gamma γ默认值为1.0,如果我们affinity使用’nearest_neighbors’或者是’precomputed’,则这么参数无意义。
-
4)核函数参数degree:如果我们在affinity参数使用了多项式核函数 ‘poly’,那么我们就需要对这个参数进行调参。这个参数对应 K ( x , z ) = ( γ x ⋅ z + r ) d K(x,z)=(\gamma x \cdot z+r)d K(x,z)=(γx⋅z+r)d中的 d d d。默认是3。一般需要通过交叉验证选择一组合适的 γ , r , d \gamma,r,d γ,r,d
-
5)核函数参数coef0: 如果我们在affinity参数使用了多项式核函数 ‘poly’,或者sigmoid核函数,那么我们就需要对这个参数进行调参。
- 多项式核函数中这个参数对应 K ( x , z ) = ( γ x ⋅ z + r ) d K(x,z)=(\gamma x \cdot z+r)d K(x,z)=(γx⋅z+r)d中的 r r r。一般需要通过交叉验证选择一组合适的 γ , r , d \gamma,r,d γ,r,d
- sigmoid核函数中这个参数对应 K ( x , z ) = t a n h ( γ x ⋅ z + r ) K(x,z)=tanh(\gamma x \cdot z+r) K(x,z)=tanh(γx⋅z+r)中的r。一般需要通过交叉验证选择一组合适的 γ , r \gamma ,r γ,r
- coef0默认为1.
-
6)kernel_params:如果affinity参数使用了自定义的核函数,则需要通过这个参数传入核函数的参数。
-
7 )n_neighbors: 如果我们affinity参数指定为’nearest_neighbors’即K邻近法,则我们可以通过这个参数指定KNN算法的K的个数。默认是10.我们需要根据样本的分布对这个参数进行调参。如果我们affinity不使用’nearest_neighbors’,则无需理会这个参数。
-
8)eigen_solver:1在降维计算特征值特征向量的时候,使用的工具。有 None, ‘arpack’, ‘lobpcg’, 和‘amg’4种选择。如果我们的样本数不是特别大,无需理会这个参数,使用’'None暴力矩阵特征分解即可,如果样本量太大,则需要使用后面的一些矩阵工具来加速矩阵特征分解。它对算法的聚类效果无影响。
-
9)eigen_tol:如果eigen_solver使用了arpack’,则需要通过eigen_tol指定矩阵分解停止条件。
-
10)assign_labels:即最后的聚类方法的选择,有K-Means算法和 discretize算法两种算法可以选择。一般来说,默认的K-Means算法聚类效果更好。但是由于K-Means算法结果受初始值选择的影响,可能每次都不同,如果我们需要算法结果可以重现,则可以使用discretize。
-
11)n_init:即使用K-Means时用不同的初始值组合跑K-Means聚类的次数,这个和K-Means类里面n_init的意义完全相同,默认是10,一般使用默认值就可以。如果你的n_clusters值较大,则可以适当增大这个值。
从上面的介绍可以看出,需要调参的部分除了最后的类别数n_clusters,主要是相似矩阵affinity的选择,以及对应的相似矩阵参数。当我选定一个相似矩阵构建方法后,调参的过程就是对应的参数交叉选择的过程。对于K邻近法,需要对n_neighbors进行调参,对于全连接法里面最常用的高斯核函数rbf,则需要对gamma进行调参。
import numpy as np
from sklearn.cluster import SpectralClustering
from sklearn import metrics
import matplotlib.pyplot as plt
def load_data(filename):
"""
载入数据
:param filename: 文件名
:return:
"""
data = np.loadtxt(filename, delimiter='\t')
return data
def plotRes(data, clusterResult, clusterNum):
"""
结果可似化
:param data: 样本集
:param clusterResult: 聚类结果
:param clusterNum: 聚类个数
:return:
"""
n = len(data)
scatterColors = ['black', 'blue', 'green', 'yellow', 'red', 'purple', 'orange']
for i in range(clusterNum):
color = scatterColors[i % len(scatterColors)]
x1= []; y1=[]
for j in range(n):
if clusterResult[j] == i:
x1.append(data[j,0])
y1.append(data[j, 1])
plt.scatter(x1, y1, c=color, marker='+')
plt.show()
if __name__ == '__main__':
cluster_num = 7
knn_k = 5
filename = '../data/Aggregation_cluster=7.txt'
datas = load_data(filename=filename)
dataMat = np.mat(datas) #转换为矩阵
data = np.array(dataMat[:,0:-1])
label = np.array(dataMat[:,-1]) # 最后一列为标签列
## 调参数============
# for i, gamma in enumerate((0.01, 0.1)):
# for j, k in enumerate((3,4,5,6,7,8)):
# y_pred = SpectralClustering(n_clusters=k, gamma=gamma).fit_predict(data)
# print("Calinski-Harabasz Score with gamma=", gamma, "n_clusters=", k, "score:",
# metrics.calinski_harabaz_score(data, y_pred))
##########
y_pred = SpectralClustering(gamma=0.1,n_clusters=7).fit_predict(data)
print("Calinski-Harabasz Score", metrics.calinski_harabaz_score(data, y_pred))
plotRes(data, y_pred, cluster_num)
六、谱聚类算法总结
谱聚类是一种基于数据相似度矩阵的聚类方法,它定义了子图划分的优化目标函数,并作出改进(RatioCut和NCut),引入指示变量,将划分问题转化为求解最优的指示变量矩阵HH。然后利用瑞利熵的性质,将该问题进一步转化为求解拉普拉斯矩阵的kk个最小特征值,最后将 HH 作为样本的某种表达,使用传统的聚类方法进行聚类。
我对于谱聚类的理解是,原本相似度矩阵就是对样本点的一种特征表达(特征维数等于样本数),现在进行了谱聚类求得的特征值矩阵,实际上是对原始特征矩阵的一种降维(也可能是升维),总之就是将样本从原始空间变换(可能是线性的也可能是非线性的)到另一个空间,在这个空间中具有良好的全局欧式性。
参考资料:
- 论文:Shi, J., and J. Malik (1997) “Normalized Cuts and Image Segmentation”
- 论文:A Tutorial on Spectral Clustering
- 谱聚类(Spectral Clustering)原理及Python实现:https://blog.csdn.net/songbinxu/article/details/80838865
- 理解谱聚类 https://zhuanlan.zhihu.com/p/57369475
- 从拉普拉斯矩阵说到谱聚类:https://blog.csdn.net/guoxinian/article/details/79532893
- 谱聚类(spectral clustering)原理总结:https://www.cnblogs.com/pinard/p/6221564.html