宏基因组学揭示海洋微生物及其病毒的生态学
本文转载自“微生态笔记”, 已获授权。
标题
Metagenomics Sheds Light on the Ecology of Marine Microbes and Their Viruses
中文标题
宏基因组学揭示海洋微生物及其病毒的生态学
期刊
Trends in Microbiology,2018
第一作者
Felipe Hernandes Coutinho
Laboratory of Microbiology, Instituto de Biologia, Universidade Federal do Rio de Janeiro (UFRJ), Rio de Janeiro, Brazil; Evolutionary Genomics Group, Departamento de Produccíon Vegetal y Microbiología, Universidad Miguel Hernández (UMH), Alicante, Spain
通讯作者
Fabiano L. Thompson
Laboratory of Microbiology, Instituto de Biologia, Universidade Federal do Rio de Janeiro (UFRJ), Rio de Janeiro, Brazil; Center of Technology-CT2, SAGE-COPPE, Federal Universidade Federal do Rio de Janeiro (UFRJ), Rio de Janeiro, Brazil
编译:沈文丽
/
摘要
/
Abstract
组学方法带来的进步彻底改变了我们对海洋古菌、细菌及病毒多样性及生态过程的认识。这篇综述讨论了如何利用基因组学、代谢组学以及生态基因组学揭示海洋微生物的生态学。本文主要包括三个方面的内容:(1) 微生物在生态过程中的新作用;(2) 病毒-宿主关系;(3) 微型真核微生物与其他微生物之间的生态学关联。此外,作者还简要评论了海洋系统中新类群的发现,原核微生物分类框架的发展,蓝藻多样性及生态学的突破以及生态模型的进展。最后,讨论了该领域的局限性,并提出了未来的研究方向。
1
研究微生物生态学的健全框架
海洋微生物群落中发生了哪些生态过程?几十年来,回答这个问题一直是微生物生态学的中心目标。微生物生态学家努力理解环境因素是如何共同作用来调节微生物群落的构建,生物地球化学循环的通量,以及生态系统与生物体之间的相互作用。这些信息使我们能够预测微生物群落对生态系统变化的反应,并可能操纵它们来执行特定的功能。在当前全球气候和生态系统变化的情况下,了解微生物群落的功能从未如此相关过。
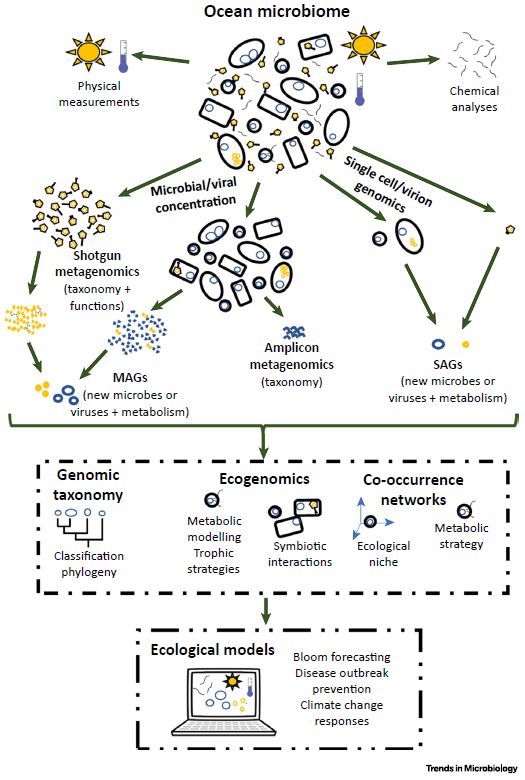
对于任何生态系统,如果不了解其生物组成,就不可能全面了解其功能。因此,确定原位微生物群落的分类组成是研究微生物生态学的基石。非培养方法 (如宏基因组学和单细胞基因组学) 在实现这一目标中发挥了根本作用,使我们现在能够绘制出与生态有关的生物体的多样性图,这些生物体长期以来一直被纯培养方法所忽视。
宏基因组组装基因组 (MAGs) 和单细胞基因组 (SAGs) 成功揭示了数千个新的微生物基因组,并且系统发育研究显示有些完全是微生物生命树的新分支。海洋生态系统一直是这些新型进化谱系的来源之一。一项单独的研究描述了近8000个高质量的MAGs,揭示了17个新的未培养细菌门 (UBP) 以及3个新的未培养古细菌门 (UAP)。关于古菌多样性的另一个重大进展是发现了Asgard总门。这一发现的意义超越了微生物学领域,因为系统基因组数据表明Asgard组中的一个门是地球真核生物的祖先。
这种新的物种多样性必须被明确分类并编组成具有进化和生态相关性的单元 (详细解读见Box1)。只有在一个明确的分类和命名框架下,我们才能跨越生态梯度调查微生物群落,并将获得的信息集成到生态模型中 (图1)。由于这些新发现的基因组中大多数都没有相关的表型信息,系统基因组学和基因组分类学在这些新种的分类中起着基本的作用。最近通过生态基因组的方法重组的蓝藻就是这样一个例子 (详细解读见Box2)。在这篇综述中,我们广泛概述了发表在2013年到2018年初的微生物生态学领域最成功的方法,旨在通过基因组学、宏基因组、生态基因方法破解海洋微生物的生态学。本文中“微生物”是指细菌、古菌和病毒,它们是本文的重点,而单细胞真核生物被称为“微真核生物”。
图1 通过生态基因组学和宏基因组学研究海洋微生物的方法![]()
![]()
Box 1
![]()
![]()
通过生态基因组学了解微生物进化以及生态策略的出现
原位研究微生物群落的一个主要屏障是微生物的分类。传统的表型分类方法通常不能在生态相关的尺度上区分微生物种类。基因分类是绕过传统方法局限性的一种选择,它被定义为一种集成比较的方法,涵盖了多个基因组特征,包括:泛基因组和核心基因组界定,平均氨基酸标识 (AAI),平均核苷酸标识 (ANI),模拟DNA-DNA杂交,核苷酸特征分析,以及基因型到表型的预测。基因组分类学和系统基因组学正成为发现新物种以及重新分类已知微生物类群的越来越普遍的方法。从表型到基因型分类学的转变重新定义了细菌和古菌的概念。从基因组学的角度来看,微生物类群可以被定义为一簇生物群之间的基因组相似性高于其他任何分支。因此,微生物分类单元可以看作是自然选择下的基因流单元簇。进化模型和基因组分析表明,尽管存在水平基因转移 (HGT) 的可能,但集群的形成需要自然选择。因此,自然选择作用于世代的基因,优化类群对其生态系统的适应性。弧菌属共生菌群适应性的快速进化就是一个例证,属于同一物种的密切相关的弧菌属种群显示出非常不同的生态策略。比较基因组学表明,弧菌基因组的多变部分通过点突变和同源重组快速进化,这些变化导致了对迅速变化的环境条件的快速适应,使得种群分为两个主要的生态位:附着型和浮游型。另外,HGT也可以驱动该属间的生态位划分过程,如降解藻酸盐的弧菌群落间的生态位快速分化,它们可以在较短的进化时间内专性进化为先驱者、收割者和食腐者。诸如此类的发现表明,即使是海洋生态系统的微尺度资源梯度也能产生基因流屏障,最终导致物种形成。
![]()
![]()
Box 2
![]()
![]()
生态基因组学重新定义了蓝藻的分类学和生态学
蓝藻是海洋生境中主要的初级生产者,因此在维持海洋生态系统方面具有根本的作用。蓝藻门的分类最初是根据形态特征确定的,不同系统发育重建表明,这种分类方法与该门的系统发育不一致,蓝藻门中的许多类群不是单细胞的,需要进行修正。因此,利用系统基因组学和基因组分类学重新定义了该门的分类。这一策略界定了57个属 (其中28新属) 和87个种 (32新种)。根据在全球宏基因组数据库中新建立的类群丰度及其与环境参数的关系,这些类群成员被划分为三个主要的生态位:低温生物、低温富营养生物和高温寡营养生物。这些类群与通过基因组分类学和系统基因组学定义的分类群相互关联,阐明了在该门中生态位的分布情况,从而将原本仅限于原绿球藻和副融合球菌 (前海洋融合球菌) 的生态型概念扩展到整个该门。
2
海洋生态系统中微生物的新作用
微生物在维持与地球生命息息相关的过程中起着根本的作用,包括:初级生产力、营养循环和寄主生理机能。MAGs (宏基因组组装基因组) 和SAGs (单细胞基因组) 对于确定在生物地球化学循环中原核生物的潜在作用尤其重要。通过这些方法发现了一些对能量和物质循环有贡献的微生物新成员,而这些成员之前是没有被解释的。SAGs的早期研究表明,来自深海的SAR324γ-变形菌门通过卡尔文 (CBB) 循环依靠硫氧化来固定碳,这就挑战了黑暗海洋中的碳固定是由泉古菌门通过3-羟丙酸/4-羟基丁酸循环驱动的观点。SAGs还证明了非光依赖型碳固定途径 (如Wood-Ljungdahl途径) 在细菌和古菌中比最初认为的更普遍。有些研究的很透彻的代谢途径受到了MAGs的挑战。例如,氨通过亚硝酸盐氧化为硝酸盐的两步过程最初被认为是由氨氧化和亚硝基氧化微生物联合单独进行的。但是硝化螺旋藻门的MAGs被证明能够执行氨氧化过程的两个步骤。此外深层含水层中MAGs的复原导致发现了深海古菌门,这些生物的基因组表明,甲烷代谢并不像之前认为的那样只发生广古菌门的微生物中。
生态基因组学还揭示了一些新的微生物生态功能,例如SAR11。这一分支最初被认为仅限于含氧海洋环境,但是单细胞基因组学和宏基因组学的结合揭示了SAR11中的一些谱系可在缺氧环境中繁殖。SAR11的这种谱系具有执行反硝化过程第一步的功能性硝酸盐还原酶,这表明SAR11物种可以促进海洋氮的消耗。好氧和缺氧谱系之间强烈的相似性表明,在这一群体的进化过程中,对缺氧环境的适应是最近才出现的。同样地,SAR11的表面和深海谱系也显示出惊人相似的基因含量,但在基因组大小、基因间隔和氨基酸含量上有细微的差异,这可能与它们适应不同的生态位有关。SAR11的好氧/缺氧以及表面/深层谱系之间这些细微的差异表明,这一群体能够迅速进化从而使它们适应更多的生态环境。
3
被忽视的大多数——海洋病毒的生态学
几十年来,由于缺乏有效的定量和分类方法,病毒一直被微生物生态学家所忽视。自从发现病毒是海洋中最丰富的微生物群后,微生物学家开始分析它们的生态功能,尤其是那些感染细菌和古菌的病毒。当病毒与宿主相互作用时,它们丰富的生物学特性就会显现出来。宏基因组学为我们理解病毒在海洋生态系统中的作用带来了许多进步。这些分析旨在扩展对海洋病毒基因和基因组多样性的认识 (详细解读见Box3),并揭示宿主和环境因素如何共同作用,从而来确定遍布地球海洋的病毒群落的组成以及病毒与宿主相互作用的机制。这些相互作用可以是变化多样的,最近的发现甚至挑战了被广泛接受的概念。
裂解病毒与宿主表面分子相互作用成功后,将其遗传物质注入宿主细胞,使其代谢失调,产生新的病毒体。据估计,海洋中每天由病毒介导的分解可释放100亿吨碳,这对全球碳循环有直接影响。尽管病毒转导通常与光区浮游细菌有关,但也有研究表明,病毒的分解作用对深海海底的古菌群有显著影响,而病毒分解是深海环境中一个重要的却被忽视的营养源。
在感染过程中,噬菌体可以表达辅助代谢基因 (AMGs),改变宿主代谢,使其向最大限度产生新病毒体的途径转变,这种现象在蓝细菌-噬藻体中表现得非常明显,它们的AMGs在补充光合电子传输的同时,将能量从碳固定转移到戊糖磷酸途径。病毒AMG的含量随环境梯度 (如透光/无光环境和冷/暖环境) 的变化而变化,这些改变涉及到光合作用、呼吸作用、碳代谢、营养获取、核苷酸代谢等许多功能的基因。因此,病毒群落的AMG含量受其宿主微生物在生态梯度中特定代谢的约束。例如,感染SUP05γ-变形菌的噬菌体编码Dsr基因,它们可能会影响宿主的硫代谢,尤其是在缺氧区和热液喷口生态系统中。噬菌体也被认为使用AMGs来调节磷和氮的获取比例。真核病毒也能影响宿主代谢,如牛黄链球菌病毒6所示,它在感染过程中表达一种宿主源转运体,从而提高宿主对铵的吸收比。同时,赫氏圆石藻病毒使用AMG改变宿主的脂质生物合成途径,产生用于病毒体组装的脂质。综上所述,这些发现表明病毒AMGs影响了大多数微生物代谢途径。因此,病毒对营养循环的重要性不只是杀死宿主,还包括改变宿主代谢的中心通路。然而,对于海洋病毒群落的AMG含量以及它们是如何根据环境梯度发生改变,目前还没有明确的认识。
另外,一些病毒可能会在溶原性周期内 (如果刺激合适的话),将它们的基因组整合到宿主的基因组中。通过溶原性病毒感染,噬菌体遗传物质随着宿主的基因组在细胞分裂过程中被动复制。然而,其他环境条件可能会再次触发裂解循环。虽然调节噬菌体感染从裂解性到溶原性转换的因素尚未完全确定,但宿主丰度和代谢活动水平等,以及光照强度、温度和养分有效性等环境因素被认为可能影响这一过程。
由于病毒通常以特定的宿主为目标,因此病毒破坏塑造了海洋微生物的组成,反之亦然。这些动态发生符合“杀死赢家”理论,即群落中最丰富、最活跃的微生物也是最容易被裂解病毒感染的目标。一旦优势微生物被消灭,病毒的数量就开始下降,因为它们缺乏可借以复制的宿主。因此,噬菌体及其宿主一直处在经典的Lotka-Volterra捕食者-猎物动力学描述的丰度振荡周期中。最近,该模型受到了病毒-宿主相互作用的“背靠赢家”理论的挑战。随着生态系统中微生物细胞浓度的增加,病毒与微生物的比率降低,这与普遍认为的模型相反。在这种模型中,细胞生产力的提高将有利于裂解感染产生更多的子代病毒。此外,微生物丰度不断增加的样品的宏基因组中富集了溶原性感染标记,而缺乏抗噬菌体感染标记。通过上述这些观察结果得出结论,溶原性感染的比例随宿主丰度的增加而增加。这使得病毒可以在不杀死宿主的情况下,通过宿主基因组的成功复制 “搭便车”复制自己的基因组。“背靠赢家”理论在病毒生态学领域仍然是一个具有争议的话题,但也有研究得出了类似的结论。此外,仲裁系统的发现揭示了一种前所未有的分子机制,噬菌体可以通过这种机制向后代发出信号,当群落中足够比例的宿主已经被感染时,将其后代转变为溶原性生活方式。
![]()
![]()
Box 3
Box 3
![]()
![]()
通过非培养方法发现新的海洋病毒谱系
研究病毒需要了解它们与宿主的相互作用,当研究病毒时,纯培养方法的限制性就被放大了。因此,只有随着分子技术的出现,才能正确地评估病毒的多样性。病毒很可能是多系的,缺乏通用的标记基因,因此必须使用鸟枪法来阐明它们的遗传多样性。由于样本浓度的限制,该技术自下一代测序仪问世以来一直是研究病毒的最先进的方法。人们从一开始就注意到,与细胞相比,病毒的基因组中编码了更多完全未知的蛋白质,它们的遗传多样性被称为生物“黑箱”。这迫使新方法的出现,即蛋白质 (包括未知蛋白) 的聚类。虽然成功地证明了病毒“黑箱”并不像最初认为的那么大,但是众多的分类限制了它们的广泛应用。其他有关宏基因组组装病毒基因组的方法最近被应用,并获得了一些生态推论。事实上,从病毒中重建完整的或部分的基因组成为研究病毒生态学的标准方法,因为病毒体序列注释的性能很差,识别这些病毒的宿主并不是那么简单,但通过基于同源匹配、CRISPR间隔、共享tRNA基因以及病毒共现相关性的生物信息学方法,可以实现暂时的宿主分配。虽然这些预测可以在属或种的层次上进行,但它们在更高的分类等级如门或纲中是最准确的。此外,还开发了新的实验方法用于预测宿主,比如对一个样品中物理连接到一起的DNA分子测序。时间序列的研究,允许病毒和宿主的相互关联;单细胞测序,可以识别活跃的病毒感染;单病毒体测序,直接识别单一的病毒基因组,这些新方法可以帮助将宿主分配给大量未培养的海洋病毒。
4
海洋微生物和微真核生物之间的联系
微真核生物 (即原生生物) 是微生物生态学的一个令人惊叹的新前沿,因为这些生物有助于海洋初级生产力和养分的循环。微真核生物涉及多种进化谱系,其基因组极其复杂,有许多未知功能的基因。宏基因组学揭示了令人震惊的海洋微真核生物多样性,并展示了它们非常广泛的生态功能和生态位模式。在这些生物体中发现了各种各样的营养方式 (如光养型、异养型、混合营养型) 和生活方式 (如浮游、底栖、共生、寄生)。微真核生物食用细菌、古生菌和病毒,甚至食用其它微真核生物。因此,它们对微生物群落起着重要的自上而下的控制作用,同时调节能量和物质向更高营养级转移。
最近的宏基因组研究扩大了已知的微真核生物的多样性,并揭示了它们与微生物群落之间新的互作方式,而不仅仅是捕食。例如,当生产毒素的微真核生物增殖到产生藻华时,往往与共生细菌形成强制性的联合。此外,通过分析全球海洋主要区域的宏基因组衍生出的全球同现网络,还发现了数千种微生物和微真核生物之间的潜在关联。虽然这些关联的生态学意义有些难以捉摸,但它表明,自由生活的微生物和微真核生物之间的许多共生关系迄今都被忽视了。
自由生活的和寄宿生活的微生物群落在分类组成上差异显著。我们目前对多细胞海洋真核生物的相关微生物群还知之甚少。最近的发现揭示了人类健康与相关微生物群落之间的多重联系。此外,对藻类、海绵动物和哺乳动物等海洋真核生物的微生物群落的研究也有了深入的发现。宏基因组学已经提出了多种假说来解释微真核生物、原核生物和病毒对珊瑚健康和疾病的作用。然而,目前还缺乏针对跨越多个真核生物类群和生态系统的比较研究。
结束语
揭示海洋微生物生态学的观点和挑战
我们才刚刚开始揭开海洋生态系统中的微生物多样性和生态群分类的神秘面纱。这些信息对于建立能够预测海洋微生物群落的组成和功能如何受到环境特征影响的机制模型至关重要 (详细解读见Box4)。尽管最近取得了突破,但我们对微生物生态学的理解仍有许多空白。生态基因组的研究通常集中在因其丰富而被认为重要的生物上 (如远洋杆菌和蓝藻),或参与生态相关过程 (如光合作用或固氮作用) 的微生物。相对而言,化能异养和化能无机自养型生物受到的关注要少得多。与此同时,宏基因组学研究主要针对的是温带和热带生态系统的上层地区的微生物群落。深海、礁系、极地水域、沉积物以及宿主相关的微生物群落的多样性分类并没有到达同一深度。要全面了解微生物对维持海洋生态系统的贡献,破译这些被忽视的生物和群落的多样性和生态作用至关重要。此外,关于海洋微生物群对人为影响的反应还需要进行更多的研究。预测这些群落如何应对未来几年全球变暖、海洋酸化、富营养化和气候变化至关重要。海洋微生物群很可能在扭转或至少减轻这些即将发生的全球变化影响的努力中发挥重要作用。
![]()
![]()
Box 4
![]()
![]()
超越基因和基因组:将数据整合到生态模型中
数学模型可以揭示微生物与环境特征之间的联系,而这些联系很难用更简单的方法来识别。在一项对广域的原氯球菌和聚球菌细胞丰度的研究中,作者利用机器学习整合了35年来这些蓝藻细菌丰度的数据,建立了强大的预测模型,该模型可以根据环境条件准确估计这些生物的全球丰度。这些模型被用来估计全球变暖对全球海洋中蓝藻细菌丰度的深远影响,提供了对海洋生态系统对气候变化响应的见解。机器学习方法也成功地预测了蓝藻水华的动态。由于毒素的产生,这些事件可能对海洋和淡水生境造成灾难性的影响,但这种水华的发生可以通过基于环境变量的模型或基于多年抽样得到的时间序列组学数据来预测。建模方法也被用于弥补公共卫生和微生物生态学之间的空白。一项研究模拟了海洋弧菌 (一种重要的水生人类病原体) 的丰度,该研究以近半个世纪的数据来确定控制这种生物丰度的因素,并发现了气候变化导致弧菌感染频率增加的证据,此外,整合了不同营养水平上的生物和非生物关联的模型可以帮助开发战略来保护易受人为影响的海洋系统。
新技术正在为深入了解海洋微生物群落的多样性和生态学铺平道路。第三代测序技术 (如纳米孔、Illumina TruSeq和Pacific Biosciences) 正在迅速优化,以提供更长的和更精确的测序读数。这些进步,加上更好的纠错和装配管道,将使获得高质量的MAGs (宏基因组组装基因组) 和SAGs (单细胞基因组) 变得更加容易,特别是MAGs的分析将受益于应变甚至分子水平的分辨率。这一进展将允许在全基因组水平上研究种群水平的变异。然而,(宏/生态) 基因组方法的一个主要局限性是,微生物基因组编码的大量蛋白质没有注释。因此,它们不能关联到任何生理或生态过程中。由宏基因组学获得的发现依赖于高质量的湿实验室分析,这些分析可以阐明微生物细胞内部和之间发生的许多代谢功能。转录组学、蛋白质组学和代谢组学等未得到充分开发的技术,可以帮助我们对微生物群落进行更深入的了解。与此同时,稳定同位素探测等方法可以对具有代谢同位素标记的化合物的微生物基因组进行定向测序。通过这些过程,就有可能获得MAGs、SAGs,甚至整个宏基因组,从而丰富参与某个特定过程的微生物,比如甲基化或碳氢化合物降解。
宏基因组学的另一个局限性是它只产生相对丰度,这阻碍了定量研究的发展。最近开发出一种产生绝对丰度的新技术。该技术通过将宏组测序与细胞丰度计数进行配对表明,相对微生物计数和绝对微生物计数可以截然不同。虽然实验是在粪便样本中进行的,但细胞计数已经有标准程序,因此该方法在海洋微生物研究中也有很大的应用前景。在利用绝对丰度分析时,许多观察到的分类单元之间或分类单元与环境参数之间的关联可能不成立。因此,从相对到绝对计数的改变,在宏基因组学中有可能通过揭示较少受偏差影响的联系,从而彻底改变我们对微生物生态学的理解。
一种新的考虑了类群的丰度分布并与基因组规模的代谢模型相关联的方法被用于预测人类代谢组中200多种代谢产物的浓度,该方法所获得的测量结果与在多个身体部位进行的实验评估结果相吻合,证实了它的有效性。日益增长的海洋样品的宏基因组数据和构建MAGs和SAGs代谢模型的可能,使基因水平的代谢模型成为研究海洋中的代谢通量的一种很有前景的方法,该方法可以对不同微生物如何使用资源并促进全球生物地球化学过程进行预测。
近年来,海洋微生物学领域迎来了激动人心的时代。新技术极大地促进了我们对海洋生态系统中微生物多样性和生态学的理解。生态基因组学成为该领域的一个中心学科,很大程度上受益于塔拉(Tara)海洋探险和地球微生物学等大型研究项目。将基于组学的研究方法与实验数据相结合是实现突破的关键。未来几年,微生物生态学的发展将继续依靠多学科交叉的方法来探索广阔的生态梯度的微生物群落。长期存在的问题正在得到解答,当我们正在接近了解微生物如何有助于维持地球上的生命时,许多新的和令人兴奋的问题也在被提出。
在本文的最后,作者提出了几个还未解决的问题,供大家思考和研究。
由于方法或取样的限制,我们是否可能忽略了微生物生命树的主要谱系?
最近发现的海洋古菌、细菌和病毒参与哪些生态交互作用和生态系统维持过程?
转录组学、蛋白质组学和代谢组学能否帮助我们更全面地了解海洋微生物群落之间的生态相互作用?
哪些生理功能是由来自海洋微生物和病毒基因组的未识别的蛋白编码的?这些蛋白质的生态相关性是什么?
宿主微生物群落的多样性是什么?在维持海洋大型生物和生态系统的健康方面起了什么作用?
像全球变暖和海洋酸化这样的人为干扰在多大程度上影响了海洋微生物群落之间的相互作用?
参考文献
Coutinho F H, Gregoracci G B, Walter J M, et al. Metagenomics Sheds Light on the Ecology of Marine Microbes and Their Viruses[J]. Trends in Microbiology, 2018.
原文链接
https://www.sciencedirect.com/science/article/pii/S0966842X18301331
中国科学院生态环境研究中心
环境生物技术重点实验室
邓晔 研究员课题组发布
作者:沈文丽
编辑:吴悦妮
关注微生态笔记~
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外2200+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()