检测X光图像中Covid-19
作者|Marcelo Rovai 编译|VK 来源|Towards Data Science
免责声明
本研究是为X光图像中COVID-19的自动检测而开发的,完全是为了教育目的。由于COVID-19没有经过专业或学术评估,最终的应用并不打算成为一个准确的用于诊断人类的COVID-19的诊断系统,。
介绍
Covid-19是由一种病毒(SARS-CoV-2冠状病毒)引起的大流行性疾病,已经感染了数百万人,在几个月内造成数十万人死亡。
据世界卫生组织(WHO)称,大多数COVID-19患者(约80%)可能无症状,约20%的患者可能因为呼吸困难而需要住院治疗。在这些病例中,大约5%可能需要支持来治疗呼吸衰竭(通气支持),这种情况可能会使重症监护设施崩溃。抗击这一流行病的关键是快速检测病毒携带者的方法。
冠状病毒
冠状病毒是引起呼吸道感染的病毒家族。这种新的冠状病毒病原体是在中国登记病例后于1919年底发现。 它会导致一种名为冠状病毒(COVID-19)的疾病。
1937年首次分离出人冠状病毒。然而,直到1965年,这种病毒才被描述为冠状病毒,因为它在显微镜下的轮廓看起来像一个树冠。在下面的视频中,你可以看到SARS-CoV-2病毒的原子级三维模型:
X光
近年来,基于计算机断层扫描(CT)的机器学习在COVID-19诊断中的应用取得了一些有希望的成果。尽管这些方法取得了成功,但事实仍然是,COVID-19传播在各种规模的社区。
X光机更便宜、更简单、操作更快,因此比CT更适合在更贫困或更偏远地区工作的医疗专业人员。
目标
对抗Covid-19的一个重大挑战是检测病毒在人体内的存在。因此,本项目的目标是使用扫描的胸部X光图像自动检测肺炎患者(甚至无症状或非病人)中Covid-19的病毒。这些图像经过预处理,用于卷积神经网络(CNN)模型的训练。
CNN类型的网络通常需要一个广泛的数据集才能正常工作。但是,在这个项目中,应用了一种称为“迁移学习”的技术,在数据集很小的情况下非常有用(例如Covid-19中患者的图像)。
目的是开发两种分类模型:
Covid-19的检测与胸片检测正常的比较
Covid-19的检测与肺炎患者的检测的比较
按冠状病毒相关论文定义的,所有类型的肺炎(COVID-19病毒引起的除外)仅被认为是“肺炎”,并用Pneumo标签(肺炎)分类。
我们使用TensorFlow 2.0的模型、工具、库和资源,这是一个开源平台,用于机器学习,或者更准确地说,用于深度学习。最后在Flask中开发了一个web应用程序(web app),用于在接近现实的情况下进行测试。
下图为我们提供了最终应用程序如何工作的基本概念:
X光扫描胸部图像(User_A.png),应用程序将图像存储在web应用程序的计算机上,决定图像是否属于受病毒污染的人(模型预测:[阳性]或[阴性])。在这两种情况下,应用程序都会通知预测的准确性(模型准确度:X%)。
为了避免两者都出错,将向用户显示原始文件的名称及其图像。图像的新副本存储在本地,其名称添加一个预测标签,并且加上准确度。
这项工作分为四个部分:
环境设置、数据清洗和准备
模型1训练(Covid/正常)
模型2训练(Covid/肺炎)
Web应用的开发与测试
灵感
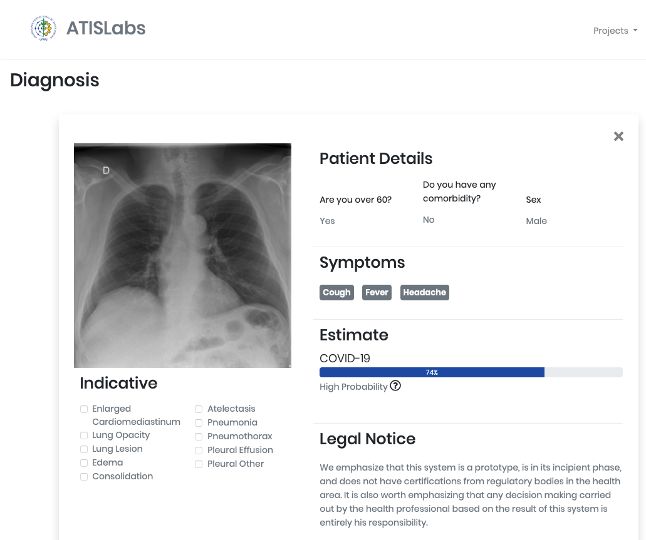
该项目的灵感来源于UFRRJ(里约热内卢联邦大学)开发的X光COVID-19项目。UFRRJ的XRayCovid-19是一个正在开发的项目,在诊断过程中使用人工智能辅助健康系统处理COVID-19。该工具的特点是易用、响应时间快和结果的有效性高,我希望将这些特点扩展到本教程第4部分开发的Web应用程序中。下面是诊断结果之一的打印屏幕(使用了Covid-19数据集1图像之一):
乔杜里等人在论文中阐述了该大学开展这项工作的科学依据,论文地址:https://arxiv.org/abs/2003.13145
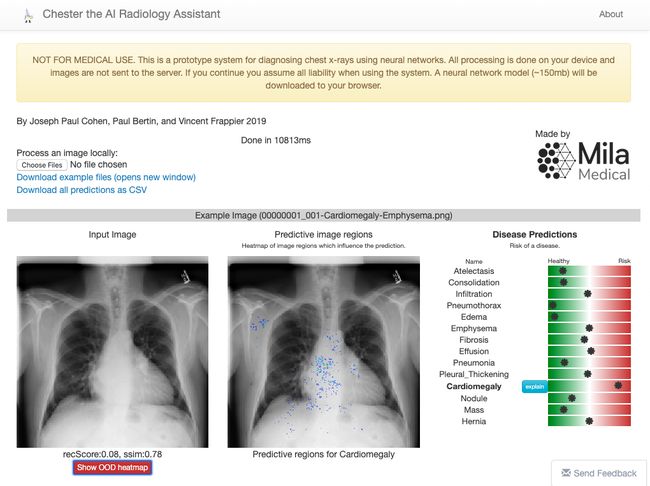
另一项工作是Chester,论文:https://arxiv.org/pdf/1901.11210.pdf,由蒙特利尔大学的研究人员开发。 Chester是一个免费且简单的原型,医疗专业人员可以使用它们来了解深度学习工具的实际情况,以帮助诊断胸部X光。 该系统被设计为第二意见,用户可在其中处理图像以确认或协助诊断。
当前版本的 Chester(2.0)使用DenseNet-121型卷积网络训练了超过10.6万张图像。该网络应用程序未检测到Covid-19,这是研究人员对应用程序未来版本的目标之一。 下面是诊断结果之一的截图(使用了Covid-19数据集的图像)
在下面的链接中,你可以访问Chester,甚至下载应用程序供脱机使用:https://mlmed.org/tools/xray/。
感谢
这项工作最初是根据Adrian Rosebrock博士发表的优秀教程开发的,我强烈建议你深入阅读。此外,我要感谢Nell Trevor,他根据罗斯布鲁克博士的工作,进一步提出了如何测试结果模型的想法。
第1部分-环境设置和数据准备
数据集
训练模型以从图像中检测任何类型的信息的第一个挑战是要使用的数据量。 原则上,可公开获取的图像数量越多越好,但是请记住,这种流行病只有几个月的历史,所以对于Covid-19检测项目来说,情况并非如此)
但是,Hall等人的研究,论文:https://arxiv.org/pdf/2004.02060.pdf,证明使用迁移学习技术仅用几百幅图像就可以获得令人鼓舞的结果。
如引言所述,训练两个模型;因此,需要3组数据:
- 确认Covid-19的X光图像集
- 常规(“正常”)患者的X光图像集
- 一组显示肺炎但不是由Covid-19引起的X光图像
为此,将下载两个数据集:
数据集1:COVID-19的图像集
Joseph Paul Cohen和Paul Morrison和Lan Dao COVID-19图像数据收集,arXiv: 2003.11597, 2020
这是一个公开的COVID-19阳性和疑似患者和其他病毒性和细菌性肺炎(MERS、SARS和ARDS)的X光和ct图像数据集。
数据是从公共来源收集的,也可以从医院和医生处间接收集(项目由蒙特利尔大学伦理委员会批准,CERSES-20-058-D)。以下GitHub存储库中提供了所有图像和数据:https://github.com/ieee8023/covid-chestxray-dataset。
数据集2:肺炎和正常人的胸片
论文:Kermany, Daniel; Zhang, Kang; Goldbaum, Michael (2018), “Labeled Optical Coherence Tomography (OCT) and Chest X-Ray Images for Classification”
通过深度学习过程,将一组经验证的图像(CT和胸片)归类为正常和某些肺炎类型。图像分为训练集和独立的患者测试集。数据可在网站上获得:https://data.mendeley.com/datasets/rscbjbr9sj/2
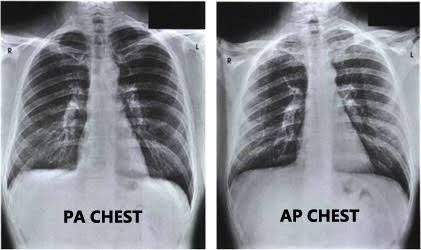
胸片的类型
从数据集中,可以找到三种类型的图像,PA,AP和Lateral(L)。L的很明显,但X光的AP和PA视图有什么区别?简单地说,在拍X光片的过程中,当X光片从身体的后部传到前部时,称为PA(后-前)视图。在AP视图中,方向相反。
通常,X光片是在AP视图中拍摄的。但是一个重要的例外就是胸部X光片,在这种情况下,最好在查看PA而不是AP。但如果病人病得很重,不能保持姿势,可以拍AP型胸片。
由于绝大多数胸部X光片都是PA型视图,所以这是用于训练模型的视图选择类型。
定义用于训练DL模型的环境
理想的做法是从一个新的Python环境开始。为此,使用Terminal定义一个工作目录(例如:X-Ray_Covid_development),然后在那里用Python创建一个环境(例如:TF_2_Py_3_7):
mkdir X-Ray_Covid_development
cd X-Ray_Covid_development
conda create — name TF_2_Py_3_7 python=3.7 -y
conda activate TF_2_Py_3_7进入环境后,安装TensorFlow 2.0:
pip install — upgrade pip
pip install tensorflow从这里开始,安装训练模型所需的其他库。例如:
conda install -c anaconda numpy
conda install -c anaconda pandas
conda install -c anaconda scikit-learn
conda install -c conda-forge matplotlib
conda install -c anaconda pillow
conda install -c conda-forge opencv
conda install -c conda-forge imutils创建必要的子目录:
notebooks
10_dataset —
|_ covid [here goes the dataset for training model 1]
|_ normal [here goes the dataset for training model 1]
20_dataset —
|_ covid [here goes the dataset for training model 2]
|_ pneumo [here goes the dataset for training model 2]
input -
|_ 10_Covid_Imagens _
| |_ [metadata.csv goes here]
| |_ images [Covid-19 images go here]
|_ 20_Chest_Xray -
|_ test _
|_ NORMAL [images go here]
|_ PNEUMONIA [images go here]
|_ train _
|_ NORMAL [images go here]
|_ PNEUMONIA [images go here]
model
dataset_validation _
|_ covid_validation [images go here]
|_ non_covidcovid_validation [images go here]
|_ normal_validation [images go here]数据下载
下载数据集1(Covid-19),并将metadata.csv文件保存在/input/10_Covid_Images/和/input/10_Covid_Images/Images/下。
下载数据集2(肺炎和正常),并将图像保存在/input/20_Chest_Xray/下(保持原始测试和训练结构)。
第2部分-模型1-Covid/正常
数据准备
从GitHub下载Notebook:https://github.com/Mjrovai/covid19Xray/blob/master/10_X-Ray_Covid_development/notebooks/10_Xray_Normal_Covid19_Model_1_Training_Tests.ipynb。将其存储在 subdirectory /notebooks中。
进入Notebook后,导入库并运行支持函数。
构建Covid标签数据集
从输入数据集(/input/10_Covid_Images/)创建用于训练模型1的数据集,该数据集将用于Covid和normal(正常)标签定义的图像分类。
input_dataset_path = ‘../input/10_Covid_images’metadata.csv文件将提供有关/images/文件中的图像的信息。
csvPath = os.path.sep.join([input_dataset_path, “metadata.csv”])
df = pd.read_csv(csvPath)
df.shapemetadat.csv文件有354行28列,这意味着在subdirectory /notebooks中有354个X光图像。让我们分析它的一些列,了解这些图像的更多细节。
通过df.modality,共有310张X光图像和44张CT图像。CT图像被丢弃。
COVID-19 235
Streptococcus 17
SARS 16
Pneumocystis 15
COVID-19, ARDS 12
E.Coli 4
ARDS 4
No Finding 2
Chlamydophila 2
Legionella 2
Klebsiella 1从可视化角度看,COVID-19的235张确认图像,我们有:
PA 142
AP 39
AP Supine 33
L 20
AP semi erect 1如引言中所述,只有142张PA型图像(后-前)用于模型训练,因为它们是胸片中最常见的图像(最终数据框:xray_cv)。
“xray_cv.patiendid”列显示,这142张照片属于96个病人,这意味着在某些情况下,同一个病人拍摄了多张X光片。由于所有图像都用于训练(我们对图像的内容感兴趣),因此不考虑此信息。
根据xray_cv.date,2020年3月拍摄的最新照片有8张。这些图像被分离在一个列表中,从模型训练中删除。 因此,以后将用作最终模型的验证。
imgs_march = [
‘2966893D-5DDF-4B68–9E2B-4979D5956C8E.jpeg’,
‘6C94A287-C059–46A0–8600-AFB95F4727B7.jpeg’,
‘F2DE909F-E19C-4900–92F5–8F435B031AC6.jpeg’,
‘F4341CE7–73C9–45C6–99C8–8567A5484B63.jpeg’,
‘E63574A7–4188–4C8D-8D17–9D67A18A1AFA.jpeg’,
‘31BA3780–2323–493F-8AED-62081B9C383B.jpeg’,
‘7C69C012–7479–493F-8722-ABC29C60A2DD.jpeg’,
‘B2D20576–00B7–4519-A415–72DE29C90C34.jpeg’
]下一步将构建指向训练数据集(xray_cv_train)的数据框,该数据框应引用134个图像(来自Covid的所有输入图像,用于稍后验证的图像除外):
xray_cv_train = xray_cv[~xray_cv.filename.isin(imgs_march)]
xray_cv_train.reset_index(drop=True, inplace=True)而最终的验证集(xray_cv_val )有8个图像:
xray_cv_val = xray_cv[xray_cv.filename.isin(imgs_march)]
xray_cv_val.reset_index(drop=True, inplace=True)为COVID训练图像创建文件
要记住,在前一项中,只有数据框是使用从原始文件metada.csv中获取的信息创建的。我们知道哪些图像要存储在最终的训练文件中,现在我们需要“物理地”将实际图像(以数字化格式)分离到正确的子目录(文件夹)中。
为此,我们将使用load_image_folder support()函数,该函数将元数据文件中引用的图像从一个文件复制到另一个文件:
def load_image_folder(df_metadata,
col_img_name,
input_dataset_path,
output_dataset_path):
img_number = 0
# 对COVID-19的行进行循环
for (i, row) in df_metadata.iterrows():
imagePath = os.path.sep.join([input_dataset_path, row[col_img_name]])
if not os.path.exists(imagePath):
print('image not found')
continue
filename = row[col_img_name].split(os.path.sep)[-1]
outputPath = os.path.sep.join([f"{output_dataset_path}", filename])
shutil.copy2(imagePath, outputPath)
img_number += 1
print('{} selected Images on folder {}:'.format(img_number, output_dataset_path))按照以下说明,134个选定图像将被复制到文件夹../10_dataset/covid/。
input_dataset_path = '../input/10_Covid_images/images'
output_dataset_path = '../dataset/covid'
dataset = xray_cv_train
col_img_name = 'filename'
load_image_folder(dataset, col_img_name,
input_dataset_path, output_dataset_path)为正常图像创建文件夹
对于数据集2(正常和肺炎图像),不提供包含元数据的文件。因此,你只需将图像从输入文件复制到末尾。为此,我们创建load_image_folder_direct()函数,该函数将许多图像(随机选择)从une文件夹复制到另一个文件夹:
def load_image_folder_direct(input_dataset_path,
output_dataset_path,
img_num_select):
img_number = 0
pathlist = Path(input_dataset_path).glob('**/*.*')
nof_samples = img_num_select
rc = []
for k, path in enumerate(pathlist):
if k < nof_samples:
rc.append(str(path)) # 路径不是字符串形式
shutil.copy2(path, output_dataset_path)
img_number += 1
else:
i = random.randint(0, k)
if i < nof_samples:
rc[i] = str(path)
print('{} selected Images on folder {}:'.format(img_number, output_dataset_path))在../input/20_Chest_Xray/train/NORMAL文件夹重复同样的过程,我们将随机复制用于训练的相同数量的图像 (len (xray_cv_train)),134个图像。这样,用于训练模型的数据集是平衡的。
input_dataset_path = '../input/20_Chest_Xray/train/NORMAL'
output_dataset_path = '../dataset/normal'
img_num_select = len(xray_cv_train)
load_image_folder_direct(input_dataset_path, output_dataset_path,
img_num_select)以同样的方式,我们分离出20个随机图像,以供以后在模型验证中使用。
input_dataset_path = '../input/20_Chest_Xray/train/NORMAL'
output_dataset_path = '../dataset_validation/normal_validation'
img_num_select = 20
load_image_folder_direct(input_dataset_path, output_dataset_path,
img_num_select)尽管我们没有用显示肺炎症状的图像(Covid-19)来训练模型,但是观察最终模型如何与肺炎症状一起工作是很有趣的。因此,我们还分离了其中的20幅图像,以供验证。
input_dataset_path = '../input/20_Chest_Xray/train/PNEUMONIA'
output_dataset_path = '../dataset_validation/non_covid_pneumonia_validation'
img_num_select = 20
load_image_folder_direct(input_dataset_path, output_dataset_path,
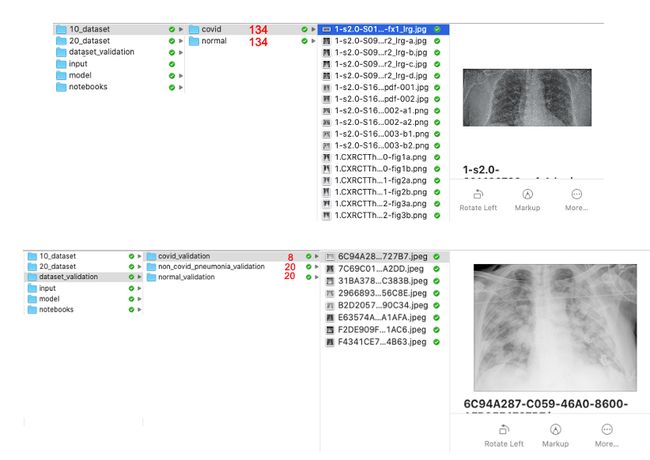
img_num_select)下面的图片显示了在这个步骤结束时应该如何配置文件夹(无论如何在我的Mac上)。此外,红色标记的数字显示文件夹中包含的x光图像的相应数量。
绘制数据集
由于文件夹中的图像数量不多,因此可以对其进行可视化检查。为此,使用 plots_from_files():
def plots_from_files(imspaths,
figsize=(10, 5),
rows=1,
titles=None,
maintitle=None):
"""Plot the images in a grid"""
f = plt.figure(figsize=figsize)
if maintitle is not None:
plt.suptitle(maintitle, fontsize=10)
for i in range(len(imspaths)):
sp = f.add_subplot(rows, ceildiv(len(imspaths), rows), i + 1)
sp.axis('Off')
if titles is not None:
sp.set_title(titles[i], fontsize=16)
img = plt.imread(imspaths[i])
plt.imshow(img)
def ceildiv(a, b):
return -(-a // b)然后,定义将在训练中使用的数据集的路径和带有要查看的图像名称的列表:
dataset_path = '../10_dataset'
normal_images = list(paths.list_images(f"{dataset_path}/normal"))
covid_images = list(paths.list_images(f"{dataset_path}/covid"))通过调用可视化支持函数,可以显示以下图像:
plots_from_files(covid_images, rows=10, maintitle="Covid-19 X-ray images")plots_from_files(normal_images, rows=10, maintitle="Normal X-ray images")图像看起来不错。
预训练的卷积神经网络模型
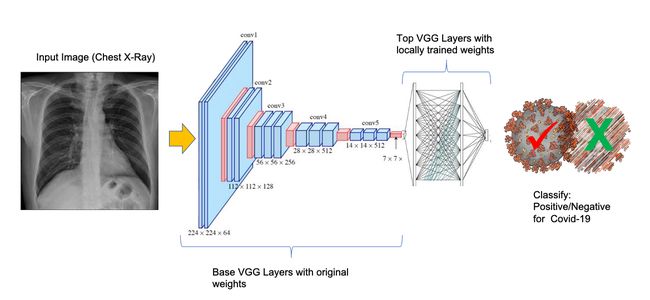
该模型的训练是使用预定义的图像进行的,应用了称为“迁移学习”的技术。
迁移学习是一种机器学习方法,其中为一个任务开发的模型被重用为第二个任务中模型的起点。
Keras应用程序是Keras的深度学习库模块,它为几种流行的架构(如VGG16、ResNet50v2、ResNet101v2、Xception、MobileNet等)提供模型定义和预训练的权重。
使用的预训练模型是VGG16,由牛津大学视觉图形组(VGG)开发,并在论文“Very Deep Convolutional Networks for Large-Scale Image Recognition”中描述。除了在开发公共的图像分类模型时非常流行外,这也是Adrian博士在其教程中建议的模型。
理想的做法是使用几个模型(例如ResNet50v2、ResNet101v2)进行测试(基准测试),甚至创建一个特定的模型(如Zhang等人在论文中建议的模型:https://arxiv.org/pdf/2003.12338.pdf)。但由于这项工作的最终目标只是概念上的验证,所以我们仅探索VGG16。
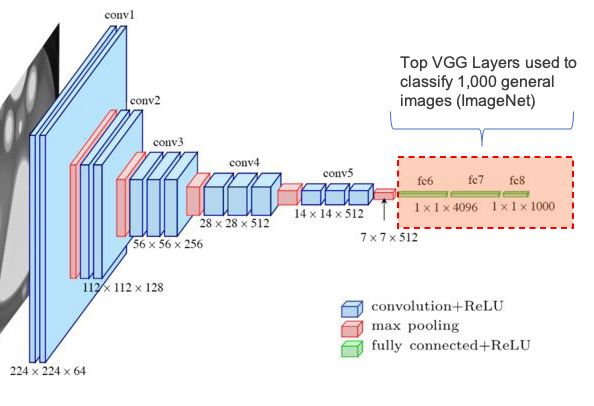
VGG16是一种卷积神经网络(CNN)体系结构,尽管在2014年已经开发出来,但今天仍然被认为是处理图像分类的最佳体系结构之一。
VGG16体系结构的一个特点是,它们没有大量的超参数,而是专注于卷积层上,卷积层上有一个3x3滤波器(核)和一个2x2最大池层。在整个体系结构中始终保持一组卷积和最大池化层。最后,该架构有2个FC(完全连接的层)和softmax激活输出。
VGG16中的16表示架构中有16个带权重的层。该网络是巨大的,在使用所有原始16层的情况下,有将近1.4亿个训练参数。
在我们的例子中,最后两层(FC1和FC2)是在本地训练的,参数总数超过1500万,其中大约有590000个参数是在本地训练的(而其余的是“frozen(冻结的)”)。
需要注意的第一点是,VNN16架构的第一层使用224x224x3的图像,因此我们必须确保要训练的X光图像也具有这些维度,因为它们是卷积网络的“第一层”的一部分。因此,当使用原始权重(weights=“imagenet”)加载模型时,我们不保留模型的顶层(include_top=False),而这些层将被我们的层(headModel)替换。
baseModel = VGG16(weights="imagenet", include_top=False, input_tensor=Input(shape=(224, 224, 3)))接下来,我们必须定义用于训练的超参数(在下面的注释中,将测试一些可能的值以提高模型的“准确性”):
INIT_LR = 1e-3 # [0.0001]
EPOCHS = 10 # [20]
BS = 8 # [16, 32]
NODES_DENSE0 = 64 # [128]
DROPOUT = 0.5 # [0.0, 0.1, 0.2, 0.3, 0.4, 0.5]
MAXPOOL_SIZE = (4, 4) # [(2,2) , (3,3)]
ROTATION_DEG = 15 # [10]
SPLIT = 0.2 # [0.1]然后构建我们的模型,将其添加到基础模型中:
headModel = baseModel.output
headModel = AveragePooling2D(pool_size=MAXPOOL_SIZE)(headModel)
headModel = Flatten(name="flatten")(headModel)
headModel = Dense(NODES_DENSE0, activation="relu")(headModel)
headModel = Dropout(DROPOUT)(headModel)
headModel = Dense(2, activation="softmax")(headModel)headModel被放置在基础模型之上,成为模型的一部分,进行实际训练(确定最佳权重)。
model = Model(inputs=baseModel.input, outputs=headModel)重要的是要记住一个预训练过的CNN模型,如VGG16,被训练了成千上万的图像来分类普通图像(如狗、猫、汽车和人)。我们现在需要做的是根据我们的需要定制它(分类X光图像)。理论上,模型的第一层简化了图像的部分,识别出其中的形状。这些初始标签非常通用(如直线、圆和正方形),因此我们不用再训练它们。我们只想训练网络的最后一层。
下面的循环在基础模型的所有层上执行,“冻结”它们,以便在第一个训练过程中不会更新它们。
for layer in baseModel.layers:
layer.trainable = False此时,模型已经准备好接受训练,但首先,我们必须为模型的训练准备数据(图像)。
数据预处理
我们先创建一个包含存储图像的名称(和路径)的列表:
imagePaths = list(paths.list_images(dataset_path))那么对于列表中的每个图像,我们必须:
提取图像标签(在本例中为covid或normal)
将BGR(CV2默认值)的图像通道设置为RGB
将图像大小调整为224x 224(VGG16的默认值)
data = []
labels = []
for imagePath in imagePaths:
label = imagePath.split(os.path.sep)[-2]
image = cv2.imread(imagePath)
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
image = cv2.resize(image, (224, 224))
data.append(image)
labels.append(label)数据和标签被转换成数组,每个像素的值从0到255改成从0到1,便于训练。
data = np.array(data) / 255.0
labels = np.array(labels)标签将使用一个one-hot编码技术进行数字编码。
lb = LabelBinarizer()
labels = lb.fit_transform(labels)
labels = to_categorical(labels)此时,训练数据集分为训练集和测试集(训练80%,测试20%):
(trainX, testX, trainY, testY) = train_test_split(data,
labels,
test_size=SPLIT,
stratify=labels,
random_state=42)最后但并非最不重要的是,我们应该应用“数据增强”技术。
数据增强
如Chowdhury等人所建议。在他们的论文中,三种增强策略(旋转、缩放和平移)可用于为COVID-19生成额外的训练图像,有助于防止“过度拟合”:https://arxiv.org/pdf/2003.13145.pdf。
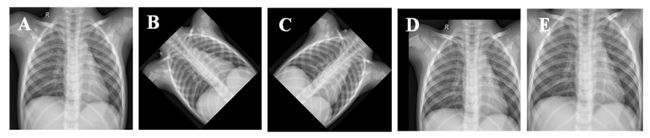
原始胸部X光图像(A),逆时针旋转45度后图像(B),顺时针旋转45度后图像,水平和垂直平移20%后图像(D),放大10%后图像(E)。
使用TS/Keras图像预处理库(ImageDataGenerator),可以更改多个图像参数,例如:
trainAug = ImageDataGenerator(
rotation_range=15,
width_shift_range=0.2,
height_shift_range=0.2,
rescale=1./255,
shear_range=0.2,
zoom_range=0.2,
horizontal_flip=True,
fill_mode='nearest')一开始,仅应用图像最大旋转15度来评估结果。
trainAug = ImageDataGenerator(rotation_range=ROTATION_DEG, fill_mode="nearest")此时,我们已经定义了模型和数据,并准备好进行编译和训练。
模型构建与训练
编译允许我们给模型添加额外的特性,比如loss函数、优化器和度量。
对于网络训练,我们使用损失函数来计算网络预测值与训练数据实际值之间的差异。伴随着优化器算法(如Adam)对网络中的权重进行更改。这些超参数有助于网络训练的收敛,使损失值尽可能接近于零。
我们还指定了优化器(lr)的学习率。在这种情况下,lr被定义为1e-3。如果在训练过程中注意到“跳跃”的增加,即模型不能收敛,则应降低学习率,以达到最小值。
opt = Adam(lr=INIT_LR, decay=INIT_LR / EPOCHS)
model.compile(loss="binary_crossentropy", optimizer=opt, metrics=["accuracy"])让我们来训练模型:
H = model.fit(
trainAug.flow(trainX, trainY, batch_size=BS),
steps_per_epoch=len(trainX) // BS,
validation_data=(testX, testY),
validation_steps=len(testX) // BS,
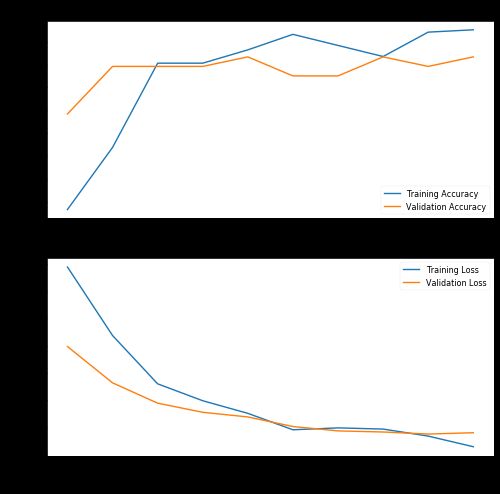
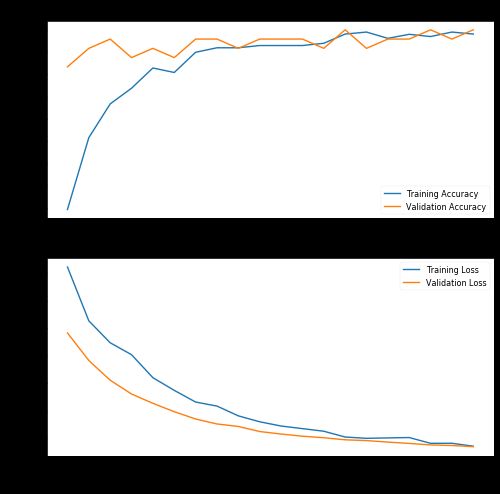
epochs=EPOCHS)结果看起来已经相当有趣了,验证数据的精度达到了92%!绘制精度图表:
评估训练模型:
看看混淆矩阵:
[[27 0]
[ 4 23]]
acc: 0.9259
sensitivity: 1.0000
specificity: 0.8519从用最初选择的超参数训练的模型中,我们得到:
- 100%的sensitivity(敏感度),也就是说,对于COVID-19阳性(即真正例)的患者,我们可以在100%的时间内准确地将其识别为“COVID-19阳性”。
- 85%的specificity(特异性)意味着在没有COVID-19(即真反例)的患者中,我们只能在85%的时间内准确地将其识别为“COVID-19阴性”。
结果并不令人满意,因为15%没有Covid的患者会被误诊。我们先对模型进行微调,更改一些超参数:
因此,我们有:
INIT_LR = 0.0001 # 曾经是 1e-3
EPOCHS = 20 # 曾经是 10
BS = 16 # 曾经是 8
NODES_DENSE0 = 128 # 曾经是 64
DROPOUT = 0.5
MAXPOOL_SIZE = (2, 2) # 曾经是 (4, 4)
ROTATION_DEG = 15
SPLIT = 0.2结果
precision recall f1-score support
covid 0.93 1.00 0.96 27
normal 1.00 0.93 0.96 27
accuracy 0.96 54
macro avg 0.97 0.96 0.96 54
weighted avg 0.97 0.96 0.96 54以及混淆矩阵:
[[27 0]
[ 2 25]]
acc: 0.9630
sensitivity: 1.0000
specificity: 0.9259结果好多了!现在具有93%的特异性,这意味着在没有COVID-19(即真反例)的患者中,在93%到100%的时间内我们可以准确地将他们识别为“COVID-19阴性”。
目前看来,这个结果很有希望。让我们保存这个模型,在那些没有经过训练的图像上测试(Covid-19的8个图像和从输入数据集中随机选择的20个图像)。
model.save("../model/covid_normal_model.h5")在真实图像中测试模型(验证)
首先,让我们检索模型并显示最终的体系结构,以检查一切是否正常:
new_model = load_model('../model/covid_normal_model.h5')
# 展示模型架构
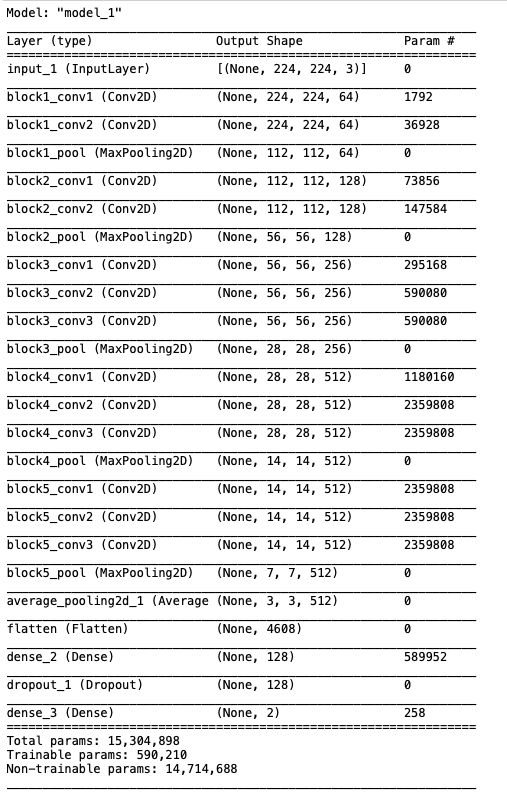
new_model.summary()这个模型看起来不错,是VGG16的16层结构。请注意,可训练参数为590210,这是最后两层的总和,它们被添加到参数为14.7M的预训练模型中。
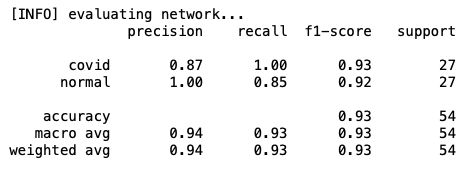
让我们验证测试数据集中加载的模型:
[INFO] evaluating network...
precision recall f1-score support
covid 0.93 1.00 0.96 27
normal 1.00 0.93 0.96 27
accuracy 0.96 54
macro avg 0.97 0.96 0.96 54
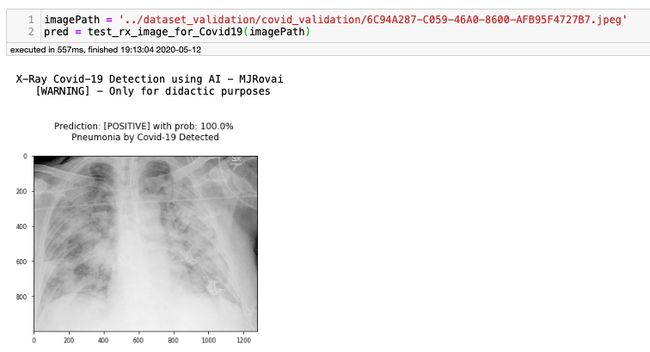
weighted avg 0.97 0.96 0.96 54很好,我们得到了与之前相同的结果,这意味着训练的模型被正确地保存和加载。现在让我们用之前保存的8个Covid图像验证模型。为此,我们创建了另外一个函数,它是为单个图像测试开发的
def test_rx_image_for_Covid19(imagePath):
img = cv2.imread(imagePath)
img = cv2.cvtColor(img, cv2.COLOR_BGR2RGB)
img = cv2.resize(img, (224, 224))
img = np.expand_dims(img, axis=0)
img = np.array(img) / 255.0
pred = new_model.predict(img)
pred_neg = round(pred[0][1]*100)
pred_pos = round(pred[0][0]*100)
print('\n X-Ray Covid-19 Detection using AI - MJRovai')
print(' [WARNING] - Only for didactic purposes')
if np.argmax(pred, axis=1)[0] == 1:
plt.title('\nPrediction: [NEGATIVE] with prob: {}% \nNo Covid-19\n'.format(
pred_neg), fontsize=12)
else:
plt.title('\nPrediction: [POSITIVE] with prob: {}% \nPneumonia by Covid-19 Detected\n'.format(
pred_pos), fontsize=12)
img_out = plt.imread(imagePath)
plt.imshow(img_out)
plt.savefig('../Image_Prediction/Image_Prediction.png')
return pred_pos在Notebook上,此函数将显示以下结果:
通过更改其余7个图像的imagePath值,我们获得以下结果:
所有图像均呈阳性,确认100%灵敏度。
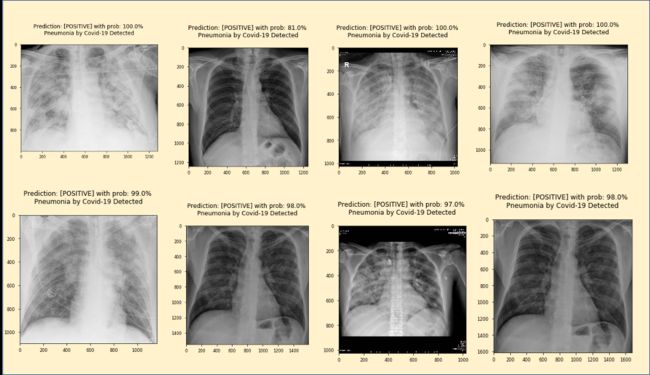
现在让我们测试20个单独的图像,以验证标记为NORMAL的有效性。Notebook上的第一个应该是:
一个接一个的测试可以确认预测,但是由于我们有更多的图像,让我们使用另一个函数来测试一组图像,一次完成: test_rx_image_for_Covid19_batch (img_lst) 。
批处理测试图像
让我们创建包含在验证文件夹中的图像的列表:
validation_path = '../dataset_validation'
normal_val_images = list(paths.list_images(
f"{validation_path}/normal_validation"))
non_covid_pneumonia_validation_images = list(paths.list_images(
f"{validation_path}/non_covid_pneumonia_validation"))
covid_val_images = list(paths.list_images(
f"{validation_path}/covid_validation"))test_rx_image_for_Covid19_batch (img_lst) 函数如下:
def test_rx_image_for_Covid19_batch(img_lst):
neg_cnt = 0
pos_cnt = 0
predictions_score = []
for img in img_lst:
pred, neg_cnt, pos_cnt = test_rx_image_for_Covid19_2(img, neg_cnt, pos_cnt)
predictions_score.append(pred)
print ('{} positive detected in a total of {} images'.format(pos_cnt, (pos_cnt+neg_cnt)))
return predictions_score, neg_cnt, pos_cnt将该函数应用于我们先前分离的20幅图像:
img_lst = normal_val_images
normal_predictions_score, normal_neg_cnt, normal_pos_cnt = test_rx_image_for_Covid19_batch(img_lst)
normal_predictions_score我们观察到,所有20人被诊断为阴性,得分如下(记住,接近“1”代表“阳性”):
0.25851375,
0.025379542,
0.005824779,
0.0047603976,
0.042225637,
0.025087152,
0.035508618,
0.009078974,
0.014746706,
0.06489486,
0.003134642,
0.004970203,
0.15801577,
0.006775451,
0.0032735346,
0.007105667,
0.001369465,
0.005155371,
0.029973848,
0.014993184只有2例图像的评估(1-准确度)低于90%(0.26和0.16)。
请记住,输入数据集/input/20_Chest_Xray/有两个文件夹,/train和/test。只有/train中的一部分图像用于训练,并且模型从未看到测试图像:
input -
|_ 10_Covid_Imagens _
| |_ metadata.csv
| |_ images [used train model 1]
|_ 20_Chest_Xray -
|_ test _
|_ NORMAL
|_ PNEUMONIA
|_ train _
|_ NORMAL [used train model 1]
|_ PNEUMONIA然后,我们可以利用这个文件夹测试所有图像。首先,我们创建了图像列表:
validation_path = '../input/20_Chest_Xray/test'
normal_test_val_images = list(paths.list_images(f"{validation_path}/NORMAL"))
print("Normal Xray Images: ", len(normal_test_val_images))
pneumo_test_val_images = list(paths.list_images(f"{validation_path}/PNEUMONIA"))
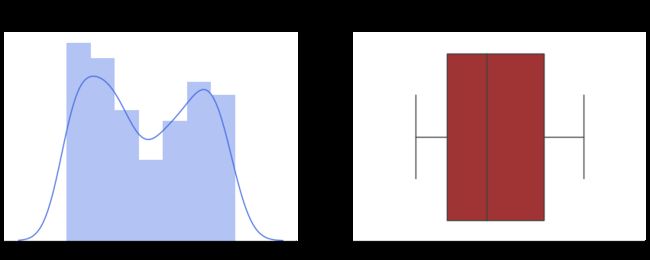
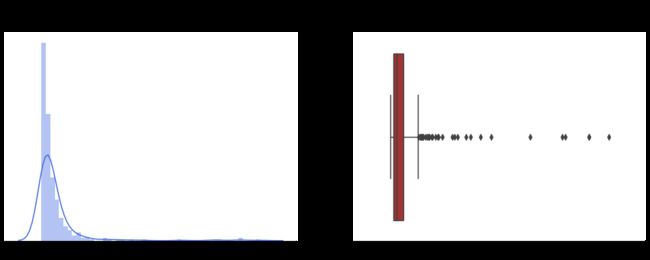
print("Pneumo Xray Images: ", len(pneumo_test_val_images))我们观察了234张诊断为正常的“未公开”图片(还有390张不是由Covid-19引起的肺炎)。应用批处理函数,我们观察到24幅图像出现假阳性(约10%)。让我们看看模型输出值是如何分布的,记住函数返回的值计算如下:
pred = new_model.predict(image)
pred_pos = round(pred[0][0] * 100)我们观察到,预测精度的平均值为0.15,并且非常集中于接近于零的值(中值仅为0.043)。有趣的是,大多数误报率接近0.5,少数异常值高于0.6。
除了改进模型外,还值得研究产生假阳性的图像。
测试不是由Covid引起的肺炎图像
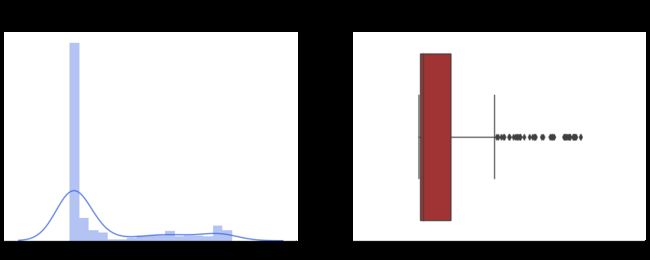
由于输入数据集也有肺炎患者的X光图像,但不是由Covid引起的,所以让我们应用模型1(Covid/Normal)来查看结果是什么:
结果非常糟糕,在390张图片中,185张有假阳性。而观察结果的分布,发现有一个峰值接近80%,也就是说,这是非常错误的!
回顾这一结果在技术上并不令人惊讶,因为该模型没有经过普通肺炎患者图像的训练。
不管怎样,这是一个大问题,因为我认为专家可以用肉眼区分病人是否患有肺炎。然而,也许更难区分这种肺炎是由Covid-19(SARS-CoV-2)、任何其他病毒,甚至是细菌引起的。
将Covid-19引起的肺炎患者与其他类型的病毒或细菌区分开来的模型更有 用。为此,另一个模型将被训练,现在有感染Covid-19的病人和感染肺炎但不是由Covid-19病毒引起的病人的图像。
第3部分-模型2-Covid/普通肺炎
数据准备
从我的GitHub下载Notebook放入subdirectory /notebooks目录:https://github.com/Mjrovai/covid19Xray/blob/master/10_X-Ray_Covid_development/notebooks/20_Xray_Pneumo_Covid19_Model_2_Training_Tests.ipynb。
导入使用的库并运行。
模型2中使用的Covid图像数据集与模型1中使用的相同,只是现在它存储在不同的文件夹中。
dataset_path = '../20_dataset'肺炎图像将从文件夹/input/20_Chest_Xray/train/PNEUMONIA/下载并存储在/20_dataset/pneumo/中。使用的函数与之前相同:
input_dataset_path = '../input/20_Chest_Xray/train/PNEUMONIA'
output_dataset_path = '../20_dataset/pneumo'
img_num_select = len(xray_cv_train) # 样本数量与Covid数据相同这样,我们调用可视化支持函数,检查得到的结果:
pneumo_images = list(paths.list_images(f"{dataset_path}/pneumo"))
covid_images = list(paths.list_images(f"{dataset_path}/covid"))
plots_from_files(covid_images, rows=10, maintitle="Covid-19 X-ray images")plots_from_files(pneumo_images, rows=10, maintitle="Pneumony X-ray images"图像看起来不错。
预训练CNN模型及其超参数的选择
要使用的预训练模型是VGG16,与模型1训练相同
baseModel = VGG16(weights="imagenet", include_top=False, input_tensor=Input(shape=(224, 224, 3)))接下来,我们必须定义用于训练的超参数。我们与模型1的参数相同:
INIT_LR = 0.0001
EPOCHS = 20
BS = 16
NODES_DENSE0 = 128
DROPOUT = 0.5
MAXPOOL_SIZE = (2, 2)
ROTATION_DEG = 15
SPLIT = 0.2然后,构建模型:
headModel = baseModel.output
headModel = AveragePooling2D(pool_size=MAXPOOL_SIZE)(headModel)
headModel = Flatten(name="flatten")(headModel)
headModel = Dense(NODES_DENSE0, activation="relu")(headModel)
headModel = Dropout(DROPOUT)(headModel)
headModel = Dense(2, activation="softmax")(headModel)将headModel模型放在最后,成为用于训练的真实模型。
model = Model(inputs=baseModel.input, outputs=headModel)在基础模型的所有层上执行的以下循环将“冻结”它们,以便在第一个训练过程中不会更新它们。
for layer in baseModel.layers:
layer.trainable = False此时,模型已经准备好接受训练,但是我们应该首先为模型准备数据(图像)。
数据预处理
我们先创建一个包含存储图像的名称(和路径)的列表,然后执行与模型1相同的预处理:
imagePaths = list(paths.list_images(dataset_path))
data = []
labels = []
for imagePath in imagePaths:
label = imagePath.split(os.path.sep)[-2]
image = cv2.imread(imagePath)
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
image = cv2.resize(image, (224, 224))
data.append(image)
labels.append(label)
data = np.array(data) / 255.0
labels = np.array(labels)标签使用one-hot。
lb = LabelBinarizer()
labels = lb.fit_transform(labels)
labels = to_categorical(labels)此时,我们将把训练数据集分为训练和测试(80%用于训练,20%用于测试):
(trainX, testX, trainY, testY) = train_test_split(data,
labels,
test_size=SPLIT,
stratify=labels,
random_state=42)最后,我们将应用数据增强技术。
trainAug = ImageDataGenerator(rotation_range=ROTATION_DEG, fill_mode="nearest")我们已经定义了模型和数据,并准备好进行编译和训练。
模式2的编译和训练
编译:
opt = Adam(lr=INIT_LR, decay=INIT_LR / EPOCHS)
model.compile(loss="binary_crossentropy", optimizer=opt, metrics=["accuracy"])训练:
H = model.fit(
trainAug.flow(trainX, trainY, batch_size=BS),
steps_per_epoch=len(trainX) // BS,
validation_data=(testX, testY),
validation_steps=len(testX) // BS,
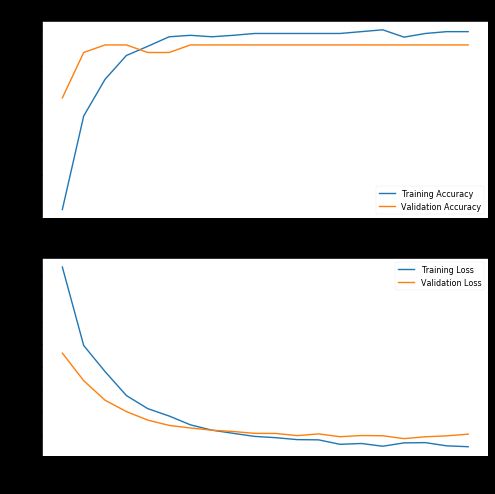
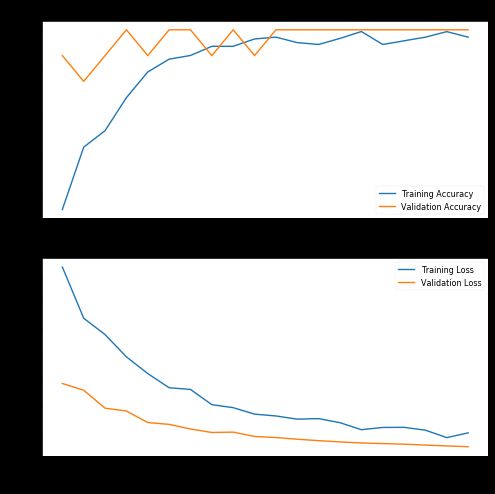
epochs=EPOCHS)使用20个阶段和初始参数,结果看起来非常有趣,验证数据的精度达到100%!让我们绘制精度图表,评估训练的模型,并查看混淆矩阵:
precision recall f1-score support
covid 0.96 1.00 0.98 27
pneumo 1.00 0.96 0.98 27
accuracy 0.98 54
macro avg 0.98 0.98 0.98 54
weighted avg 0.98 0.98 0.98 54混淆矩阵
[[27 0]
[ 1 26]]
acc: 0.9815
sensitivity: 1.0000
specificity: 0.9630通过训练模型(初始选择超参数),我们得到:
- 100%敏感度,也就是说,对于COVID-19阳性(即真正例)的患者,我们可以在100%的时间内准确地确定他们为“COVID-19阳性”。
- 96%特异性,也就是说,在没有COVID-19(即真反例)的患者中,我们可以在96%的时间内准确地将其识别为“COVID-19阴性”。
结果完全令人满意,因为只有4%的患者没有Covid会被误诊。但与本例一样,肺炎患者和Covid-19患者之间的正确分类是最有益处的;我们至少应该对超参数进行一些调整,再次进行训练。
第一件事,我试图降低最初的lr一点点,这是一场灾难。所以我恢复了原值。
我还减少了数据的分割,稍微增加了Covid图像,并将最大旋转角度更改为10度,这是在与原始数据集相关的论文中建议的:
INIT_LR = 0.0001
EPOCHS = 20
BS = 16
NODES_DENSE0 = 128
DROPOUT = 0.5
MAXPOOL_SIZE = (2, 2)
ROTATION_DEG = 10
SPLIT = 0.1因此,我们有:
precision recall f1-score support
covid 1.00 1.00 1.00 13
pneumo 1.00 1.00 1.00 14
accuracy 1.00 27
macro avg 1.00 1.00 1.00 27
weighted avg 1.00 1.00 1.00 27以及混淆矩阵:
[[13 0]
[ 0 14]]
acc: 1.0000
sensitivity: 1.0000
specificity: 1.0000结果看起来更好,但我们使用了很少的测试数据!让我们保存模型,并像以前一样用大量图像对其进行测试。
model.save("../model/covid_pneumo_model.h5")我们观察到390张标记为非Covid-19引起的肺炎的图像。应用批测试功能,我们发现总共390张图片中只有3张出现假阳性(约0.8%)。此外,预测精度值的平均值为0.04,并且非常集中于接近于零的值(中值仅为0.02)。
总的结果甚至比以前的模型所观察到的还要好。有趣的是,几乎所有的结果都在前3个四分位之内,只有很少的异常值有超过20%的误差。
在这种情况下,还值得研究产生假阳性的图像(仅3幅)。
用正常(健康)患者的图像进行测试
由于输入数据集也有正常患者(未经训练)的X光图像,让我们应用模型2(Covid/普通肺炎)看看结果如何
在这种情况下,结果并没有模型1测试中看到的那么糟糕,在234幅图像中,有45幅出现了假阳性(19%)。
好吧,理想情况是对每种情况使用正确的模型,但是如果只使用一种,那么模型2是正确的选择。
注:在最后一次测试中,我做了一个基准测试,我尝试改变增强参数,正如Chowdhury等人所建议的,令我惊讶的是,结果并不好。
第4部分-Web应用程序
测试Python独立脚本
对于web应用的开发,我们使用Flask,这是一个用Python编写的web微框架。它被归类为微框架,因为它不需要特定的工具或库来运行。
此外,我们只需要几个库和与单独测试图像相关的函数。所以,让我们首先在一个干净的Notebook上工作,在那里使用已经训练和保存的模型2执行测试。
从我的GitHub加载:https://github.com/Mjrovai/covid19Xray/blob/master/10_X-Ray_Covid_development/notebooks/30_AI_Xray_Covid19_Pneumo_Detection_Application.ipynb
现在只导入测试前一个Notebook中创建的模型所需的库。
import numpy as np
import cv2
from tensorflow.keras.models import load_model- 然后执行加载和测试图像的函数:
def test_rx_image_for_Covid19_2(model, imagePath):
img = cv2.imread(imagePath)
img_out = img
img = cv2.cvtColor(img, cv2.COLOR_BGR2RGB)
img = cv2.resize(img, (224, 224))
img = np.expand_dims(img, axis=0)
img = np.array(img) / 255.0
pred = model.predict(img)
pred_neg = round(pred[0][1]*100)
pred_pos = round(pred[0][0]*100)
if np.argmax(pred, axis=1)[0] == 1:
prediction = 'NEGATIVE'
prob = pred_neg
else:
prediction = 'POSITIVE'
prob = pred_pos
cv2.imwrite('../Image_Prediction/Image_Prediction.png', img_out)
return prediction, prob- 下载训练模型
covid_pneumo_model = load_model('../model/covid_pneumo_model.h5')然后,从上传一些图像,并确认一切正常:
imagePath = '../dataset_validation/covid_validation/6C94A287-C059-46A0-8600-AFB95F4727B7.jpeg'
test_rx_image_for_Covid19_2(covid_pneumo_model, imagePath)结果是:(‘POSITIVE’, 96.0)
imagePath = '../dataset_validation/normal_validation/IM-0177–0001.jpeg'
test_rx_image_for_Covid19_2(covid_pneumo_model, imagePath)结果是: (‘NEGATIVE’, 99.0)
imagePath = '../dataset_validation/non_covid_pneumonia_validation/person63_bacteria_306.jpeg'
test_rx_image_for_Covid19_2(covid_pneumo_model, imagePath)结果是:(‘NEGATIVE’, 98.0)
到目前为止,所有的开发都是在Jupyter Notebook上完成的,我们应该做最后的测试,让代码作为python脚本运行在最初创建的开发目录中,名称为:covidXrayApp_test.py。
# 导入库和设置
import numpy as np
import cv2
from tensorflow.keras.models import load_model
# 关闭信息和警告
import os
os.environ['TF_CPP_MIN_LOG_LEVEL'] = '2'
def test_rx_image_for_Covid19_2(model, imagePath):
img = cv2.imread(imagePath)
img_out = img
img = cv2.cvtColor(img, cv2.COLOR_BGR2RGB)
img = cv2.resize(img, (224, 224))
img = np.expand_dims(img, axis=0)
img = np.array(img) / 255.0
pred = model.predict(img)
pred_neg = round(pred[0][1]*100)
pred_pos = round(pred[0][0]*100)
if np.argmax(pred, axis=1)[0] == 1:
prediction = 'NEGATIVE'
prob = pred_neg
else:
prediction = 'POSITIVE'
prob = pred_pos
cv2.imwrite('./Image_Prediction/Image_Prediction.png', img_out)
return prediction, prob
# 载入模型
covid_pneumo_model = load_model('./model/covid_pneumo_model.h5')
# ---------------------------------------------------------------
# 执行测试
imagePath = './dataset_validation/covid_validation/6C94A287-C059-46A0-8600-AFB95F4727B7.jpeg'
prediction, prob = test_rx_image_for_Covid19_2(covid_pneumo_model, imagePath)
print (prediction, prob)让我们直接在终端上测试脚本:
一切工作完美
创建在Flask中运行的环境
第一步是从一个新的Python环境开始。为此,使用Terminal定义一个工作目录(covid19XrayWebApp),然后在那里用Python创建一个环境
mkdir covid19XrayWebApp
cd covid19XrayWebApp
conda create --name covid19xraywebapp python=3.7.6 -y
conda activate covid19xraywebapp进入环境后,安装Flask和运行应用程序所需的所有库:
conda install -c anaconda flask
conda install -c anaconda requests
conda install -c anaconda numpy
conda install -c conda-forge matplotlib
conda install -c anaconda pillow
conda install -c conda-forge opencv
pip install --upgrade pip
pip install tensorflow
pip install gunicorn创建必要的子目录:
[here the app.py]
model [here the trained and saved model]
templates [here the .html file]
static _ [here the .css file and static images]
|_ xray_analysis [here the output image after analysis]
|_ xray_img [here the input x-ray image]从我的GitHub复制文件并将其存储在新创建的目录中
Githhub:https://github.com/Mjrovai/covid19Xray/tree/master/20_covid19XrayWebApp
执行以下步骤
在服务器上负责后端执行的python应用程序称为app.py,必须位于主目录的根目录下
在/template中,应该存储index.html文件,该文件将是应用程序的前端
在/static将是style.css文件,负责前端(template.html)的样式。
在/static下,还有接收待分析图像的子目录,以及分析结果(其原始名称以及诊断和准确性百分比)。
所有文件安装到正确的位置后,工作目录如下所示:
在本地网络上启动Web应用
将文件安装到文件夹中后,运行app.py,这是我们的web应用程序的“引擎”,负责接收存储在用户计算机的图像。
python app.py在终端我们可以观察到:
在浏览器上,输入:
http://127.0.0.1:5000/
应用程序将在你的本地网络中运行:
使用真实图像测试web应用程序
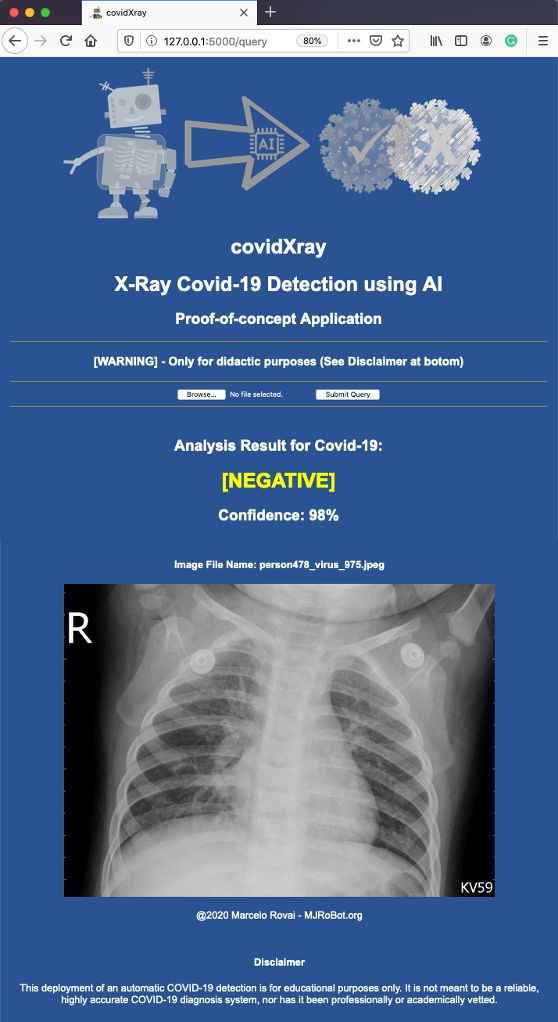
我们可以选择启动一个Covid的X光图像,它已经在开发过程中用于验证。
步骤顺序如下:
对其中一张有肺炎但没有Covid-19的图片重复测试:
建议
正如导言中所讨论的,本项目是一个概念证明,证明了在X光图像中检测Covid-19的病毒的可行性。要使项目在实际情况中使用,还必须完成几个步骤。以下是一些建议:
与健康领域的专业人员一起验证整个项目
寻找最佳的预训练模型
使用从患者身上获得的图像训练模型,患者最好是来自应用程序将要使用的同一区域。
使用Covid-19获取更广泛的患者图像集
改变模型的超参数
测试用3个类标(正常、Covid和肺炎)训练模型的可行性
更改应用程序,允许选择更适合使用的模型(模型1或模型2)
本文中使用的所有代码都可以Github仓库下载:https://github.com/Mjrovai/covid19Xray。
原文链接:https://towardsdatascience.com/applying-artificial-intelligence-techniques-in-the-development-of-a-web-app-for-the-detection-of-9225f0225b4
欢迎关注磐创AI博客站: http://panchuang.net/
sklearn机器学习中文官方文档: http://sklearn123.com/
欢迎关注磐创博客资源汇总站: http://docs.panchuang.net/