转录组背景知识
文章目录
- RNA-seq 相关概念
- RNA-Seq
- 转录组(transcriptome)
- RNA种类
- 可变剪切体(Alternative splicing isoform)
- junction reads
- Read count
- RPKM
- FPKM
- 安装软件
- 安装miniconda
- 添加channel
- 创建虚拟环境
- 查看虚拟环境
- 搜索bioconda镜像中的软件
- 安装相关软件
RNA-seq 相关概念
RNA-Seq
具体来说,首先对生物样品中的RNA反转录为cDNA,而后,将这些cDNA打碎为较小片段后,上机进行测序
转录组(transcriptome)
是指特定类型细胞中全体转录本(transcript)的集合
RNA种类
| RNA | explanation |
|---|---|
| coding RNA:mRNA | 信使RNA |
| noncoding RNA:rRNA | 核糖体RNA(ribosomalRNA) |
| noncoding RNA:tRNA | 转移RNA(transferRNA) |
| noncoding RNA:snRNA | 小核RNA(small nuclearRNA) |
| noncoding RNA:snoRNA | 最早在核仁发现的小RNA,称作小核仁RNA |
| noncoding RNA:asRNA | 反义antisense RNA 是一类能够与mRNA互补配对的单链RNA分子。细胞中引入反义RNA,可与mRNA发生互补配对,抑制mRNA的翻译 |
| noncoding RNA:lincRNA | 长链非编码RNA,长度大于200个核苷酸的一类非蛋白质编码转录物 |
| noncoding RNA:miRNA | microRNA,长度小于50nt |
| noncoding RNA:siRNA | Small interfering RNA,25nt左右的双链RNA |
| noncoding RNA:piRNA | 与Piwi蛋白相作用的RNA,长度小于50nt |
可变剪切体(Alternative splicing isoform)
junction reads
在DNA转录成mRNA的过程中,内含子被切掉,外显子会在剪切位点连接到一起。对于这些跨过剪切位点的reads,称为junction reads。如果不把它们从中间断开,就无法准确贴到基因组上。 这些junction reads是确定剪切位点的直接证据,对于正确重构转录本结构至关重要
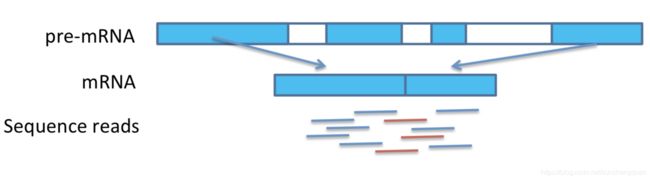
Read count
比对到gene A的reads数,(1)换算RPKM,FPKM等后续其他指标 (2)作为基因表达差异分析(DESeq,edgeR)的输入数据
RPKM
Reads Per Kilobase of exon model per Millon mapped reads
每百万条reads中每1000bp基因中reads的数量
R P K M = C L N RPKM = \frac {C} {LN} RPKM=LNC
C: 比对上的reads数(外显子,转录本)
L: 特征长度(1000bp)
N:比对上的总reads数(100万)
示意图:
 RPKM:消除基因长度和测序量差异对计算基因表达的影响 不适用:发生了可变剪切的RNA-seq数据
RPKM:消除基因长度和测序量差异对计算基因表达的影响 不适用:发生了可变剪切的RNA-seq数据
FPKM
F=Fragment,即测序片段的数量,比对上的一对reads为一个fragment
本质:以文库中的片段为计算单位,是在PE测序上对RPKM的矫正
FPKM表示:每一百万个map上的reads中map到外显子的每1k个碱基上的reads个数
F P K M = t o t a l e x o n F r a g m e n t s m a p p e d r e a d s ( M i l l i o n s ) × e x o n l e n g t h ( k b ) FPKM = \frac {total exon Fragments} {mapped reads(Millions) \times exon length(kb)} FPKM=mappedreads(Millions)×exonlength(kb)totalexonFragments
安装软件
安装miniconda
https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
添加channel
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/cloud/bioconda/
conda config --set show_channel_urls yes
#查看
conda config --show-sources
创建虚拟环境
conda create -n RNA
查看虚拟环境
(RNA) [sunchengquan 10:50:34 ~]
$ conda env list
# conda environments:
#
base /home/sunchengquan/local/app/miniconda3
R /home/sunchengquan/local/app/miniconda3/envs/R
RNA * /home/sunchengquan/local/app/miniconda3/envs/RNA
搜索bioconda镜像中的软件
http://bioconda.github.io/recipes.html#recipes
安装相关软件
conda install star hisat2 tophat bowtie2 bwa subread cutadapt trim-galore bedtools htseq deeptools trimmomatic fastqc samtools bcftools cufflinks
