MER:1.8万字带你系统了解宏组学实验与分析(高通量测序应用于病原体和害虫诊断——综述与实用性建议)...
高通量测序应用于病原体和害虫诊断——综述与实用性建议
High‐throughput identification and diagnostics of pathogens and pests: Overview and practical recommendations
译者:中国科学院动物研究所 和平鸽
责任编辑:中国科学院遗传与发育研究所 刘永鑫
DOI: https://doi.org/10.1111/1755-0998.12959
PDF: https://onlinelibrary.wiley.com/doi/pdf/10.1111/1755-0998.12959
译者的话
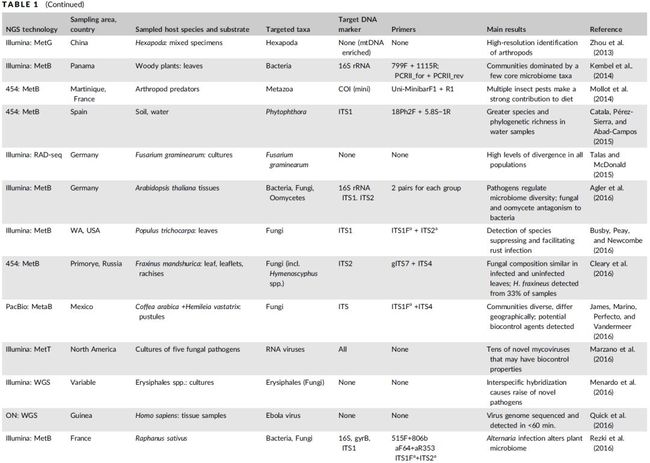
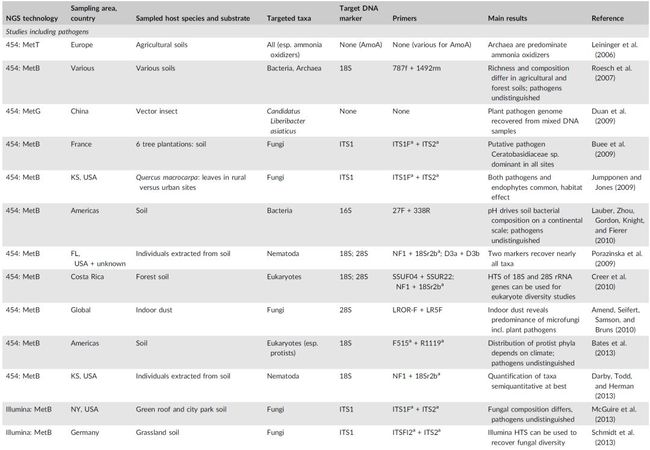
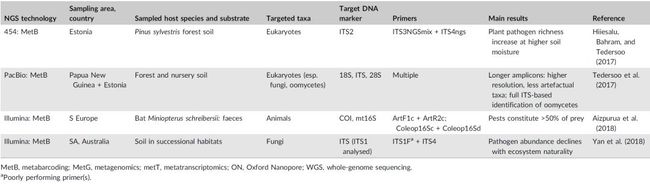
高通量测序技术(High-throughput Sequencing, HTS)在生物学领域的应用越来越广泛,此综述虽为其用于病原体和害虫诊断而著,但生物学其它研究领域,如生物进化、生物多样性与保护、动物食性与栖息地选择、动物营养学、动物行为学等多个领域均可借鉴此文归纳总结的方法和经验,尤其是在实验设计时参考其建议。图1展示了高通量测序总体实验与数据分析流程。表1总结了HTS应用于不同生物类群最具代表性的工作;表2对比了不同测序仪器的经费预算;表3总结了不同目标类群扩增子测序常用的引物标记。
摘要
基于高通量测序技术的鉴定手段为理解微生物的生态与功能提供了有效工具。虽然这些方法在农业、林业和植物群落动态研究中极为重要,然而它们到目前为止还很少被应用于检测和检验与植物病原体和害虫相关的生态假设的研究。这篇文章有以下目标:1. 综合回顾基于高通量的定量和分子鉴定技术在微生物种群动态、群落生态学和寄主相关性研究中的最新进展;2. 汇总以细菌、真菌、卵菌和昆虫类害虫为目标的具体实验方案和可用引物信息,并提供有用的实验设计建议;3. 提供在微生物相关学科得到应用,而又能够在检验病理学特定生物学假设中具有潜在应用价值新方法的案例。最后,作者对病原体和害虫相关的诊断、种群与群落水平生态学研究已经出现或正在出现的最新方法进行展望。
关键词
群落生态学,作物病害,森林病理学,高通量测序,宏分子标记,宏基因组学,分子诊断,植物病原体,前哨种植(sentinel plantations)
1 前言
1 | INTRODUCTION
植物的全球化和国际贸易大大加快了害虫和病原体侵入农林业系统的频率和程度,导致与植物宿主的新接触。在一些罕见的情况下,这些入侵的有害生物通过引起本地寄主物种的灭绝和古代文明的崩溃,导致了本地生态系统的大规模转变,并改变了生态动态。此外,气候变化促进了外来害虫和病原体的种群在新领地建立,并推进其现有种群扩张。植物园和早期预警系统代表着识别自然植物群落面临的新的和正在出现的风险以及改善全球监测的手段。
除了对包括人类在内的动植物造成经济损失和疾病外,在自然生态系统中,病原体和害虫在维持农业多样性和初级生产力方面发挥着关键作用。这种Janzen Connell假说(同种个体或者异种个体所在空间距离越近,对资源的竞争也就越激烈。与此同时,相同种在空间上的聚集,容易导致疾病传播,容易被捕食或者取食)主要通过六足动物的植食性或微生物病原体导致根系腐烂而发生,这些六足动物或微生物病原体专门针对优势植物物种,并在苗期选择性地增加它们的死亡率。
传统的研究中,包括病原体在内的微生物是根据疾病症状或作物的形态来识别的,而害虫的检测通常依赖于具有代表性个体的形态特征。许多专性细胞内病原体不能在纯培养中生长,也不能形成生殖结构,这使得它们的检测和鉴定非常困难。此外,微生物病原体和动物害虫都可能表现出高度的种内变异,或包含在生态位和侵略性上可能有很大差异的隐存物种 (cryptic species)。在生物物种中,基因型或种族不同,其致病性也可能不同,有时取决于副致病性位点(accessory pathogenicity loci)和染色体的存在。这些种间和种内的差异强调了在物种和致病类型(pathotypes)或菌株(strains)水平上精确检测生物体的重要性。
迅速和准确地识别病原微生物和害虫是发现和采取适当减灾对策的必要条件。自20世纪90年代初以来,分子方法给我们对微生物物种身份和起源的认识带来了一场革命,并使我们对微生物群落(包括病原体)的种群结构和群落组成有了更清晰的认识。此前,已有一些综述概括了早期分子技术在害虫鉴定、生物防治和微生物病原鉴定中的应用。关于近期高通量测序方法和分析方案的信息则较为分散,较多强调的是总体概念、遗传标记、具体方法或者是在具体某个类群,如植物、动物、昆虫、真菌、细菌的应用。除了病毒,只有少数人专注于高通量鉴定原核生物、原生生物和真菌,也很少有来自病原体的例子。
这篇文章的目的是提供关于植物病原体和害虫识别前沿分子方法的概述,重点是在物种水平上的高通量识别,以及在适当的时候探讨基因型和种群水平的诊断方法。基于研究中可能遭遇的陷阱与经验积累,我们为从取样设计到分子分析、生物信息学分析、分类学和功能预测以及统计学的分析工作流程提供实用的建议。以病虫害为例,阐述了最新的分析步骤和主要成果。最后,我们提供了用于监测和有效诊断害虫和病原体种群和物种水平分析的综合展望。
2 新兴方法
2 | THE EMERGING METHODS
在过去的15年里,研究人员利用高通量分子鉴定方法的快速发展来发掘地表和地下微生物生命的巨大多样性。这些方法能够同时从数百个样本中识别每个样本中的数千个分类单元,并便于同时研究任何生物体和病毒组。在技术方面,高通量识别方法可分为基于PCR的量化方法、基于杂交的方法(如基因芯片)、基于第二代指纹识别方法(如RADseq),和基于序列的方法:宏条形码(如metabarcoding)、宏基因组学(getagenomics)和宏转录组(metatranscriptomics)。表1总结了这些方法的第一个最有影响力的研究案例,以及它们在病原体和害虫中的应用。
2.1 定量方法
2.1 | Quantification methods
类群特异性引物和定量PCR (qPCR)方法被用于测定病原微生物的相对丰度和/或绝对丰度已有20年的历史。最近的技术进步使并行运行数千个样本和模板组合成为可能。例如,Muurinen等人(2017)同时对数百个抗生素耐药基因进行了重复筛选。 液滴数字PCR (Droplet digital PCR, ddPCR)是基于微流体技术,将扩增反应分离为>20 000个个体的液滴,允许同时对多达4个目标生物或基因的DNA进行绝对定量,检测限为10-5相对丰富。Dreo等(2014)用最优引物和次优引物对两种植物病原菌进行ddPCR定量,其准确度比普通qPCR高得多。目前,ddPCR可以在96孔和384孔板中运行,但在技术上可以通过增加样品的通量,也可以增加荧光染料的用量,使之能够完成多重反应(>4)。
通过spiking方法结合HTS鉴定方法,可以对标记物或功能基因进行定量。 对于spiking,在获取DNA/RNA和目标生物体的量之前,将已知量的对照DNA或个体添加到样本中,目标生物或者基因是根据获得的序列相对量来检测的。理论上,spiking允许对任何致病生物或基因的DNA标记物含量进行定量,但这种方法迄今为止还很少得到使用。序列长度、碱基组成、DNA二级结构等方面的差异都可能影响spiking方法的准确性。
2.2 基因芯片
2.2 | Microarrays
基因芯片技术基于将多个目标‐模板杂交反应预先锚定到小芯片上,利用机器人技术生成阵列并同时执行多个杂交反应。基因芯片已广泛应用于物种诊断、功能基因检测和基因表达。诊断基因芯片是最早的高通量识别方法,能够在物种水平上针对特定的预先选定的病毒、细菌和真菌病原体和害虫分类群进行鉴定。第一个用于选定的植物病原真菌和卵菌的诊断基因芯片仅包括>10种,并能定量低丰度存在的病原体。Mendes等人(2011)通过结合经典拮抗试验和由>106探针组成的高密度基因芯片,鉴定出根际微生物分类群和对真菌病原体索兰根octonia solani具有拮抗作用的特定基因。基于系统芯片的分析显示,致病的阿拉伯树突状卵菌(Oomycete Hyaloperonospora arabidopsidis)对植物的感染促进了根际微生物的生长,从而触发了植物的系统抗性,并减少了植物后代的损伤。
虽然早期基因芯片使用PCR扩增模板,但灵敏度的微调能够从基因组DNA中检测分类群。基因芯片还可以检测单核苷酸多态性(SNPs),这使得植物病原体的基因分型和入侵性菌株的检测成为可能。可重复使用的基因芯片运行费用较低, 并能迅速提供高度敏感的结果, 从复杂样本中监测特定致病类群和致病性相关基因的存在和丰富程度,从而满足监控需要, 他们的主要缺点是无法检测出特定目标环境和各种非最佳紧度探针中的大部分物种和其功能。因此,基因芯片已经逐渐被高通量测序技术所取代。
2.3 高通量测序技术应用于物种鉴定
2.3 | HTS methods for identification of species
高通量测序代表了几种正在发展的新兴技术,它们在记录核苷酸的变异方式上有根本的不同。此外,这些方法在通量、读取长度、准确度和技术偏差方面存在显著差异。在上市的头5年,HTS平台通常在通量、数据质量和分析成本降低方面发展迅速,但此后不久,技术限制就成为最主要的限定因素。HTS方法几乎每年都会更新,但其中只有一小部分得到了公众的关注和认可,大约一半能够上市。表2概述了使用各种不同的HTS平台开展的研究。
第一个商业上可用的HTS方法,即454焦磷酸测序(罗氏诊断公司,瑞士巴塞尔),是在2000年代早期开发的。454技术比Sanger测序便宜100倍(10 -2欧元/读长),其化学分析过程被迅速优化,并提供每条50到700 - 1000个碱基,总共120万序列的测序通量。454技术被微生物生态学家迅速采用,他们对原核生物的超高多样性研究有了突破性的发现。有趣的是,这些发现的许多多样性最终被证明是来源于人为产物(analytical artefacts),这表明有必要对样品制备和分析步骤进行严密的质量控制和优化。排除稀有样品中的人为影响仍然是所有HTS技术中最大的问题。
在这些开创性的针对原核生物的研究之后不久,454焦测序被用于鉴定真核生物,并根据室内环境、动物样本、土壤和树叶的分类学信息,将潜在的致病类群从其他类群中分离出来。在实施数年后,454方法被用于从哺乳动物和六足动物的食物中鉴定植物等大型生物,还包括动物, 寄生线虫和其他害虫。
Illumina (www.illumina.com)和Ion Torrent (www.iontorre nt.com)技术在2010年代早期取代了454,因为这些新技术具有更大的测序通量和更低的成本。然而,Ion Torrent持续受到短序列长度(高达450 bp)和波动的序列质量的困扰,这限制了它在土壤和植物样本分析中的应用。 与454平台相比,Illumina技术提供了高达3000倍的测序通量,数倍的精度和相对较低的成本(10-5-10-4欧元/读长)和最高550 bp(2×300对‐端选项)的序列长度的可能性。与其他平台相比,Illumina测序的主要不足之处在于嵌合序列的产生和长时间的人为插入或删除的存在。目前,考虑到序列质量和文库制备和测序的分析成本,Illumina测序是短DNA/RNA条形码研究和宏基因组学的最佳选择。尽管自2015年以来的技术进步微不足道,但到本世纪末,它无疑仍将是使用最广泛的HTS方法。Illumina测序的超高通量允许在足够测序深度的单次运行中分析> 1000个样品。Illumina测序显示,豆科作物轮作极大地增加了土壤中的病原体负荷,并产生了若干年的遗留效应。Cline等(2017)研究表明,草原土壤病原菌相对丰度随着植物生物量的增加而增加。
2015年,华大基因发布了(paired‐end ultra‐HTS platform) (www.seq500.com/en/)。迄今为止,它已被用于人类病原体的宏基因组检测。 因为它的读数比较短, 长度(2×150对/端或400个碱基的单端长度),目前是基于扩增子的生物体检测和识别的次优选择。
作为一项重大的技术进步,跨越更长的DNA片段可以使用太平洋生物科学
(PacBio, www.pacificbiosciences.com)和牛津纳米孔Nanopore
(www.nanoporetech.com)技术,它们分别于2011年和2015年上市。然而,这两个平台都有非常高的初始错误率(每个基础错误率为10%-15%),在过去几年中仅略有改善。在PacBio平台上,循环的DNA分子被多次测序,将错误率降低到最低(0.1%)在9到11倍的一致性
。 给定平均原始读取长度为30 kb, PacBio允许最多
5 kb DNA片段的测序,质量令人满意。对单个分子的长片段进行测序在DNA条形码中变得很有吸引力。例如,Hebert等人(2017)报道了同时对约10,000个节肢动物标本的DNA条形码进行测序。在一项开创性的研究中,PacBio被成功地应用于鉴别咖啡锈病的潜在真菌寄生虫,Hemileia vastatrix。一般而言,长片段通过对不保守区域更强的分类分辨率和基于相对保守区域的系统发育分析大大提高了识别能力。 然而,与Illumina测序相比,PacBio的文库制备和测序步骤都相对昂贵(约300欧元/文库,10-2欧元/序列)。因此,在资金充足的情况下,PacBio是>550 bp宏分子标记测序的较优选择方案。
尽管双链(1D2 流动细胞)和分子内一致性测序(INC‐Seq)技术已开发完成,但Nanopore在生态学和病理学上的应用一直受到序列质量低的困扰。最近的发展将错误率降低到2%-5%,但是仍然不足以区分亲缘关系非常近的物种。生态学中的第一次应用案例还强调需要更长的标签,并避免使用聚类方法。因此,在基因组测序中, Nanopore MinION平台主要作为一种廉价的选择,用于填补序列之间的缺口、解决长重复片段和作为支架(scaffolds),或用于进行全基因组重测序。例如Rhodes等人(2018)对一种机会性人类病原体, Candida auris的多个菌株进行测序, 并认为这是一种印度起源的致病菌株(虽然还有待证实)。Nanopore一致性序列已被用于同时从多个节肢动物标本中获取长DNA条形码。 然而,使用Min ION设备对真菌和卵菌进行常规的基于宏条型码的识别存在着以下问题:有意义的序列比例非常低、标签混淆事件极其常见,以及样本之间高度不均一的测序深度等问题。 尽管有很高的错误率,Nanopore技术成本低,分析时间仅为1-2天,在疾病诊断领域具有广阔的前景。独特的直接RNA测序方法(没有cDNA逆转录步骤)特别有趣,但它需要对环境样本和分析偏差进行测试。
2.4 宏基因组与宏转录组
2.4 | Metagenomics and metatranscriptomics
PacBio和Oxford Nanopore有时被称为第三代测序技术,因为其序列读长较长,而且有可能不需要PCR步骤。此外,Sanger方法(Green Tringe et al., 2005)和multiple HTS方法(Frey et al., 2014)分别用于从DNA和RNA(通过逆转录)分子中生成无扩增鸟枪法宏基因组和宏转录组序列数据集,以解决动物病原体的分类多样性。除了病毒(见下文),鸟枪法宏基因组学和宏转录组学专门针对植物病原体的研究很少,尽管除了自由存活的微生物及其功能外,有几个项目无意中涵盖了病原体。更具体地说,是Doonan、Denman、McDonald和Golyshin
(2017)对结节茄软腐斑的宏基因组进行测序,鉴定多种微生物的相对丰度及其潜在功能。宏转录组方法显示,致病的octonia solani根定植改变了根细菌群落,并诱导了相关应激基因的表达。 一项基于Nanopore的宏基因组研究显示,入侵性褐家鼠在新西兰主要以植物和昆虫为食。 病毒没有通用引物,因此宏基因组学和宏转录组学是检测以前未被识别病毒的唯一合适方法。
由于引物选择的不同而产生的各种具有偏差的PCR扩增过程,免PCR技术为生物,特别是病毒和细菌的分子鉴定提供了广阔的前景。尽管产生了大量的序列数据,分类鉴定真核生物研究中鸟枪法宏基因组或宏转录组是一个低效的方法,因为只有一小部分序列可能来自相关的标记基因。此外,宏基因组和宏转录组分析还存在一些技术问题。由于生物体在AT:CG比值上存在较大差异,因此在序列分析中可能不太接受具有极端比值的基因组片段,这取决于分析平台。宏基因组片段覆盖了标记基因在其他基因组区域中的随机延伸,因此不可能在自然群落中处理物种级别的分类学丰富度。基于标记基因的参考数据库,如UNITE 和SILVA,它们包含相对丰富的短rRNA基因标记数据,但是很少有分类单元有完整长度的参考序列进行比较。基因组参考数据库的不足,可能导致门级水平识别错误。尽管如此,宏基因组学和宏转录组学为同时识别分类群和描述它们的潜在功能提供了一种选择。
线粒体宏基因组学由于线粒体的丰度高、进化快、无PCR偏倚,已成为土壤节肢动物生物多样性研究的高质量替代方法。Gomez‐Rodriguez、Timmermans、Crampton‐Platt和Vogler(2017)发现,与COI宏条形码技术相比,线粒体宏基因组学对分类单元发现更为敏感,它允许处理种内变异和构建支持率更高的系统发育关系。他们的研究结果表明,与表土相比,底层土壤甲虫的物种丰富度和系统发育多样性较高。原则上,线粒体宏基因组学可以应用于任何拥有这类细胞器的真核生物。其主要缺点包括生物体及其不同生命阶段的线粒体数量差异较大,以及在参考数据库中的代表性较差(动物除外)。
3 高通量技术应用于个体鉴定
3 | HTS METHODS FOR IDENTIFICATION OF INDIVIDUALS
当针对快速进化的位点、部分基因组或全基因组中的SNPs时,HTS方法可用于区分个体。 这些比较基因组学方法揭示了拮抗剂的起源、迁移途径、物种形成、宿主转移、协同进化、杂交和水平基因转移,并能够检测毒性基因型及其潜在的遗传机制。
群体基因组学方法也可以通过多倍体化和杂交来检测基因组从其他物种的渗入和新物种的进化,这是新型致病真菌和卵菌产生的常见机制。 普通的宏条形码技术可能无法区分来自父本类群最近的杂交和亲本分类群,因为杂交通常携带核标记基因(单倍体),或线粒体标记基因(几乎所有真核生物),它们来自双亲之一,或来自双亲不同的核等位基因(包括二倍体和多倍体生物)。
早期的HTS群体基因组学方法主要研究SNP在某些可变区域的分布。例如,使用靶向454焦磷酸测序检测人类病原体结核分枝杆菌对药物潜在耐药性的突变。随后的人群水平研究的目标是整个基因组多个基因组片段限制位点的侧翼,这被称为限制位点相关
DNA测序(RAD-seq)。该方法显示了作物病原菌禾本科镰刀菌的高遗传变化率和重组,表明这些特征有助于快速适应抗病品种和生物杀灭剂。RAD‐seq还揭示了几种共生真菌不规则根瘤菌的共存群体,其中大多数分布在全球。
随着HTS成本的急剧下降,可以很容易地从纯培养物、宿主组织和土壤环境中确定致病生物体的部分和全部基因组。全基因组测序(WGS)的主要优势是产生了几个数量级的更多关于多态性位点的信息,更好地理解它们在外显子和内含子中的连接和发生。低覆盖基因组和生物体的标记基因和功能基因可以从向微量DNA进行测定,如基于从旧植物标本和根中提取的DNA浓度。因此,从同一物种的多个分离株中测序基因组已成为微生物学和最近在植物病理学的主要方法之一。由于基因组的大小和遗传物质组织成多个染色体,组装真核生物的基因组比原核生物更困难,需要更高的费用。Duan等人(2009)使用鸟枪宏基因组方法对一种来源于亚洲白念珠菌的木虱载体 的未培养的植物致病菌的基因组进行测序。在群体基因组学研究中,Cooke等人(2012)发现了疫霉菌中一个高毒基因型群的近期进化及其克服宿主抗性的遗传机制。在全球范围内,胎生疟原虫的WGS表现出明显的种群分化和地方性。WGS分析使用牛津Nanopore正在成为一种常规,用于“实时”鉴定致病复合物中的菌株和隐存物种,这已在大肠杆菌(Escherichia coli)和埃博拉病毒中得到证实。
优化的样品制备和HTS使单细胞WGS成为可能。麦克莱恩等(2013)对一种机会性人类病原体的基因组进行测序。对于细菌和古生菌来说,由于基因组规模较小,单细胞测序可能表现良好,但在真核生物中问题较多。单细胞WGS显示,人类神经元的部分基因组拷贝数不同,一些细胞在特定区域有多个突变。使用基于微流控的单细胞制备和测序技术,Gawad、Koh和Quake(2014)测定了来自白血病细胞的SNP,揭示了癌症发展的机制。Nair等人(2014)展示了单细胞测序在小基因组原生生物中的潜力,展示了疟原虫对人类患者感染的复杂性,以及该方法在区分高毒性菌株方面的效率。单细胞WGS还未被应用于植物病原体,但它在理解无法培养的单细胞病原体的功能方面具有最大的前景,如早期分化的真菌谱系和肺泡的成员(Ahrendt等,2018)。
4 以活的生物为目标
4 | TARGETING ACTIVE ORGANISMS
DNA分子通常寿命较长,在死后数周至数月仍可检测到,这取决于片段长度和栖息地性质。短的(小于200 bp) DNA片段可能在缺氧沉积物和永久冻土中保存数千年(Allentoft et al., 2012)。相反地,rRNA(包括ITS区域)和功能基因mRNA提供了在几小时到几天的时间框架内研究转录基因的可能,有效地排除了休眠的生物体(卵、孢子、菌核、囊肿等)。然而,RNA的采样需要额外的小心和快速的预处理,例如在液氮中冷冻或储存在RNA保存缓冲液中,这是昂贵的,有时是低效的。然而,在RNA病毒的病理研究中,靶向RNA是不可避免的。
作为RNA的替代品,微生物生态学家使用了13C 和核苷酸类似物3‐溴‐脱氧尿苷与底物结合并与代谢这些富集底物的生物体DNA同化。13C‐稳定同位素探测(SIP)在自然条件下难以进行,因为多个碳源稀释了同位素信号。 这与真核病原体的生长和积累特别相关。
13C或核苷酸类似物缓慢进入它们的DNA,并可能使用许多标记的碳进行呼吸。尽管如此,13C与DNA和脂肪酸结合后发现,在次生演替过程中,植物源碳通过土壤食物网流动,病原体与菌根比下降。该方法可用于处理在其他生物体中使用最新光合作用产物的害虫和病原体,或检测潜在的生物控制剂。
5 高通量测序方法的技术偏差
5 | TECHNOLOGICAL BIASES OF HTS METHODS
所有的基于分子的分析方法都存在特定的分析偏差。 标记偏倚可能选择高拷贝数量的生物体。引物偏倚会对引物‐模板不匹配的目标产生歧视,尤其是在引物的3 ‘端,这会使它们的相对放大效率降低1-2个数量级。ITS区域的引物偏差在几种动植物病原真菌类群、线虫和肺泡菌中都很重要。PCR偏倚表现为由于AT:CG比值、DNA二级结构和标记物长度的差异导致的靶物种扩增不均匀。 一些节肢动物和真菌组在rRNA基因或长ITS1或ITS2区域中表现出内含子,这可能导致相应的分类群完全不被代表。 最好的例子是灰分枯死病病原体Framenoscyphus fraxineus,它展示了一个长3 ‘端18S内含子,这使得使用其1f / its前引物无法检测该物种。大多数Oomycota具有较长的ITS2区,在针对所有真核生物的研究中可能会被忽略。在克隆和分子标识符标记连接过程中也可能会出现特异性偏差。在此过程中,可能会偏重或忽略具有高度倾斜于:CG比值和特定末端核苷酸的变异。在同一测序平台上,不同的模型和化学分析方法之间也可能存在偏差。
标签混淆和嵌合分子形成是HTS中常见的技术产物。嵌合分子通常在PCR过程中形成,当扩增不完全时(由于低加工性、较短的扩增时间或核苷酸耗尽),这些短片段在随后的周期中启动为模板。随着PCR周期的增加和群落的复杂性的增加,嵌合分子在两个密切相关的生物体之间的形成更为普遍。从本质上讲,标签混淆(tag‐switch)人工产物也是嵌合分子,在PCR后文库制备步骤中在多组分样品之间形成。
6 基于高通量测序技术的实用性建议
6 | PRACTICAL RECOMMENDATIONS FOR HTS‐BASED RESEARCH
6.1 实验设计
6.1 | Design of HTS studies
研究设计取决于研究的目标。除了基因组、转录组测序,或验证新方法之外,单纯的描述性研究很难发表,且重复性较低。检验生态假说需要一个适当的、重复良好的取样设计。许多研究人员似乎忘记了技术重复、多个空间自相关的子样本和数千个发现的OTU并不能作为独立的生物重复。这在具有固有层次设计和多级空间自相关的地理结构抽样中尤为重要。
病理学和微生物学研究的主要问题之一是是否要混合子样本。混合池(Pooling)可能强烈降低分析成本,但也降低了小规模的分辨率。是否选择混池测序方案取决于研究目标、时空尺度和特定研究样本的性质。如果个体样本(例如,树叶,土样/土芯)都很小,并且很可能不能很好地代表群落,收集多个样本是一个可行的选择。在分层设计的情况下(即,以块、地或地点为结构),在不关心内部变化的情况下,汇集多个子样本是很有用的。然而,在大多数其他情况下,由于能够估计抽样误差和处理时空变化的重要性,对多个独立样本进行分析是可取的。HTS可以很容易地恢复分类学和基因组成的微小变化; 因此,多变量统计分析技术只需要3-4次重复就可以检测到群落组成的重要生物学变化。应该考虑额外的重复,因为从一些样本(通常为1%-10%)中获得低质量的DNA或有限数量的序列是很常见的。由于单变量试验具有较低的统计能力,因此对丰富性、多样性和病原菌载量的分析需要更多的样本。
6.2 样品准备
6.2 | Sample preparation for HTS analysis
HTS对腐败、外部污染和交叉污染非常敏感,因此需要仔细收集、处理和预处理,以防止快速生长的模板或DNA/RNA降解造成污染和过度生长。 对于预处理和储存,快速空气干燥、冷冻干燥、深度冷冻和固定在保存缓冲液中同样适用于DNA。在-80°C深度冷冻和储存对于进一步分析RNA、蛋白质和脂肪酸非常有效。干燥和冷冻的样品和冷冻的DNA溶液通常可以保存几十年,质量损失较小。
为了避免在分析的各个步骤中受到污染,建议将实验室空间划分为湿实验室、DNA提取与PCR实验室、PCR后实验室和HTS制备实验室。以往项目的PCR产物可能是HTS研究的主要污染源,因为单个漂浮的DNA分子可能被逐步扩增和测序。这可以通过使用紫外线和含DNase的溶液对实验室进行有效的清洗来控制。所有分析阶段的阴性对照能够检测和跟踪污染物。
DNA或RNA提取可以使用自20世纪90年代以来开发的商业试剂盒或经典方案进行。由于提取过程可能会影响后续分析中发现的群落组成,因此应考虑质量、底物和目标生物选择最佳方案。 样品可以在较低的总质量(0.25 g)下提取,这能与高质量提取发现几乎相当的分类学丰富度(10 g)。对于萃取量较小的样品,更重要的是样品的完全均匀化。对于超长扩增子和基因组分析,纯化过程中与磁珠的接触应保持在最小持续时间。基于宏基因组或RNA的分析,尤其重要的是要尽量最小化萃取到的腐殖酸和糖类的浓度,因为这可能干扰下游过程。从土壤和其他富含有机基质中提取的DNA可能需要额外的净化步骤,比如使用过滤器柱或磁珠,以便在宏基因组学分析中获得最佳性能。
6.3 分子标记与引物选择
6.3 | Marker and primer selection
HTS的多样性分析中,基于所需的分类分辨率全面考虑DNA/RNA标记的选择是非常重要的。在常规的群落水平分析中,物种水平的解析应始终以避免病原微生物与密切相关的内生菌和腐生菌聚集在一起为目标。尽管如此,同种拮抗剂的菌株在致病性上可能存在很大差异,这使得基于条形码标记物的病原体检测结果有些不明确。细菌的种类通常是根据16S rRNA基因进行鉴定,因为这个标记能有效地完成亚属和属水平上的鉴别。 ITS区域包括16S - 23S rRNA基因之间的tRNA基因,其分类学分辨率高得多,但其长度变化较大。在原核生物的HTS研究中,AT:GC比列可能会产生偏差。真核生物中,真菌和卵菌都是基于ITS区域进行常规研究的,ITS区域已被建立为后者的正式条形码。原生动物门和后生动物门中,18S和28S rRNA基因及其与线粒体细胞色素C氧化酶I亚基(COI)得以使用,需要综合考虑分类分辨率和可用参考数据。 官方动物条码COI在HTS分析中表现不佳,原因是缺乏包含引物的保守区域,且随着多次退火,引物‐模板特异性丧失。 ITS区域和28S提供了类似于物种水平的分辨率,并允许在大多数动物群体中使用强引物(strong primers),如线虫、Acari、Collembola和六足动物。然而,特别是在ITS区,数据库中很少有来自大多数节肢动物、线虫和原生生物群体的物种和属级参考序列,因为这些被排除在经典条形码之外,部分原因是杂合等位基因Sanger测序困难,以及系统发育推断能力较差。
在标记基因选择的指导下,引物的选择非常重要。考虑到高测序深度,明智的做法是从广泛的角度关注生物体,以确保包含几乎所有预期的目标分类群。由于分类的特异性引物很少具有高覆盖度,因此建议在处理真菌时关注所有真核生物,或在针对卵菌纲时关注Stramenopila。
用于靶向组织中的病原体植物或任何其他特定生物体,研究人员可能会考虑排除宿主DNA的引物,或包括额外的阻断引物(blocking primers),特别是如果预期目标病原体的相对丰度非常低。阻断引物具有3 ‘端核苷酸修饰,且浓度较高,通过与常规引物下游的宿主DNA特异性结合,阻止宿主DNA标记的退火和伸长。这一程序经常用于线粒体DNA分析,但不可能有效地设计核RNA基因。另一方面,协同靶向宿主DNA可以确定病原体DNA标记相对于宿主DNA标记的相对丰度,且具有可比性(即,无系统偏差)。
回顾早期广泛应用的引物设计, 1990年代显示了目标群体中多个分类单元的次优性能和多重意外不匹配。 虽然1-2个非末端错配在Sanger测序样品制备中并不重要,但单个错配可能导致分类单元被低估1-2个数量级。积累的序列数据使分子生态学家能够为原核生物、真菌、卵菌和动物构建更有效的简并引物或引物混合物。 表3提供了高亲和力引物的选择,推荐用于HTS研究。
在分析杂食动物复杂的病理系统、食物网和饮食结构时,可以同时针对多个门的生物体。这方面的策略包括一个通用标记,例如所有真核生物的18S rRNA基因或ITS,或每个门的不同标记。rRNA标记物存在通用引物和引物混合物(表3)。 相同长度的不同标记物,经分离放大后,可进行多重或混合分析,形成一个公共文库。然而,标记物在平均测序深度上可能产生>2个数量级的差异,表明需要事先考虑相对判别因素。宏基因组方法已被证明是相对定量土壤中目标生物DNA和肠道内容物的可行方法。
用于HTS的一个或两个引物应该用分子标签标记,以支持多样品混合测序。这些典型的6-12个碱基的标签之间应该至少有4个碱基/indel不同(例如,“error‐correct” Golay标识符)来防止标签中的随机突变或不纯合成错误地在样本之间切换序列。标记引物可能还包括平台特异性测序引物,但如此长的引物可能表现不佳。 Illumina公司测序, 96种Nextera序列标签的组合可以通过PCR进行接接。仅用标识符标记的引物更便宜,可以用于使用任何测序平台的分析,使这些引物可用多年。为了减少连接步骤中标记扩增子之间的竞争,建议选择所有标识符以相同的核苷酸开始,并使用2‐碱基连接子序列,而不与任何模板匹配。具有AT:CG比值小于或大于0.25-4的标识符标记的性能往往较差。 强烈建议在反向引物和正向引物中添加标识符标签,以最小化标签‐切换。
6.4 扩增
6.4 | PCR
在PCR之前,建议对DNA进行量化,并对每个样本使用等量的模板,以便能够在整个研究中使用相同数量的PCR循环数。聚合酶链反应混合物应包括高亲和力和高保真性聚合酶(如Pfu, Phusion, Q5)尽量减少错误核苷酸的掺入,并在随后的扩增周期中生成可转换为嵌合序列的部分序列。这些更昂贵的聚合酶大大减少了嵌合序列的数量,以及由于错配产生的人工分类群。对于HTS分析,与常规PCR相比,引物退火温度可降低约5℃,以促进与引物有1 ~ 2个不匹配模板的扩增。
PCR周期的数量应保持在最低水平,以便在琼脂糖凝胶上看到一个相对较弱的、片段长短合适的条带。延长扩增时间也可能减少不完全扩增,从而减少嵌合体的形成。低输入DNA含量导致较少的抑制剂和较少的嵌合序列。由于随机变异,建议使用至少两个PCR重复,可以扩增后合并。
扩增子纯化依赖于下一步的分析和测序平台的选择。建议将样品间扩增子浓度标准化,以减少样品间测序深度的倍数变化。 在库准备步骤中,等量混合扩增子,并与适用于特定测序平台的接头进行连接(adaptor ligation)。建议从测序服务提供者处订购接头序列,以确保它们的质量标准,并将失败的风险留给服务提供者。
研究人员应该提前咨询每个服务提供者所需样品数量和质量的要求,因为不同的测序公司可能有很大的不同。数量似乎是可以协商的,因为服务提供商通常要求5-10倍于他们测序实际需要的材料。由于需求量大,一般需要1-2个月才能收到序列。订购生物信息学分析服务并不划算,因为公司提供标准质量的fastq格式化文件。这些可以在任何生物信息学平台中使用自定义选项来处理,而服务提供者的分析流程可能不是最优配置(即针对细菌16S rRNA基因、小鼠或人类样本进行优化)或他们的分析方法并不透明。
6.5 对照与重复
6.5 | Controls and technical replication
为了量化污染和人为影响,如测序错误、嵌合体形成和标签‐切换(tag‐switching),建议同时运行三种类型的对照样品。在样品准备的各个步骤中,均应纳入阴性对照样品(DNA / RNA提取,PCR)。一个或多个阳性对照。
已知成分的模拟群落可以提供关于嵌合体形成速率和成分分类单元发现效率的信息。利用人工DNA分子进行阳性对照和模拟群落样品是可行的,因为它们的长度、在AT:CG比和均聚物含量可以被控制,而且它们的浓度可以被精确地测定。
在大多数情况下,技术重复是不必要的,因为这些观察结果不能用作分析中的独立数据点。然而,少数样本有限的技术复制可能是可行的,以估计该方法的性能和重现性。
6.6 数据质控
6.6 | Quality filtering of HTS data
由于HTS数据量大,没有清晰可读的结果,因此对HTS数据的分析和质量筛选远比查看和编辑Sanger测序读取更具挑战性。有无数可用的生物信息学数据分析软件,其中大多数,如mothur (www.mothur.org)和QIIME (www.qiime.org)都是在命令行上运行的。这两种流行的生物信息学平台都针对基于比对的细菌16S rRNA基因分析进行了优化。QIITA (https://qiita.microbio.me)是最近开发的一个基于web的生物信息学分析平台和分析工作流程。PipeCraft是一款用户友好型软件,具有图形界面,多种选项与其他程序相结合,能够分析来自所有测序平台的宏组学数据,兼容Linux、Mac和Windows,这些都吸引了非生物信息学研究背景的用户。在分析非校准标记物(如ITS)时,PipeCraft在输入数据格式、可用分析选项和输出质量方面优于bioinformat的其它方法。关于扩增子和宏组编码数据的可用分析平台的全面概述见Oulas等(2015)。Nilsson等人(2018)综述了真菌数据的生物信息学分析。
虽然HTS平台的输出被转换为相同的格式,但是这些数据的错误分布不同,需要不同的分析选项。 质量控制是生物信息学分析的第一步,通常是在序列(或整个序列)低于特定的质量阈值时,去除3 ‘端序列(或整个序列),其最优值因测序平台的不同而不同。在同时进行的样品多组拆分过程中(demultiplexing),序列根据分子标识符被重新分配给特定的生物样品。对于使用Golay条形码分组的序列数据,我们建议允许1-2个标签不匹配和1-2个引物不匹配来解释随机错误和自然产生的引物‐模板不匹配。从单端进行分组通常能够发现40%-70%的序列,但是当含有其他标记引物时,大约有四分之一的序列会丢失。然而,双‐标签多样本混合测序能够减少标签切换和人为产物,以及不完整的序列。
为了减少计算时间,经过质量筛选的序列通常使用100%或99%的标识进行预聚类,并移除嵌合体。嵌合体检测是在结合de novo和UCHIME中实现的,可以在所有生物信息学平台上运行。
可变区域的提取可以在嵌合体检查之前或之后进行。rRNA基因、ITS和其他可变区域的提取能够同时去除非目标生物,并专注于更短但更可变的条形码,从而提高分类分辨率。
6.7 序列聚类与可操作分类单元(OTU)
6.7 | Sequence clustering and Operational Taxonomic Units
经过过滤和修剪的序列被聚类成OTUs,其中存在多种算法。在使用带有单‐连接算法的开源从头/无参(de novo)聚类时,获得了最佳结果。 除了Illumina的数据,建议将均聚物(homopolymers)折叠成三聚体进行聚类,或降低空缺扩展罚分(gap extension penalty),因为其他平台对均聚物中的插入和缺失很敏感。尽管许多分析流程建议删除含有>8或>10 均聚物碱基的序列(低复杂度序列,如PolyA),我们不鼓励对非编码区采用这种做法,因为许多生物体的这些分子标记本身就含有均聚物。
虽然分类学分辨率不同,但细菌16S和真核生物18S、28S、ITS和COI序列的聚类相似阈值一般为97%,这被认为是自然种内和种间序列变异与随机测序误差的折衷。所有这些标记基因97%的序列相似性阈值(某些群体中COI除外)对于大多数分类单元的物种水平识别来说过于保守。例如,一些种类的镰刀菌在其相对不保守的区域根本没有表现出任何变异。 因此, HTS分析其 28 s rRNA基因和转录延长因子时使用低分辨率标记物,可以使用100%相似阈值进行分析。
随着测序深度的增加,所有聚类方法在任何条形码阈值下产生的OTU都比预期的要多,这表明PCR的积累和测序错误引入了罕见的“卫星”分类群(“satellite” taxa)。在随机误差被消除的前提下,可以通过执行两个或多个连续的聚类步骤来改善分析结果,即基于共发生或系统发生算法的聚类后分类移除或者关注更长的DNA片段。 进一步建议是删除单体(Singlton),或者删除序列<5或<10的OTUs(视测序深度而定),因为这可能是人为的。
6.8 基于序列的分类鉴定
6.8 | Sequence‐based taxonomic identification and taxon communication
选择一个或多个参考数据库对基于序列的识别至关重要。 由于INSDc中多达20%的数据质量低劣或鉴定错误,诸如UNITE (https://unite.ut.ee/)、SILVA (www.arb-silva.de)和UniEuk (https://unieuk.org/)生成了数据库和引用数据集,其中补充了经过过滤和第三方注释的序列。**SILVA主要研究原核生物和真核生物的核SSU和LSU序列,但卵菌和真菌的代表性都很差,在分类学上也存在一些问题**。 UniEuk项目的重点是分类和管理真核生物的高质量18S rDNA序列。目前版本的UNITE包括所有真核生物的SSU、ITS和LSU序列数据,尽管只有真菌和卵菌对其序列进行了详细的分类、序列质量和生态元数据注释。特别是一些致病真菌群,在UNITE中已有较好的研究基础,并标注和分配了类型状态。在BOLD数据库中,保存了动物、卵菌纲和其他特定原生生物群体的COI序列数据。因此,这些数据库为病原生物的一般分子鉴定提供了最适合的物种级参考数据。然而,研究人员侧重于更窄的群体,如镰刀菌或疫霉菌可以使用镰刀菌Fusarium‐ID
和Phytophthora数据库。
动物和人类病原体已经在ISHAM‐ITS数据库中标注了序列数据。宏基因组和宏转录组分析需要包含功能基因和基因组数据库,用于实现分类和功能分析的结合。检测基因组、宏基因组和宏转录组序列之间的病毒需要一些特定的数据挖掘工作,比如VirusFinder等流程。
VirFind 能够从自定义序列中搜索病毒基序。病毒学家主要使用INSDc的RefSeq数据库和综合植物病原学基因组资源数据库(Comprehensive Phytopathogen Genomics Resource, CPGR)作为鉴定的参考。
对于分类分配,最常用的方法是使用基于BLAST的相似度搜索方法来搜索每一个具有代表性的序列OTU。 朴素贝叶斯分类器(Naive Bayesian Classifier)被广泛用于原核生物的保守鉴定,但由于该方法在种或属水平的分类单元中所占比例较低,因此在真菌学家中不太受欢迎。ProTax在真菌中得到了改进,它提供了从物种到门级别的不同分类单元分配精度的统计评估。 在真菌和卵菌中,其序列的种、属、科、目水平与最接近的序列相似性分别可近似为> 97%-99%、>90%、>85%和>80%。在细菌16S(全长)中,这些数字为> 98%-99%,>94.5%,>86.5%, >82%。由于不同的rRNA基因的进化率不同(有多个较为意外的案例),sordariomycete(子囊菌科)和Oomycete(卵菌)表现出较近的相似性,而早期分化的真菌类群相似性较低。这些差异在动物和原生动物之间甚至更大,但对大多数类群仍知之甚少。
一个可选的步骤是将功能特征(如致病性信息)分配给OTUs,有相关的数据库提供了这样的工具。比如对细菌而言,一个自动化的SINAPS分析流程 能够使用已有的性状参考数据库搜索和预测自定义性状
。真菌的基本特征可以使用FunGuild数据库中的工具分配到分类配置文件。它的主要限制是属水平的操作(尽管它警告说许多属同时含有病原体和腐殖质或内生植物)。如上所述,检测到的“病原性”OTU在非宿主上可能是非病原性的,这使得赋值非常依赖已有的研究。因此,迫切需要更精确的宏数据,包括与寄主和特定组织相关的物种特征、物种假设(见下一段)或个体和序列。
HTS研究能够发现数以万计的OTU,其中大多数通常不能被分配到所描述的物种,这使得这些OTU很难在不同的研究者之间进行交流。UNITE和BOLD数据库使用与数字对象标识符(DOIs)相关联的分类单元代码(分别是物种假设和箱子BIN)。这些机器可读的DOIs应承担使命名和未命名的分类单元能够跨研究和时间实现不同课题的交流。
6.9 后生信数据质控
6.9 | Post‐bioinformatics data quality control
对于具有非最佳贮藏条件的土壤和原始组织样品,由于霉菌可能过度生长,估计样品质量可能很重要。这可以通过测量提取的DNA/RNA分子在凝胶上的平均大小或计算霉菌的相对丰度来实现。单个霉菌的(single mould)OTU极丰富通常导致分类学上的丰富性降低有关,可以认为是样品变质的标志。
同样,排除序列小于中位数5‐10倍的样本也是可行的。这种较差的回收率可能归因于未能对样本进行标准化、特定标识符标签的性能较差和/或样本中特定生物体的优势,这些在库准备、测序或质量筛选步骤中是不受欢迎的。尽管试图标准化扩增子的起始测序上样量,但收到的序列数量通常大于标准化的3倍。将所有样本稀释到相同的最小测序深度是很常见的策略,但这会丢失绝大多数分类信息。因此,建议计算相对于平方根或排序深度的对数函数(哪个更合适?要根据实际情况考虑)的丰富度残差,或者在单变量和多变量统计中使用这些函数作为协变量。
由于高灵敏度,HTS通常遭受环境污染或标签‐切换的影响(见上文)。关于质控OTU和实验样本的信息可以评估这些技术偏差,并需要额外的质量过滤。在广泛的标签‐切换的情况下,可以根据统计公式删除序列。 尽管标签‐切换的人工制品通常占所有序列的0.1%-3% ,这些可能模糊定性多样性分析,特别是对添加低‐丰度敏感的网络分析。更重要的是,标签切换可能会产生假阳性的影响,低水平的病原体或生物控制有机体的存在,特别是当这些占主导地位的一些样本在测序文库中。
6.10 数据分析
6.10 | HTS data analysis
HTS为每个样本生成大量OTU数据矩阵,这些矩阵有时不能完全加载到常见的电子表格程序中。因此,专家使用python或perl脚本解析和转换其文本格式(责编注:目前分析更多使用R语言的矩阵或数据框结构)。这些大型矩阵也测试了统计软件和处理器的极限。许多常用的系统发育、自举检验和网络分析方法在计算上变得非常困难。因此,使用高效的计算算法是必要的。为了减少对计算资源的需求,可以通过删除稀有物种来压缩数据,这通常会减少无法解释的方差,提高统计能力,但在多元或网络分析中,它对潜在的I型和II型误差的影响是未知的。
对于多变量分析,我们建议降低OTUs丰度Hellinger(平方根)变换来解释HTS的半定量性质。不推荐使用定性的二进制数据(存在/不存在),因为由于丢失了(半)定量信息和人工均衡潜在的人工低丰度(包括人造的标签互换)和真实高丰度OTUs,拟合度较低。我们建议使用PERMANOVA, 因为它允许包括交互作用、随机因素和嵌套设计,所以可以对群落组成的变化进行显式统计检验。ANCOM和随机森林机器学习算法提供了关于群落矩阵中每个OTU性能的统计信息。适用于HTS的多元分析方法的一般信息见Buttigieg和Ramette(2014)。值得注意的是,同样的多变量技术通常用于分析标准化基因芯片和宏基因组和宏转录组数据。
在单变量分析中,可将OTU的丰富度、多样性、定植性、损伤和某些分类或功能的相对丰度作为因变量。除了考虑测序深度和罕见OTUs的发现外,分析还应遵循最佳统计实践,包括适当的转换、测试假设等。Balint等人(2016)概述了高通量数据统计分析的一般建议、高效计算程序和潜在缺陷。
6.11 数据储存与报告
6.11 | HTS data storage and reporting
HTS数据集既作为原始数据文件存储,也作为详细的数据集存储。原始的fastq文件、元数据文件以及带有标识符标记和基本信息的文件保存在SRA中。这些文件使用户能够执行生物信息学分析的所有步骤,包括生成OTU表和鉴定。这在几个方面都很重要,比如用更新的过滤程序确认早期发现,解决其他问题,使用标准化的数据过滤程序进行再分析。但是,不鼓励只提交代表性序列到公共数据库,因为OTU序列短、具有潜在的人为影响和不可靠的分类注释。这些环境序列将增加不良注释和错误数据的比例,并在后续研究中使对它们的识别更加复杂化。
包括技术和环境元数据、代表性序列以及分类和功能注释在内的按样本排列的OTU‐矩阵应存储通用的数据格式,并保存于特定的数据库,如Repository (www.datadryad.org)和DataOne (www.dataone.org)。
Darwin Core (https://rs.tdwg.org/dwc/)是生物多样性数据的主要标准,它与MIxS (https://gensc.org/mixs/)和
mimark (https://wiki.gensc.org/index.php?title= mimark)标准,由基因组标准协会制定,与所有测序数据相关。
宏基因组数据应该遵循MIxS和MIMS (https://wiki)规则。机器可读的FAIR数据格式允许研究人员理解并快速将数据合并到分析中。这种数字存储库中的标准化数据集支持独立的doi引用。
在出版物中,重要的是在附录文件中提供文中所有数据的存储信息。准确地记录和描述所有分析步骤(包括数据筛选中的特定选项)也很重要,因为这些信息向读者提供了有关数据质量和筛选严格程度的重要细节。尼尔森等人(2011)对分子和生物信息学分析所需的细节提供了详尽的建议。
7 展望
7 | PERSPECTIVES
目前只有一小部分可用的高通量鉴定潜力被用于植物病理学。这与植物病理学家和昆虫学家以实际监测为导向的工作有关,但分子病理学家关注的是人类和动物课题。政府的植物健康监测机构需要遵循经过认证的诊断规程,由于耗时的检测,这些规程发展缓慢。有限的预算也阻碍了政府机构购买高通量分析设备的可能性。考虑到分析成本和时间,专业病理学家肯定会利用qPCR/ddPCR对特定病原体进行实时定量和定制基因芯片同时检测和定量多个选定的病原体。在不久的将来,有可能使用便携式口袋大小的测序仪进行高通量测序来检测包括病原体在内的多种生物,就像使用Nanopore平台来检测病毒一样。对于一个简化的程序,基本上需要一个工作日来收集、分析和解释结果。利用基于抗体受体的光学或电化学检测,可以同时识别多个特定的挥发性分子,从而更快地检测生物体。利用微芯片格式进行信号检测,可大大提高这些生物传感器的通量。
其他高通量识别方法更耗时,但也更灵敏,因此更适合用于研究。宏基因组和宏转录组提供了巨大的潜力,可以同时对目标病毒或原核生物和真核生物病原体和害虫进行研究。此外,线虫、害虫、卵菌和真菌都可以通过使用(针对同一标记物的简并引物混合物)或宏分子标记(针对不同标记物的多重引物)来进行研究。 利用特异性杂交探针和免疫化学方法靶向捕获模板,可使拮抗物的标记基因和致病相关基因得到富集,可进一步使用PCR‐free方法鉴定。
由于具有很高的种内分辨率,高通量指纹和群体基因组学方法为诊断入侵性菌株或病原类型提供了巨大的潜力,并揭示了它们的传播模式和潜在的杂交。在适当的质量过滤下,这些方法足够敏感,能够从数百个样本中同时区分稀有等位基因和SNP 。全基因组测序和转录组分析能够阐明发病机制、促进疫苗和生物杀灭剂的产生以及生物控制剂的选择。
为了正确识别,迫切需要科学领域策划和建立分类注释的参考数据库。这样的数据库目前只维护了少数几个最重要的病原体群,覆盖了主要的条形码标记基因。无论何时这些信息在其中一个数据库中更新, 序列数据库应共享第三方元数据和分类注释信息。
尽管有大量的错误数据,INSDc肯定会继续在连接包含生命各个领域基因的更具体数据库方面发挥核心作用。因此,让我们为INSDc贡献高注释和高质量的序列数据,以造福病理学家的研究者们。这也适用于HTS数据集和数据矩阵,具有很大的实用和科学价值。 这些研究的价值也许几十年后就能识别出来。除了存储序列数据外,重要的是保存组织和土壤样本,这些样本可以在室温下有效地保持干燥。
尽管体积较小,但积累的DNA样本往往会迅速填满整个房间的冰箱,但是冰箱容易出现技术故障和断电。除了基于形态学的重新鉴定和描述新物种的可能性外,植物性和病理性标本馆都为追溯病原体和害虫的进化和传播提供了极好的资源。
总而言之,高通量识别技术为检测和快速识别新的病原体和人类疾病,以及树木和作物种植园,以及哨兵苗圃和植物园等早期预警系统提供了巨大的希望。HTS技术已经在土壤和植物相关微生物群落的研究中证明了它的有用性,可以在新病原体和潜在入侵物种进入新环境并与新宿主接触之前检测它们。我们预测,诸如纳米孔测序、基因芯片和纳米生物传感器等快速监测方法将对疾病早期诊断和生物杀灭剂和生物控制剂等对策的智能应用特别有用。
Reference
Tedersoo, L, Drenkhan, R, Anslan, S, Morales‐Rodriguez, C, Cleary, M. High‐throughput identification and diagnostics of pathogens and pests: Overview and practical recommendations. Mol Ecol Resour. 2019; 19: 47– 76. https://doi.org/10.1111/1755-0998.12959
葛德燕 中科院动物所 副研
主要从事动物系统进化研究,重点探讨不同时期地质气候变化事件对不同生态型动物类群的演化进程与当前生物多样性格局产生的影响,重要功能基因和形态特征的适应性演化。2018年底开始应用宏基因组方法对哺乳动物食性选择和肠道微生物群开展研究。曾参与国家自然科学基金,中国科学院知识创新工程等多个项目的研究工作。目前参与国家自然科学基金,科技部基础专项《藏东南地区生物多样性考察》等项目,参与国家自然科学基金重点项目《中国动物志.鼠亚科》的编撰。曾主持国家自然科学青年基金一项。目前主持英国皇家学会牛顿高级研究者计划基金一项(2016-2020年),国家自然科学基金面上项目一项(2019-2022年)。已发表科研论文近60篇,参与三部专著的编研,一项专利申请。任《Journal of Zoology》副主编,中国核心期刊《兽类学报》编委,IUCN兔形目动物专家组成员,美国哺乳动物学会特别资助会员。在宏基因组公众号发表《PNAS:影响哺乳动物皮肤菌群组成的因素》、《FM:大熊猫的肠道菌群并没特化发酵纤维素的能力》 、《MER:高通量测序应用于病原体和害虫诊断综述》等。
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]() 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读