KOMODO:用16S rDNA序列预测其培养基配方的网站
本文转载自“微生物生态”,己获授权,本平台编辑对内容进行测试和更新。
今天卢瑟菌给大家隆重推出一个超级好的网站,网站名字叫KOMODO(Known Media Database),没错,网站作者就是严(tiao)肃(pi)地把科莫多巨蜥的照片放在了网站主页,见下图1左上角。言归正传,这个网站的神奇之处就在于可以根据细菌或古菌16S rDNA的序列来预测培养该菌的培养基配方!文章发表在2015年10月13号的Nature communication上[1],见下图2。
网址:http://komodo.modelseed.org/
图1. KOMODO网站主页截图
图2. 15年10月发表在NC上的微生物培养基预测文章
1. 该网站的意义
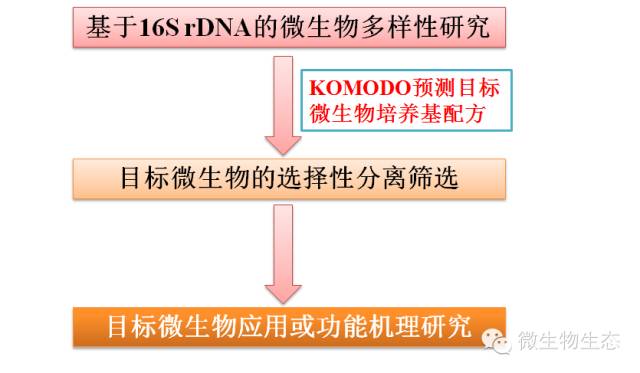
这是个很了不起的工作,说他了不起是因为目前环境中的微生物绝大多数(90%~99%)是不可培养的[2],因对不同微生物培养条件(尤其是培养基配方)的不了解,阻碍了微生物的分离培养。随着高通量测序技术的发展,目前有大量文章是在做基于16S rDNA的微生物非培养多样性,但拿不到菌种使得目标微生物的应用和机理研究成为空中楼阁。有了这个网站就可以有依据的预测目标微生物的培养基配方了。因此可将基于16S rDNA的微生物多样性数据与目标菌株的分离培养有机衔接起来,大大加速从生态研究到微生物资源挖掘的进程,研究思路如下图3。
图3. KOMODO将非培养的16S rDNA多样性研究与目标微生物分离有机结合起来
2. 该网站的原理
该网站结合了NCBI的微生物分类和微生物培养基数据库(DSMZ)等(图4A),依据传递预测模式(Transitive prediction schema),即若微生物A和B均可利用培养基1、微生物B和C可利用培养基2,微生物C可利用培养基3,那么依次传递推理,微生物A应该也可以利用培养基3(图4B);和协同过滤预测(collaborative filtering predictor),即根据16S rDNA的系统进化相似性,根据已知培养基的物种来推断其相似性高的近缘物种的培养基(图4C)。
图4 KOMODO网站工作原理
3. 该网站的用法
(1) 打开链接(http://komodo.modelseed.org/)进入KOMODO页面,如下:

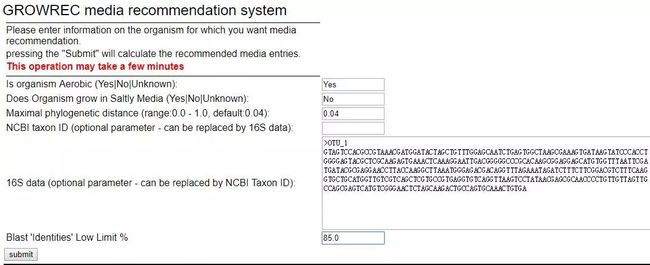
(2) 输入相应内容后,点击提交。如下图以某土壤样品中的16S rDNA为例。
内容填写时注意:
Is organism Aerobic (Yes|No|Unknown):根据实际情况填写物种的需氧情况,只能填Yes或No或Unknown;
Does Organism grow in Saltly Media (Yes|No|Unknown): 根据实际情况填写物种的盐度情况,只能填写Yes或No或Unknown;
Maximal phylogenetic distance (range:0.0 - 1.0, default:0.04):可输入0-1之间的数字,一般默认即可。
NCBI taxon ID (optional parameter - can be replaced by 16S data):和下面的16S data (optional parameter - can be replaced by NCBI Taxon ID):两者只能输入一个,建议最好直接在16S data右边的框里输入FASTA格式的序列即可。
Blast 'Identities' Low Limit % 表示与你16S序列相似的最低限是多少,默认85%,可根据实际情况适当提高。
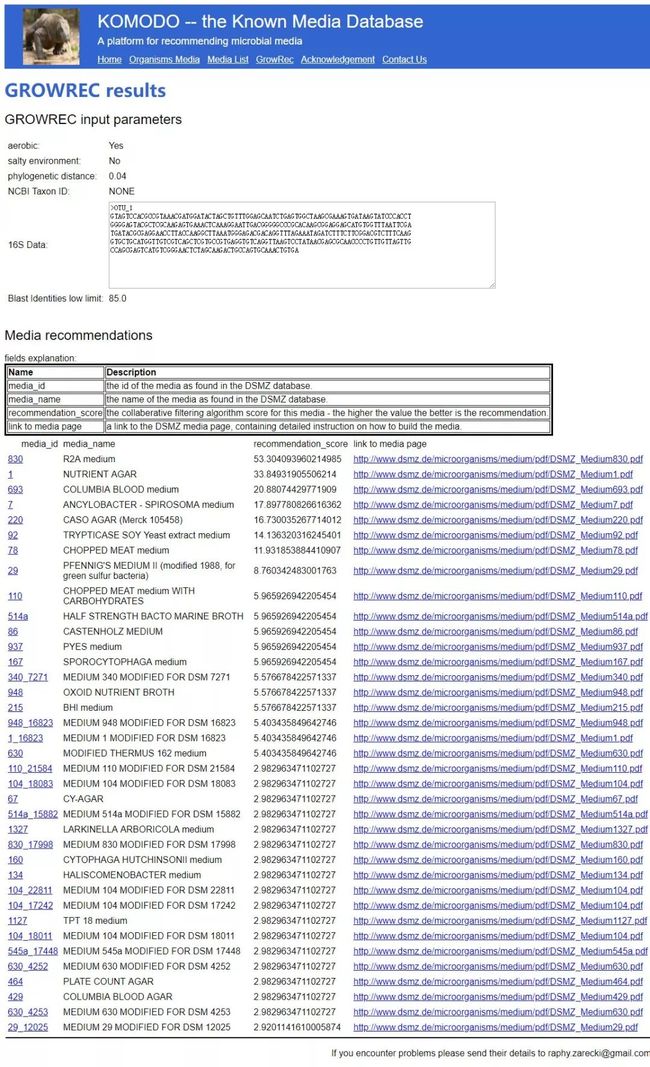
如上图中,填写完整后,点击左下角“submit”提交后等几分钟即可得预测结果,如下图:
网站会根据你填写的信息给出推荐的不同培养基,如上图例子中给出了四种培养基,点击培养基对应的链接可以直接下载PDF版的培养基配方,如下图PDF版推荐培养基示例:
 哦,对了,网站的作者很欢(dou)乐(bi),因为,如果你输错了参数就会出现下面的一幕。。。。。
哦,对了,网站的作者很欢(dou)乐(bi),因为,如果你输错了参数就会出现下面的一幕。。。。。



参考文献:
[1] Oberhardt M A, Zarecki R, Gronow S, et al. Harnessing the landscape of microbial culture media to predict new organism-media pairings[J]. Nature communications, 2015, 6.
[2] Schleifer K H. Microbial diversity: facts, problems and prospects[J]. Systematic and applied microbiology, 2004, 27(1): 3-9.
答谢:
感谢微生物物种分类领域的专家刘阳博士对此文章的推荐
![]()
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()