Linux下从NCBI批量下载SRA数据的sra和aspera方法
Minus_yao 2018.04.25 [email protected]
#从NCBI下载SRA数据,最近在疯狂下载宏基因组数据,试着解决一下这个问题~
方法一:
软件准备:
使用ncbi提供的下载工具sratoolkit,下载到本地服务器上
Wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/2.9.0/sratoolkit.2.9.0-centos_linux64.tar.gz
解压:
tar -zxvf sratoolkit.2.9.0-centos_linux64.tar.gz
添加环境变量
echo'exportPATH=/home/gyao/program/sratoolkit.2.9.0-centos_linux64.tar.gz/bin:$PATH'>> ~/.bashrc
source~/.bashrc
数据列表准备:
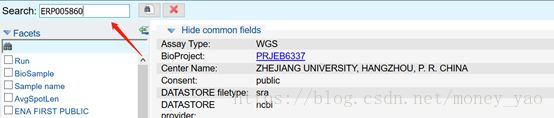
以ERP005860数据为例,包含314个sra文件,先从NCBI上拿到SRR_Acc_List.txt文件。下载这个AccessionList文件,里面是314行形如ERR526291的编号
下载命令行:
nohup/home/gyao/program/sratoolkit/sratoolkit.2.9.0-centos_linux64/bin/prefetch -a"/home/gyao/.aspera/connect/bin/ascp|/home/gyao/.aspera/connect/etc/asperaweb_id_dsa.openssh"--ascp-options "-QT -l 40m" --option-file SRR_Acc_List.txt &
(注意--option-file应在当前文件夹下,如前添加了环境变量,可直接使用prefetch;不过,默认文件下载的位置是~/ncbi/public/sra,试图修改时很麻烦遂放弃)。
这个方法能够批量下载,但是用了几天发现速度变得超级慢,还莫名其妙断掉,想其他办法,选择aspera下载。
软件准备:
下载软件:
Wgethttp://download.asperasoft.com/download/sw/connect/3.7.4/aspera-connect-3.7.4.147727-linux-64.tar.gz
解压:
tarzxvf aspera-connect-3.7.4.147727-linux-64.tar.gz
安装:
bashaspera-connect-3.7.4.147727-linux-64.sh
添加环境变量:
echo'export PATH=~/.aspera/connect/bin:$PATH' >> ~/.bashrc
source~/.bashrc
命令ascp –help查看帮助文档说明安装成功。
从NCBI上单个文件下载命令:
ascp-i /home/gyao/.aspera/connect/etc/asperaweb_id_dsa.openssh -k 1 -T -l 200m [email protected]:/sra/sra-instant/reads/ByRun/sra/ERR/ERR526/ERR526291/ERR526291.sra./download_dir/
速度还是挺快的~
关键的问题是解决上述314个文件批量下载,我的方法是利用前面下载的SRR_Acc_List.txt文件,用python写一个简单的脚本,批量执行断点下载。
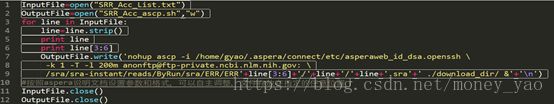
基本思路是:写一个translate.py脚本,将SRR_Acc_List.txt内容转换为ascpera可识别的格式,并写入SRR_Acc_ascp.sh文件(这是一个可执行脚本.sh),bash运行SRR_Acc_ascp.sh脚本文件。
Translate.py的coding:输出的SRR_Acc_ascp.sh文件内容(部分)如下:
命令行:
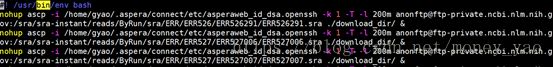
nohup bash SRR_Acc_ascp.sh &
可以关机回去睡觉了,目前速度还不错,希望明天早上起来有好消息~