医学图像处理(一)——分割中常用的度量指标
下列参数主要参考自MICCAI2007
- 首先定义以下标识符:Vgt代表的是ground truth的分割结果,Vpred代表的是预测的分割结果.
- DICE: 这个相信大家最熟悉,因为使用的频率应该是最多的.它的定义如下所示. 直观上来说代表的是两个体相交的面积占总面积的比值.完美分割该值为1. 【1】
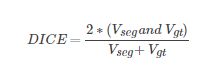
GT(ground truth)图像:我理解的就是含有理论分割结果的图像,用来和结果图像进行比较的参照图像。那么GT图像怎么来,一般通过专家手工勾画出来,得到理论值。当然为避免偶然性,你可以选择多个专家,得到多个GT分割结果,取各项评估参数的平均值。【2】
分割精度:个人理解就是分割准确的面积占GT图像中真实面积的百分比。找了半天找了这么个公式(如下)。
![]()
不难理解,其中Rs表示专家手工勾画出的分割图像的参考面积,Ts表示算法分割得到的图像的真实面积,|Rs-Ts|表示错误分割的像素点个数。
过分割率:即分割在GT图像参考面积之外的像素点的比率,计算公式如下:
![]()
含义同上,Os表示本不应该包含在分割结果中的像素点个数,实际却在分割结果中的像素点个数。换句话讲,Os中的像素点出现在实际分割图像中,但不出现在理论分割图像Rs中。
欠分割率:即分割在GT图像参考面积之中欠缺的像素点的比率,计算的公式如下:
![]()
含义同上,Us表示本应该包含在分割结果中的像素点个数,实际却不在分割结果中的像素点个数。换句话讲,Us中的像素点出现在理论分割图像中,但不出现在实际分割图像中。
代码(由于当时时间紧张,很多可以封装成函数):
获得肝血管GT图像:
import pydicom
from pylab import *
import os
import PIL.Image as image
from Anisotropicdiffusion import *
PathDicom = "./PATIENT_DICOM20/" # 与python文件同一个目录下的文件夹
lstFilesDCM1 = []
number1 = 0
for dirName, subdirList, fileList1 in os.walk(PathDicom):
for filename in fileList1:
lstFilesDCM1.append(os.path.join(dirName, filename))
number1 += 1
RefDs = pydicom.read_file(lstFilesDCM1[0])
ConstPixelSpacing = (float(RefDs.PixelSpacing[0]), float(RefDs.PixelSpacing[1]), float(RefDs.SliceThickness))
print(ConstPixelSpacing)
PathDicom = "./liver20/" # 与python文件同一个目录下的文件夹
lstFilesDCM2 = []
number2 = 0
for dirName, subdirList, fileList2 in os.walk(PathDicom):
for filename in fileList2:
lstFilesDCM2.append(os.path.join(dirName, filename))
number2 += 1
PathDicom = "./artery20/" # 与python文件同一个目录下的文件夹
lstFilesDCM3 = []
number3 = 0
for dirName, subdirList, fileList3 in os.walk(PathDicom):
for filename in fileList3:
lstFilesDCM3.append(os.path.join(dirName, filename))
number3 += 1
PathDicom = "./portalvein20/" # 与python文件同一个目录下的文件夹
lstFilesDCM4 = []
number4 = 0
for dirName, subdirList, fileList4 in os.walk(PathDicom):
for filename in fileList4:
lstFilesDCM4.append(os.path.join(dirName, filename))
number4 += 1
PathDicom = "./venacava20/" # 与python文件同一个目录下的文件夹
lstFilesDCM5 = []
number5 = 0
for dirName, subdirList, fileList5 in os.walk(PathDicom):
for filename in fileList5:
lstFilesDCM5.append(os.path.join(dirName, filename))
number5 += 1
if number1 != number2:
print("fileerro")
for i in range(number1):
dcm1 = pydicom.read_file(lstFilesDCM1[i])
dcm1.image = dcm1.pixel_array * dcm1.RescaleSlope + dcm1.RescaleIntercept
slices = []
slices.append(dcm1)
img1 = slices[int(len(slices) / 2)].image.copy()
dcm2 = pydicom.read_file(lstFilesDCM2[i])
dcm2.image = dcm2.pixel_array * dcm2.RescaleSlope + dcm2.RescaleIntercept
slices = []
slices.append(dcm2)
img2 = slices[int(len(slices) / 2)].image.copy()
dcm3 = pydicom.read_file(lstFilesDCM3[i])
dcm3.image = dcm3.pixel_array * dcm3.RescaleSlope + dcm3.RescaleIntercept
slices = []
slices.append(dcm3)
img3 = slices[int(len(slices) / 2)].image.copy()
dcm4 = pydicom.read_file(lstFilesDCM4[i])
dcm4.image = dcm4.pixel_array * dcm4.RescaleSlope + dcm4.RescaleIntercept
slices = []
slices.append(dcm4)
img4 = slices[int(len(slices) / 2)].image.copy()
dcm5 = pydicom.read_file(lstFilesDCM5[i])
dcm5.image = dcm5.pixel_array * dcm5.RescaleSlope + dcm5.RescaleIntercept
slices = []
slices.append(dcm5)
img5 = slices[int(len(slices) / 2)].image.copy()
img = np.zeros((512, 512))
img30 = np.zeros((512, 512))
img40 = np.zeros((512, 512))
img50 = np.zeros((512, 512))
height, width = img2.shape
for k in range(height):
for j in range(width):
if img2[k, j] != 0:
img2[k, j] = 1
if img3[k, j] != 0:
img3[k, j] = 1
if img4[k, j] != 0:
img4[k, j] = 1
if img5[k, j] != 0:
img5[k, j] = 1
img1[k, j] *= img2[k, j]
img30[k, j] = img1[k, j]*img3[k, j]
img40[k, j] = img1[k, j]*img4[k, j]
img50[k, j] = img1[k, j]*img5[k, j]
img[k, j] = img30[k, j]+img40[k, j]+img50[k, j]
img = np.uint8(img)
img6 = image.fromarray(img)
filename = "D:/Pychram/liverpicture/mask_" + fileList1[i] + ".png"
img6.save(filename)
print(i)计算分割精度:
import os
import PIL.Image as Image
from Anisotropicdiffusion import *
PathDicom = "C:\\Users\\LCSH\\Desktop\\Pychram\\liverpicture" # 与python文件同一个目录下的文件夹
lstFilesDCM1 = []
number1 = 0
for dirName, subdirList, fileList1 in os.walk(PathDicom):
for filename in fileList1:
lstFilesDCM1.append(os.path.join(dirName, filename))
number1 += 1
PathDicom = "C:\\Users\\LCSH\\Desktop\\Pychram\\liverpicture1" # 与python文件同一个目录下的文件夹
lstFilesDCM2 = []
number2 = 0
for dirName, subdirList, fileList2 in os.walk(PathDicom):
for filename in fileList2:
lstFilesDCM2.append(os.path.join(dirName, filename))
number2 += 1
p = 0
for i in range(number1):
img = Image.open(lstFilesDCM1[i])
img1 = img.load()
img = Image.open(lstFilesDCM2[i])
img2 = img.load()
f = h = 0
height, width = img.size
for k in range(height):
for j in range(width):
if img1[k, j] > 0:
f += 1
if img1[k, j] > 0 and img2[k, j] == 0:
h += 1
g = h/f
p += g
print(i)
L = p / number1
print(L)- VOE: volumetric overlap error(体积重叠误差). 这个和DICE比较类似,它的定义如下所示, 相比于DICE它将and操作换成了减法操作以此来代表错误率
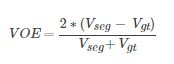
- RVD: relative volume difference( 相对体积差). 这个代表的是Vpred和Vgt体积之间的差异,它的定义如下所示:
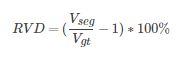
- ASD: average symmetric surface distance(平均对称表面距离). 这个的定义相比前几个略为复杂一些. 我们先定义Apred代表的是预测的Vpred中的边界的像素,同样的我们可以得到Agt的定义.然后我们对Bpred的定义如下.同理我们可以得到Bgt的定义.那么ASD的定义也就如下所示.
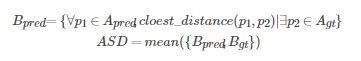
- MSD: maximum symmetric surface distance(最大对称表面距离). 这个定义和ASD定义比较类似,只不过把计算平均的操作替换成了计算最大值的操作.
转自:
【1】医学图像分割中常用的度量指标
【2】图像分割结果的评估---DICE参数