Tidyverse|数据列的分分合合,爱恨情仇
本文首发于“生信补给站”Tidyverse|数据列的分分合合,一分多,多合一
TCGA数据挖掘可做很多分析,前期数据“清洗”费时费力但很需要。
比如基因列为ID的需要转为常见的symbol,基因列为symbol|ID的就需要拆开了!
excel分列可以解决,但是表达量数据较大,且excel容易产生“数据变形”。
一, 载入数据,R包
使用TCGA下载的数据,使用以下几行几列, 作为示例
library(tidyverse)
data <- read.csv("separate.csv",header = TRUE, check.name = FALSE)
head(data)
ID ID2 TCGA-18-3406-01A-01R-0980-07 TCGA-18-3407-01A-01R-0980-07
1 A1BG|1 A1BG/1 741.6929 46.7127
2 A1CF|29974 A1CF/29974 0.0000 0.4757
3 A2BP1|54715 A2BP1/54715 0.0000 0.0000
4 A2LD1|87769 A2LD1/87769 170.2362 118.4063
5 A2ML1|144568 A2ML1/144568 128.3465 1413.4158
6 A2M|2 A2M/2 9074.6772 11310.1713
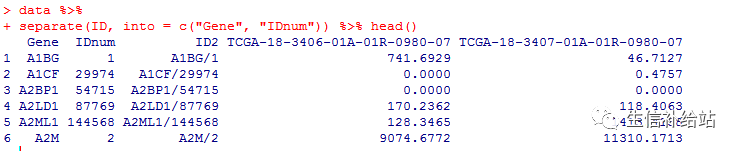
可以看到第一列的ID,和人为添加的ID2,名称不规则,我们只需要前面的基因名。
二,合久可分 - 一列拆多列
使用separate函数, 将**“指定”**分隔符出现的位置一列分成多列
2.1 默认,不指定分隔符
data %>%
separate(ID, into = c("Gene", "IDnum")) %>% head()
2.2 指定分隔符且保留原始列
data %>%
separate(ID2, into = c("Gene2", "IDnum2"),sep = "/",remove = FALSE) %>%
head()
2.3 特殊字符的指定方式
data %>%
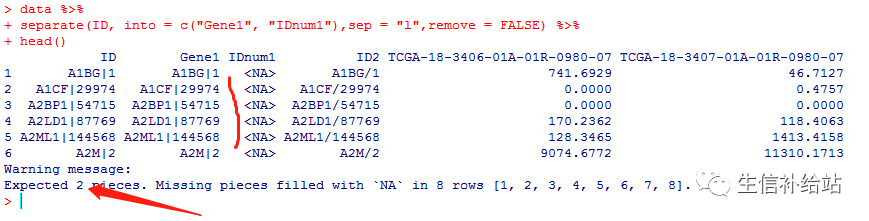
separate(ID, into = c("Gene1", "IDnum1"),sep = "l",remove = FALSE) %>%
head()
按照同样的方式,想把"|"分隔的ID拆分,发现报错。?黑人问号脸
有问题可以先?separate或者help(separate)查询帮助函数,"|"为特殊字符,可以使用"[|]"括起来或者"\\|",均可:
data %>%
separate(ID, into = c("Gene1", "IDnum1"),sep = "[|]",remove = FALSE) %>% head()
#或者
data2 <- data %>%
separate(ID, into = c("Gene1", "IDnum1"),sep = "\\|",remove = FALSE)
2.4,按照第几个字符拆
根据第几个字符拆分,适合数据规整的,,,
可以用来将TCGA中的sampleID转为常见的16位,需要先转置
#
data2 %>% select(Gene1,contains("TCGA")) %>% #选择指定列
column_to_rownames(var = "Gene1") %>% # 将Gene1列转为rownames
t() %>% as.data.frame() %>% #数据转置,样本为行名
rownames_to_column(var="Sample") %>% #行名变为数据中的列
separate(Sample, into = c("Sample", "bar"),sep = 16) %>% #按照规则取前16个字符
select(-bar) #去掉分割后不需要的bar列
可参考:盘一盘Tidyverse| 筛行选列之select,玩转列操作
Tips:
- 1)可以先默认试一下,如2.1所示;
- 2)使用R的帮助,一定!
三,分久必合-多列合并一列
使用unite函数, 可将多列按照**“指定”**分隔符合并为一列
data %>%
unite(ID_new, ID:ID2, sep = "_") %>% head()
◆ ◆ ◆ ◆ ◆
精心整理(含图版)|你要的全拿走!有备无患
![]()