libtorch+VS2015配置与应用
背景:用Unet训练了脑肿瘤分割模型,导出了pytorch中的模型与参数.pth文件。
目的:将.pth文件应用于C++中,形成分割功能,移植到实验室成员一同开发医学图像软件中。
环境配置:pytorch 1.3 + libtorch 1.3 + VS 2015 + ITK 4.13 + cmake 3.12
ITK 4.13与VS2015的配置方法可以在我另一篇文档或在社区中寻找配置方法,这里不再赘述,使用ITK的目的有两个:1、我比较熟悉ITK读入多种图片格式的方法;2、ITK提供比较多的图像处理方式,可对体数据预处理。
移植过程一共分成4步:
1、输出.pt文件,在C++上测试模型;
2、得出测试结果,将结果转换成掩模图像,得到每个类别的轮廓线;
3、将轮廓线覆盖到原图像上;
4、用QT写软件界面+功能接口;
1st,输出.pt文件:
import torch
import torch.nn as nn
from unet import Unet #根据自己写的模型来导入
saved_model_path = 'E:/Training/ckpt1/best_epoch.pth'
cuda_available = torch.cuda.is_available()
if cuda_available:
device_ids = [0] # multi-GPU, but i have only one
torch.cuda.set_device(device_ids[0])
def load_model():
net = Unet(1, 5, 32 ) #我这里是 inch = 1, outch = 5
if cuda_available:
net = net.cuda()
state_dict = torch.load(saved_model_path)
net.load_state_dict(state_dict, strict=False)
return net
net = load_model()
net.eval()
example = torch.rand(1, 1, 240, 240) #随便搞一个输入,但务必请符合你的网络的输入才行
traced = torch.jit.trace(net, example)
traced.save('E:/Training/ckpt1/best_epoch.pt')
成功追踪到的模型如图所示:
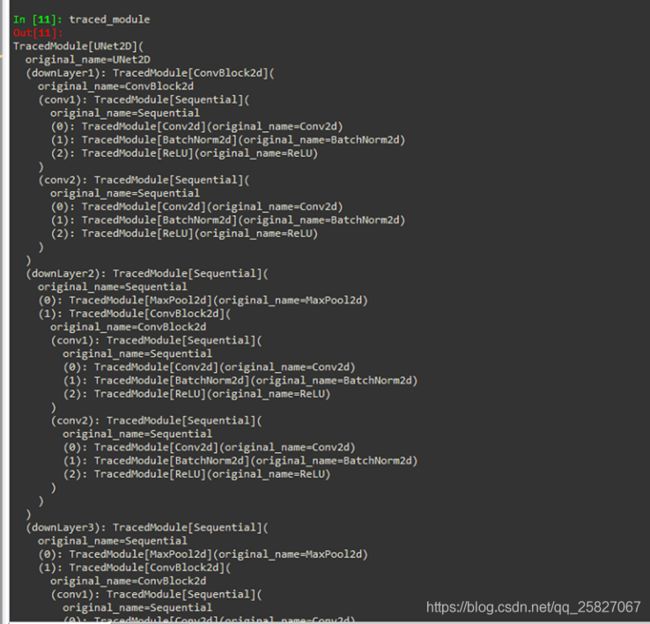
注意的问题:
如果训练过程使用了nn.DataParallel,即多GPU并行训练,则导出的pt文件可能在后续C++使用中出现问题(至少我是这样),所以并不适合用在多GPU并行训练的模型中,目前解决的方法尚未找到(能力有限)

2nd,在VS2015中使用libtorch:
1、下载libtorch: 注意CUDA版本和DEBUG还是RELEASE版本,项目需求我用了DEBUG的

(测试了好多例,心疼自己的网速,等了好久才下完)
2、解压,在文件夹内创建文件夹example,在example中创建build文件夹;
3、创建C++文件和CMAKELISTS.txt,并将.pt文件放在同一文件夹
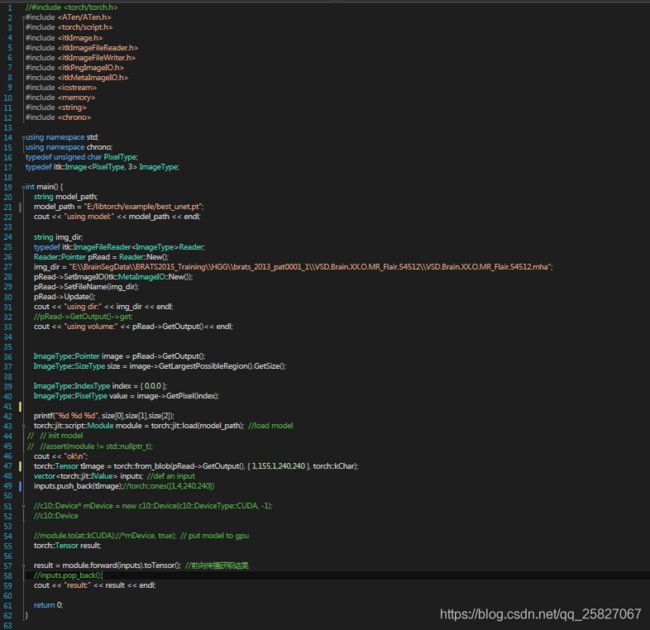
请按照自己的项目需求写Cpp文件
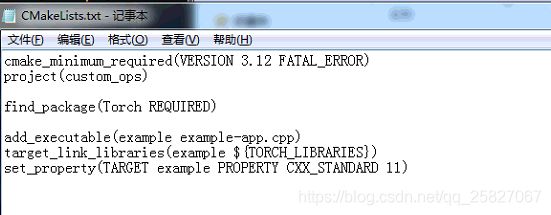
4、在命令行输入(记得提前安装好cmake)
cmake –DCMAKE_PREFIX_PATH=(libtorch文件夹路径) -DCMAKE_BUILD_TYPE=Debug(如果是Release就改成Release) –G “Visual Studio 14 Win64” ..
5、makefile之后出现这些内容,打开example.vcxproj
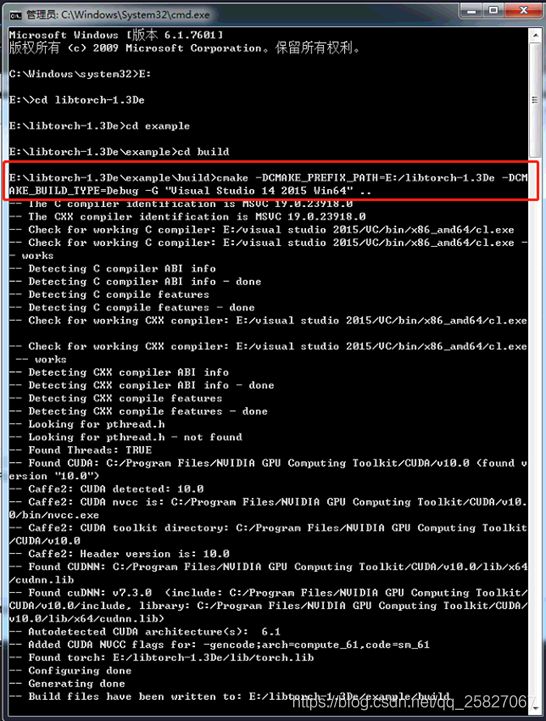
成功的输出

打开project后有几个地方需要注意:
1、启动项更改

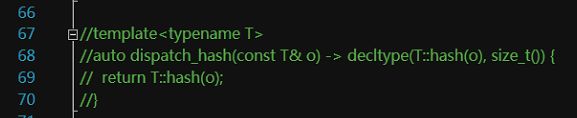
2、如果出现C2995错误,屏蔽掉hash.h这段代码
3、前向传播时Runtime::Error,可能是模型的问题,也就是pt文件没有转换好
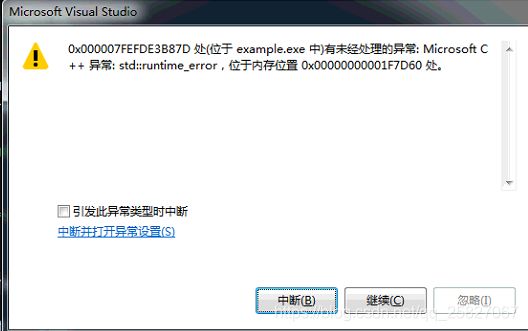
4、shared_ptr错误,版本问题,删掉替换最新写法即可
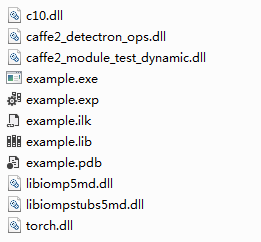
5、需将dll文件复制进debug文件夹中:
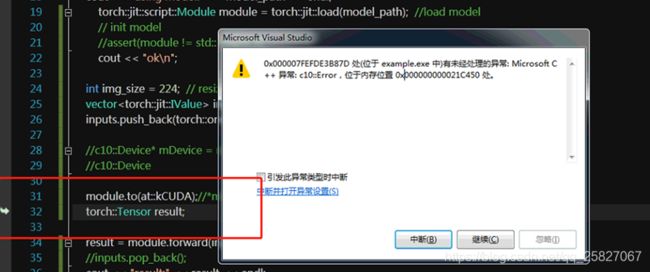
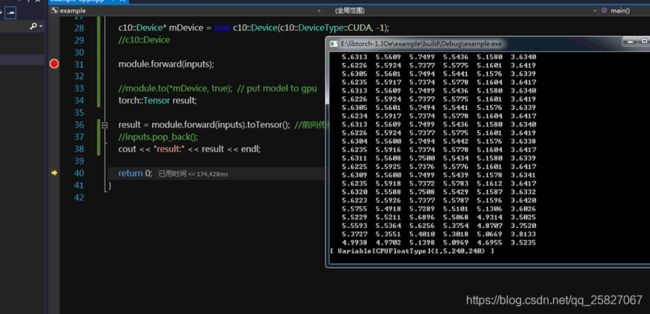
成功复现的输出,因为用GPU跑会出现C10.dll错误,所以这里用CPU先复现一下:
后面用ITK读文件的地方有空继续记录(太懒~)