RAxML进化树构建换代啦(2018.11)
RAxML一直是最大似然法(maximum likelihood)建树的经典工具,其由来自德国海德堡理论科学研究所(Heidelberg Institute for Theoretical Studies)的Alexandros Stamatakis开发。近年来,其江湖地位也受到来自其他软件,尤其是IQ-Tree的挑战(关于IQTREE更多内容可参见新一代建树工具IQ-Tree介绍)。Zhou et al. 2018对RAxML,IQ-TREE,FastTree,Phyml四个最大似然法建树软件的实际效果和表现进行了系统比较,其中一个结论是IQTREE在准确性方面要略胜一筹:
IQ-TREE consistently achieved the best-observed likelihoods for all data sets, and RAxML/ExaML was a close second
近日,RAxML的升级版,raxml-ng的预印本文章在bioRxiv平台发布!当然,这里的ng和二代测序无关,而是代表next generation 的raxml。为简明起见,下文用raxml-ng表示二代raxml,而用RAxML表示旧版raxml。
文章简述
本文短小精悍,由Stamatakis课题组的Alexey Kozlov做通讯作者,主要对raxml-ng的新的特点进行了介绍,包括但不限于:
1. 速度较RAxML有较大幅度提升。raxml-ng对算法有改进,其中也借鉴了IQTREE的部分思路。作者利用Zhou的数据将raxml-ng与IQTREE进行了比较,发现raxml-ng在可以得出更准确的结果的同时其速度是IQTREE的1.3到4.5倍(这里的速度应该不包括IQTREE的ultrafast bootstrap approximation,更多信息请见下文)。
2. RAxML对于DNA替换模型的吝啬是其饱受诟病的一个原因。而这一问题在raxml-ng中得到了完美解决。raxml-ng现在支持22个核酸进化模型。
3. FreeRate model of rate heterogeneity (Yang, 1995)得到支持
4. Transfer bootstrap得到支持,改法是一种年初发表在nature上的改进版计算bootstrap的方法(Lemoine et al 2018)
5. 多线程效率上有一定提升
使用简介
大家可以在github上找到升级后的raxml-ng,地址是:
https://github.com/amkozlov/raxml-ng
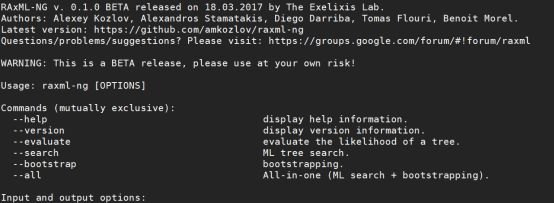
下载后,我们在终端键入raxml-ng,便会出现下面的界面:
这里我们非常简明地说一下raxml-ng的一些基础操作。
从最简单的开始。一个DNA比对,核苷酸替代模型为GTR,速率异质性(rate heterogeneity)设置为gamma分布,不做bootstraping,命令如下:
raxml-ng --msa seq.aln --model GTR+G
这样会在当前文件夹下生成包括seq.aln.raxml.log在内的几个文件:
如果需要在当前文件夹下继续用raxml-ng建树,则会收到如下提示:
ERROR: Result files for the run with prefix `seq.aln` already exist!
解决办法是在运行时加--redo。
实际操作中,大家时常为了检查树的鲁棒性(robustness)进行自展(bootstrap)检验。在raxml-ng里,这一操作通过--bs-trees参数实现。如下,进行1000次bootstrapping抽样。
raxml-ng --msa seq.aln --model GTR+G --bs-trees 1000
输出文件中可见计算出的带有bootstrap值的最优树:seq.aln.raxml.support。.log文件为日志。其他的输出文件含义由于篇幅所限大家可以在文末给出的官方说明链接里查询。
请注意,raxml-ng里进化模型的基本格式是:
raxml-ng提供了”--all”这样一个参数,all-in-one打包操作,最大似然搜索+bootstrapping,默认bootstrap次数是100次
如果希望输出结果到指定文件夹,请使用--refix参数。不过前提是该文件夹存在,raxml-ng不会自己生成文件夹。比如
raxml-ng --msa seq.aln --model GTR+G --bs-trees 1000 –prefix out/seq
指定起始树:--tree topology.tree
指定数据类型:--data-type (默认是自动检测)
给定线程:--threads
Transfer bootstrap:--bs-metric TBE
不确定需要多少次bootstraps,那么可以交由raxml-ng帮你决断:--bs-trees autoMRE
限定拓扑结构:--constraint-tree
Partitioned alignment的输入:--model partition.txt
潜在短板
raxml-ng不支持IQ-Tree的超快自展近似ultrafast bootstrap approximation(好像该方法和IQtree的算法有关,特异性较高,无法移植到raxml-ng)。所以,虽然raxml-ng的作者在文中表示raxml-ng的运算速度快过IQTREE数倍,但如果在IQTREE里使用1000次超快自展的话,那么小编窃以为IQTREE还是要略胜一筹吧(更多信息请见 新一代建树工具IQ-Tree介绍)?据悉,raxml-ng方面也在尝试开发有别于传统的bootstrap的更加快速的拓扑结构鲁棒性检验的方法,期待在未来的版本里可以使用到。另外,raxml-ng暂时还不支持自动检测最优进化模型,而这一点在IQ-TREE通过modelfinder(Kalyaanamoorthy, et al. 2017) 获得了很好的支持。
此外,raxml-ng目前尚有一些旧版raxml具备的特征没有完成开发。作者特别提到其中之一是RAxML里面针对速率异质性的CAT模型在raxml-ng暂时不支持,但会在未来得到开发。
其他资源
raxml-ng更多的内容,包括raxml-ng和RAxML参数对比,大家可以参见以下链接,十分详尽:
https://github.com/amkozlov/raxml-ng/wiki
除standalone之外,raxml-ng还支持在线运行,该服务由Swiss Institute of Bioinformatics负责,网址如下:https://raxml-ng.vital-it.ch/#/
小编这里把自己关于raxml-ng的一些心得体会和大家分享一下,但很多内容都是浅尝辄止,且不少东西未有涉及。错漏之处希望大家指正。也欢迎用过的读者在下方和大家分享一下你的经验吧。
引文
1. Kalyaanamoorthy S, Minh BQ, Wong TKF, von Haeseler A, Jermiin LS. ModelFinder: fast model selection for accurate phylogenetic estimates. Nat Methods. 2017;14(6):587-589
2. Lemoine F, Domelevo Entfellner JB, Wilkinson E, et al. Renewing Felsenstein's phylogenetic bootstrap in the era of big data. Nature. 2018;556(7702):452-456
3. Yang Z. A space-time process model for the evolution of DNA sequences. Genetics. 1995;139(2):993-1005
4. Zhou X, Shen XX, Hittinger CT, Rokas A. Evaluating Fast Maximum Likelihood-Based Phylogenetic Programs Using Empirical Phylogenomic Data Sets. Mol Biol Evol. 2018;35(2):486-503
本文为作者原创,原载于生信人微信公众号