数据挖掘——层次聚类(Hierarchical clustering)学习及python实现
文章目录
- 一、前言
- 二、自底向上的层次算法
- 三、 python实现层次聚类
- 四、使用Sklearn中的层次聚类
- 五、使用Scipy库中的层次聚类
- (1). linkage(y, method=’single’, metric=’euclidean’)
- (2).fcluster(Z, t, criterion=’inconsistent’, depth=2, R=None, monocrit=None)
- 六、层次聚类的优缺点
- 参考资料:
一、前言
层次聚类(Hierarchical Clustering)是聚类算法的一种,通过计算不同类别数据点间的相似度来创建一棵有层次的嵌套聚类树。在聚类树中,不同类别的原始数据点是树的最低层,树的顶层是一个聚类的根节点。层次聚类算法相比划分聚类算法的优点之一是可以在不同的尺度上(层次)展示数据集的聚类情况。
根据创建聚类树有的两种方式:自下而上合并和自上而下。基于层次的聚类算法可以分为:凝聚的(Agglomerative)或者分裂的(Divisive)。
- 自下而上法就是一开始每个个体(object)都是一个类,然后根据linkage寻找同类,最后形成一个“类”。
- 自上而下法就是反过来,一开始所有个体都属于一个“类”,然后根据linkage排除异己,最后每个个体都成为一个“类”。
这两种路方法没有孰优孰劣之分,只是在实际应用的时候要根据数据特点以及你想要的“类”的个数,来考虑是自上而下更快还是自下而上更快。至于根据Linkage判断“类”的方法就是最短距离法、最长距离法、中间距离法、类平均法等等(其中类平均法往往被认为是最常用也最好用的方法,一方面因为其良好的单调性,另一方面因为其空间扩张/浓缩的程度适中)。为弥补分解与合并的不足,层次合并经常要与其它聚类方法相结合,如循环定位。
层次聚类方法中比较新的算法有BIRCH(Balanced Iterative Reducingand Clustering Using Hierarchies利用层次方法的平衡迭代规约和聚类)主要是在数据量很大的时候使用,而且数据类型是numerical。首先利用树的结构对对象集进行划分,然后再利用其它聚类方法对这些聚类进行优化;ROCK(A Hierarchical ClusteringAlgorithm for Categorical Attributes)主要用在categorical的数据类型上;Chameleon(A Hierarchical Clustering AlgorithmUsing Dynamic Modeling)里用到的linkage是kNN(k-nearest-neighbor)算法,并以此构建一个graph,Chameleon的聚类效果被认为非常强大,比BIRCH好用,但运算复杂度很高,O(n^2)。
二、自底向上的层次算法
层次聚类的合并算法通过计算两类数据点间的相似性,对所有数据点中最为相似的两个数据点进行组合,并反复迭代这一过程。简单的说层次聚类的合并算法是通过计算每一个类别的数据点与所有数据点之间的距离来确定它们之间的相似性,距离越小,相似度越高。并将距离最近的两个数据点或类别进行组合,生成聚类树。
绝大多数层次聚类属于凝聚型层次聚类, 它的算法流程如下:
- (1) 将每个对象看作一类,计算两两之间的距离;
- (2) 将距离最小的两个类合并成一个新类;
- (3) 重新计算新类与所有类之间的距离;
- (4) 重复(2)、(3),直到所有类最后合并成一类。
整个过程就是建立一棵树,在建立的过程中,可以在步骤四设置所需分类的类别个数,作为迭代的终止条件,毕竟都归为一类并不实际。
相似度的计算
层次聚类使用欧式距离来计算不同类别数据点间的距离(相似度)。
D = ( x 1 − x 2 ) 2 + ( y 1 − y 2 ) 2 D = \sqrt {(x_{1}-x_{2})^{2}+(y_{1}-y_{2})^{2} } D=(x1−x2)2+(y1−y2)2
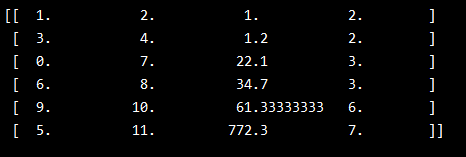
实例:数据点如下
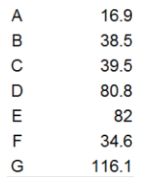
分别计算欧式距离值(矩阵)
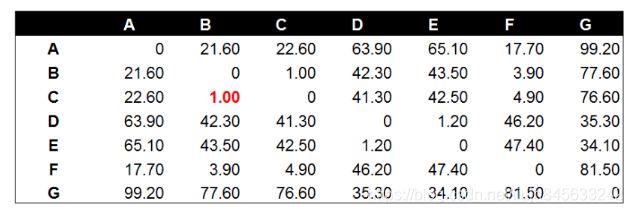
将数据点B与数据点C进行组合后,重新计算各类别数据点间的距离矩阵。数据点间的距离计算方式与之前的方法一样。这里需要说明的是组合数据点(B,C)与其他数据点间的计算方法。当我们计算(B,C)到A的距离时,需要分别计算B到A和C到A的距离均值。
D = ( B − A ) 2 + ( C − A ) 2 2 = 21.6 + 22.6 2 \mathbf {D=\frac {\sqrt {(B-A)^{2}} + \sqrt {(C-A)^{2}}}{2}=\frac {21.6 +22.6} {2}} D=2(B−A)2+(C−A)2=221.6+22.6
经过计算数据点D到数据点E的距离在所有的距离值中最小,为1.20。这表示在当前的所有数据点中(包含组合数据点),D和E的相似度最高。因此我们将数据点D和数据点E进行组合。并再次计算其他数据点间的距离。
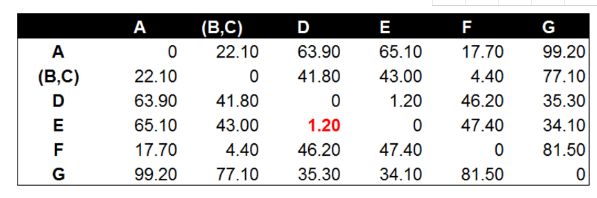
后面的工作就是不断的重复计算数据点与数据点,数据点与组合数据点间的距离。这个步骤应该由程序来完成。这里由于数据量较小,我们手工计算并列出每一步的距离计算和数据点组合的结果。
聚类之间(两个组合数据点间)的相似度
计算两个组合数据点间距离的方法有三种,分别为Single Linkage,Complete Linkage 和 Average Linkage。在开始计算之前,我们先来介绍下这三种计算方法以及各自的优缺点。
-
Single Linkage(单连接):方法是将两个组合数据点中距离最近的两个数据点间的距离作为这两个组合数据点的距离。这种方法容易受到极端值的影响。两个很相似的组合数据点可能由于其中的某个极端的数据点距离较近而组合在一起。
-
Complete Linkage(全连接):Complete Linkage的计算方法与Single Linkage相反,将两个组合数据点中距离最远的两个数据点间的距离作为这两个组合数据点的距离。Complete Linkage的问题也与Single Linkage相反,两个不相似的组合数据点可能由于其中的极端值距离较远而无法组合在一起。
-
Average Linkage(平均连接):Average Linkage的计算方法是计算两个组合数据点中的每个数据点与其他所有数据点的距离。将所有距离的均值作为两个组合数据点间的距离。这种方法计算量比较大,但结果比前两种方法更合理。
我们使用Average Linkage计算组合数据点间的距离。下面是计算组合数据点(A,F)到(B,C)的距离,这里分别计算了(A,F)和(B,C)两两间距离的均值。
D = ( A − B ) 2 + ( A − C ) 2 + ( F − B ) 2 + ( F − C ) 2 4 \mathbf {D=\frac {\sqrt {(A-B)^{2}} + \sqrt {(A-C)^{2}}+\sqrt {(F-B)^{2}} + \sqrt {(F-C)^{2}}}{4}} D=4(A−B)2+(A−C)2+(F−B)2+(F−C)2
三、 python实现层次聚类
import math
import numpy as np
import sklearn
from sklearn.datasets import load_iris
def euler_distance(point1: np.ndarray, point2: list) -> float:
"""
计算两点之间的欧式距离,支持多维
"""
distance = 0.0
for a, b in zip(point1, point2):
distance += math.pow(a - b, 2)
return math.sqrt(distance)
class ClusterNode(object):
def __init__(self, vec, left=None, right=None, distance=-1, id=None, count=1):
"""
:param vec: 保存两个数据聚类后形成新的中心
:param left: 左节点
:param right: 右节点
:param distance: 两个节点的距离
:param id: 用来标记哪些节点是计算过的
:param count: 这个节点的叶子节点个数
"""
self.vec = vec
self.left = left
self.right = right
self.distance = distance
self.id = id
self.count = count
class Hierarchical(object):
def __init__(self, k = 1):
assert k > 0
self.k = k
self.labels = None
def fit(self, x):
nodes = [ClusterNode(vec=v, id=i) for i,v in enumerate(x)]
distances = {}
point_num, future_num = np.shape(x) # 特征的维度
self.labels = [ -1 ] * point_num
currentclustid = -1
while len(nodes) > self.k:
min_dist = math.inf
nodes_len = len(nodes)
closest_part = None # 表示最相似的两个聚类
for i in range(nodes_len - 1):
for j in range(i + 1, nodes_len):
# 为了不重复计算距离,保存在字典内
d_key = (nodes[i].id, nodes[j].id)
if d_key not in distances:
distances[d_key] = euler_distance(nodes[i].vec, nodes[j].vec)
d = distances[d_key]
if d < min_dist:
min_dist = d
closest_part = (i, j)
# 合并两个聚类
part1, part2 = closest_part
node1, node2 = nodes[part1], nodes[part2]
new_vec = [ (node1.vec[i] * node1.count + node2.vec[i] * node2.count ) / (node1.count + node2.count)
for i in range(future_num)] ##??
new_node = ClusterNode(vec=new_vec,
left=node1,
right=node2,
distance=min_dist,
id=currentclustid,
count=node1.count + node2.count)
currentclustid -= 1
del nodes[part2], nodes[part1] # 一定要先del索引较大的
nodes.append(new_node)
self.nodes = nodes
self.calc_label()
def calc_label(self):
"""
调取聚类的结果
"""
for i, node in enumerate(self.nodes):
# 将节点的所有叶子节点都分类
self.leaf_traversal(node, i)
def leaf_traversal(self, node: ClusterNode, label):
"""
递归遍历叶子节点
"""
if node.left == None and node.right == None:
self.labels[node.id] = label
if node.left:
self.leaf_traversal(node.left, label)
if node.right:
self.leaf_traversal(node.right, label)
if __name__ == '__main__':
# iris = load_iris()
# my = Hierarchical(4)
# my.fit(iris.data)
# print(np.array(my.labels))
data = [[16.9,0],[38.5,0],[39.5,0],[80.8,0],[82,0],[834.6,0],[116.1,0]]
my = Hierarchical(4)
my.fit(data)
print(np.array(my.labels))
运行结果如下:
![]()
四、使用Sklearn中的层次聚类
klearn库下的层次聚类是在sklearn.cluster的 AgglomerativeClustering中:
AgglomerativeClustering (
affinity=‘euclidean’,
compute_full_tree=‘auto’,
connectivity=None,
linkage=‘ward’,
memory=None,
n_clusters=2,
pooling_func=
)
AgglomerativeClustering类的构造函数的参数有:n_clusters,linkage,affinity三个重要参数。下面就这三个参数进行描述。
-
n_clusters:(簇的个数)是需要用户指定的,按照常理来说,凝聚层次聚类是不需要指定簇的个数的,但是sklearn的这个类需要指定簇的个数。算法会根据簇的个数判断最终的合并依据,这个参数会影响聚类质量。
-
linkage:(连接方法)指的是衡量簇与簇之间的远近程度的方法。具体说来包括最小距离,最大距离和平均距离三种方式。对应于簇融合的方法,即簇间观测点之间的最小距离作为簇的距离,簇间观测点之间的最大距离作为簇的距离,以及簇间观测点之间的平均距离作为簇的距离。一般说来,平均距离是一种折中的方法。
-
affinity:(连接度量选项)是一个簇间距离的计算方法,包括各种欧式空间的距离计算方法以及非欧式空间的距离计算方法。此外,该参数还可以设置为precomputed,即用户输入计算好的距离矩阵。距离矩阵的生成方法:假设用户有n个观测点,那么先依次构造这n个点两两间的距离列表,即长度为n*(n-1)/2的距离列表,然后通过scipy.spatial.distance的dist库的squareform函数就可以构造距离矩阵了。这种方式的好处是用户可以使用自己定义的方法计算任意两个观测点的距离,然后再进行聚类。 。
if __name__ == '__main__':
data = [[16.9,0],[38.5,0],[39.5,0],[80.8,0],[82,0],[834.6,0],[116.1,0]]
from sklearn.cluster import AgglomerativeClustering
clustering = AgglomerativeClustering(n_clusters=4).fit(data)
print(clustering.labels_)
print(clustering.children_)
打印出的clustering.labels_为:
![]()
打印出的 clustering.children_为:
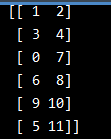
简单解释下:
- data一共有7个样本,那么在进行层次聚类是,这5个样本各自一类,类别名称是0、1、2、3、4、5、6
- 第一行:[1, 2]意思是类别1和类别2距离最近,首先聚成一类,并自动定义类别为7(=7-1+1)
- 第二行:[3, 4]意思是类别3和类别4距离最近,3、4聚成一类,类别为8(=7-1+2)
- 第三行:[0, 7]意思是类别0、类别7距离最近,聚成一类,类别为9(=7-1+3)
。。。。。。
五、使用Scipy库中的层次聚类
linkage方法用于计算两个聚类簇s和t之间的距离d(s,t),这个方法的使用在层次聚类之前。当s和t形成一个新的聚类簇u时,s和t从森林(已经形成的聚类簇群)中移除,而用新的聚类簇u来代替。当森林中只有一个聚类簇时算法停止。而这个聚类簇就成了聚类树的根。 距离矩阵在每次迭代中都将被保存,d[i,j]对应于第i个聚类簇与第j个聚类簇之间的距离。每次迭代必须更新新形成的聚类簇之间的距离矩阵。
(1). linkage(y, method=’single’, metric=’euclidean’)
共包含3个参数:
- y:是距离矩阵,可以是1维压缩向量(距离向量),也可以是2维观测向量(坐标矩阵)。若y是1维压缩向量,则y必须是n个初始观测值的组合,n是坐标矩阵中成对的观测值。
- method:是指计算类间距离的方法,比较常用的有3种:
- (1)single:最近邻,把类与类间距离最近的作为类间距
- (2)complete:最远邻,把类与类间距离最远的作为类间距
- (3)average:平均距离,类与类间所有pairs距离的平均 其他的method还有如weighted,centroid等等
返回值: (n-1)*4的矩阵Z(后面会仔细的讲解返回值各个字段的含义)
(2).fcluster(Z, t, criterion=’inconsistent’, depth=2, R=None, monocrit=None)
- Z:是linkage得到的矩阵,记录了层次聚类的层次信息;
- t:是一个聚类的阈值-“The threshold to apply when forming flat clusters”,在实际中,感觉这个阈值的选取还是蛮重要的.
- 其他参数说明可以参考:https://docs.scipy.org/doc/scipy/reference/generated/scipy.cluster.hierarchy.fcluster.html#scipy.cluster.hierarchy.fcluster
###cluster.py
#导入相应的包
import scipy
import scipy.cluster.hierarchy as sch
from scipy.cluster.vq import vq,kmeans,whiten
import numpy as np
import matplotlib.pylab as plt
#生成待聚类的数据点,这里生成了20个点,每个点4维:
data = [[16.9,0],[38.5,0],[39.5,0],[80.8,0],[82,0],[834.6,0],[116.1,0]]
#加一个标签进行区分
A=[]
for i in range(len(data)):
a=chr(i+ord('A'))
A.append(a)
#1. 层次聚类
#生成点与点之间的距离矩阵,这里用的欧氏距离:
disMat = sch.distance.pdist(data,'euclidean')
#进行层次聚类:
Z=sch.linkage(disMat,method='average')
#将层级聚类结果以树状图表示出来并保存为plot_dendrogram.png
fig = plt.figure()
P = sch.dendrogram(Z, labels=A)
plt.show()
print(Z)
- 第一、二列:聚类簇的编号,在初始距离前每个初始值被从0-n-1进行标识,每生成一个新的聚类簇就在此基础上增加一对新的聚类簇进行标识,
- 第三个列表示前两个聚类簇之间的距离;
- 第四个列表示新生成聚类簇所包含的元素的个数。
if __name__ == '__main__':
data = [[16.9,0],[38.5,0],[39.5,0],[80.8,0],[82,0],[834.6,0],[116.1,0]]
import scipy
import scipy.cluster.hierarchy as sch
from scipy.cluster.vq import vq, kmeans, whiten
import matplotlib.pyplot as plt
A = []
for i in range(7):
a = chr(i+ord('A'))
A.append(a)
Z = sch.linkage(data, 'ward')
f = sch.fcluster(Z, t=30, criterion='distance') # 聚类,这里t阈值的选择很重要
print(f) #打印类标签
fig = plt.figure(figsize=(5,3))
dn = sch.dendrogram(Z,labels=A)
plt.show()
当 t=30 时,运行结果
![]()
当 t=10 时, 运行结果如下
![]()
六、层次聚类的优缺点
优点:
1,距离和规则的相似度容易定义,限制少;
2,不需要预先制定聚类数;
3,可以发现类的层次关系;
4,可以聚类成其它形状
缺点:
1,计算复杂度太高;
2,奇异值也能产生很大影响;
3,算法很可能聚类成链状
参考资料:
- 机器学习–聚类系列–层次聚类:https://www.cnblogs.com/zongfa/p/9344769.html
- 聚类算法之层次聚类(Python实现):https://zhuanlan.zhihu.com/p/32438294
- 聚类算法(4)–Hierarchical clustering层次聚类https://blog.csdn.net/Andy_shenzl/article/details/83783469