聚类分析
文章目录
- 一、监督学习和无监督学习
- 二、聚类分析
- 2.1 K-Means
- 2.2 DBSCAN
- 2.3 超大数据量时应该放弃K均值算法
- 2.4 高维数据上无法应用聚类吗?
- 2.5 如何选择聚类分析算法?
- 三、K-Means算法
- 3.1 什么是K-Means算法?
- 3.2 什么是簇内误差平方和?
- 3.3 sklearn中的KMeans
- 3.4 代码实现
- 3.5 聚类算法的模型评估指标
- 1. 为什么不用簇内距离平方和
- 2. 轮廓系数
一、监督学习和无监督学习
监督学习是指从标注数据(训练集)中学习预测模型的机器学习问题。标注数据表示输入输出的对应关系,预测模型对给定的输入产生相应的输出。监督学习的本质是学习输入到输出的映射的统计规律。
无监督学习是指从无标注数据中学习预测模型的机器学习问题。无标注数据是自然得到的数据,预测模型表示数据的类别、转换或者概率。无监督学习的本质是学习数据中的统计哦规律或潜在结构。
二、聚类分析
聚类是数据挖掘和计算的基本任务,是将大量数据集中具有"相似"特征的数据点或样本划分为一个类别。聚类分析的基本思想是"物以类聚,人以群分",因此大量的数据集中必然存在相似的数据样本,基于这个假设就可以将数据区分出来,并发现不同类的特征。
聚类常用于数据探索或挖掘前期,在没有做先验经验的背景下做的探索性分析,也适用于样本量较大情况下的数据预处理工作。例如,针对企业整体的用户特征,在未得到相关知识或经验之前,先根据数据本身特点进行用户分群,然后针对不同群体作进一步分析;对连续数据离散化,便于后续做分类分析应用。
常用的聚类算法分为基于划分、层次、密度、网格、统计学、模型等类型的算法,典型算法包括K均值,DBSCAN、两步聚类、BIRCH、谱聚类等。
聚类分析能解决的问题包括:数据集可以分为几类、每个类别有多少样本量、不同类别中各个变量的强弱关系如何、不同类别的典型特征如何。除了划分类别等,聚类还能用于基于划分的其他应用,例如图片压缩等。
2.1 K-Means
k均值是聚类中最常用的方法之一,它基于点与点距离的相似度来计算最佳类别归属。但K均值在应用之前一定要注意两种数据异常:
- 数据的异常值。数据中的异常值能明显改变不同点之间的距离相似度,并且这种影响是非常显著的。因此基于距离相似度的判别模式下,异常值的处理必不可少。
- 数据的异常量纲。不同的维度和变量之间,如果存在数值规模或量纲的差异,那么在做距离之前需要先将变量归一化或标准化。例如,跳出率的数值分布区间是[0,1],订单金额可能是[0,1000000],而订单数量则是[0,1000]。如果没有归一化或标准化操作,那么相似度将主要受到订单金额的影响。
2.2 DBSCAN
当然,K均值并不是唯一的聚类方法,如果上述两种条件受某些因素的限制无法实现,那么可以选择其他聚类方法。例如DBSCAN,其中文含义是"基于密度的带有噪声的空间聚类"。DBSCAN是一个比较有代表性的基于密度的聚类算法,与基于划分和层次的聚类方法不同,他将簇定义为密度相连的点的最大集合,能够把具有足够高密度的区域划分为簇,并可在噪声的空间数据集中发现任意形状的聚类。
DBSCAN算法的出发点是基于密度寻找被低密度区域分离的高密度空间,以此来实现不用数据样本的聚类。跟K均值相比,它具有以下优点:
- 原始数据集的分布规律没有明显要求,能适应任意数据集分布形状的空间聚类,因此数据集实用性更广,尤其是对非凸装、圆环形等异性簇分布的识别较好
- 无须指定聚类数量,对结果的先验要求不高
- 由于DBSCAN可区分核心对象、边界点和噪音点,因此对噪声的过滤效果好,能有效应对数据噪点。
但是,由于他直接对整个数据集进行操作,且聚类时使用了一个全局性的表征密度的参数,因此也存在几个明显的弱点:
- 对于高维问题,基于Eps(半径)和MinPts(密度)的定义是个很大问题
- 当簇的密度变化太大时,聚类效果较差
- 当结果量增大时,要求较大的内存支持,I/O消耗也很好。
2.3 超大数据量时应该放弃K均值算法
K均值在算法稳定性、效率和准确率上表现非常好,并且在应对大量数据时依然如此。他的算法时间复杂度上界为O(n* k * t),其中n是样本量、k是划分的聚类数,t是迭代次数。当聚类数和迭代次数不变时,K均值的算法消耗时间只跟样本量有关,因此会呈线性增长趋势。
当真正面对海量数据时,使用K均值算法将面临严重的结果延迟,尤其是当K均值被用作实时性或准实时性的数据预处理、分析和建模时,这种瓶颈效应尤为明显。针对K均值的这一问题,很多延伸算法出现了,MiniBatchKMeans就是其中一个典型代表。MiniBatch的好处是计算过程中不必使用所有的数据样本。由于计算样本量少,所以会相应减少运行时间;但另一方面,由于是抽样方法,抽样样本很难完全代表整体样本的全部特征,因此会带来准确度的下降。
2.4 高维数据上无法应用聚类吗?
在大数据背景上,数据获取难度和成本非常低,很多高维数据场景例如电子商务交易数据、Web文本数据日益丰富。在做高维数据聚类时,传统的在低维空间通用的聚类方法运用到高维空间时,通常不能取得令人满意的聚类结果,主要表现在聚类计算耗时太长、聚类结果相对于真是标签分类的准确性和稳定性都大为降低
应对高维数据的聚类主要有2种方法:降维、子空间聚类
降维是应对高维数据的有效方法,通过特征选择法或维度转换法将高维空间降低或映射到低维空间,直接解决了高维问题
子空间聚类算法是在高维数据空间中对传统聚类算法的一种扩展,其思想是选取与给定簇密切相关的维,然后在对应的子空间进行聚类。比如,谱聚类就是一种子空间聚类方法。
2.5 如何选择聚类分析算法?
聚类算法有几十种之多,聚类算法的选择主要参考以下因素:
- 如果数据集是高维的,那么选择谱聚类,他是子空间划分的一种
- 如果数据量为中小规模,例如在100万条以内,那么K均值将是比较好的选择;如果数据量超过100万条,那么可以考虑使用MiniBatchKMeans
- 如果数据集中有噪点,那么使用基于密度的DBSCAN可以有效应对这个问题。
三、K-Means算法
3.1 什么是K-Means算法?
作为聚类算法的典型代表,Kmeans可以说是最简单的聚类算法没有之一,那么他是怎么完成聚类的呢?
KMeans算法将一组N个样本的特征矩阵X划分为K个无交集的簇,直观上来看是簇是一组一组聚集在一起的数据,在一个簇中的数据就认为是同一类。簇就是聚类的结果表现。
簇中所有数据的均值 通常被称为这个簇的“质心”(centroids)。在一个二维平面中,一簇数据点的质心的横坐标就是这一簇数据点的横坐标的均值,质心的纵坐标就是这一簇数据点的纵坐标的均值。同理可推广至高维空间。
在KMeans算法中,簇的个数K是一个超参数,需要我们人为输入来确定。KMeans的核心任务就是根据我们设定好的K,找出K个最优的质心,并将离这些质心最近的数据分别分配到这些质心代表的簇中去。
1. 随机抽取K个样本作为最初的质心
2. 开始循环
3. 将每个样本分配到离他们最近的质心,生成K个簇
4. 对于每个簇,计算所有被分到该簇的样本点的平均值作为新的质心
5. 当质心的位置不再发生变化,迭代停止,聚类完成
3.2 什么是簇内误差平方和?
我们认为聚类算法聚出的类,也就是被分在同一个簇中的数据是具有相似性的,而不同簇中的数据是不同的,当聚类完毕之后,我们就要分别去研究每个簇中的样本都有什么样的性质,从而根据业务需求来制定相应的策略。
因此我们追求“簇内差异小,簇外差异大”。而这个“差异“,由样本点到其所在簇的质心的距离来衡量。
对于一个簇来说,所有样本点到质心的距离之和越小,我们就认为这个簇中的样本越相似,簇内差异就越小。而距离的衡量方法有很多种,令x表示簇中的一个样本点,μ表示该簇中的质心,n表示每个样本点中的特征数目,i表示组成点x的每个特征,则该样本点到质心的距离可以由以下距离来度量:
欧 几 里 得 距 离 : d ( x , μ ) = ∑ i = 1 n ( x i − μ i ) 2 ) 欧几里得距离:d(x,μ) = \sqrt{\sum_{i=1}^n(x_i-μ_i)^2)} 欧几里得距离:d(x,μ)=i=1∑n(xi−μi)2)
曼 哈 顿 距 离 : d ( x , μ ) = ∑ i = 1 n ( ∣ x i − μ ∣ ) 曼哈顿距离:d(x,μ) ={\sum_{i=1}^n(|x_i-μ|)} 曼哈顿距离:d(x,μ)=i=1∑n(∣xi−μ∣)
如我们采用欧几里得距离,则一个簇中所有样本点到质心的距离的平方和为:
C l u s t e r S u m o f S q u a r e ( C S S ) = ∑ j = 0 m ∑ i = 1 n ( x i − μ i ) 2 ClusterSumof Square(CSS)=\sum_{j=0}^m\sum_{i=1}^n(x_i-μ_i)^2 ClusterSumofSquare(CSS)=j=0∑mi=1∑n(xi−μi)2
T o t a l C l u s t e r S u m o f S q u a r e = ∑ l = 1 k C S S i Total Cluster Sum of Square=\sum_{l=1}^kCSS_i TotalClusterSumofSquare=l=1∑kCSSi
其中,m为一个簇中样本的个数,j是每个样本的编号。这个公式被称为簇内平方和,。而将一个数据集中的所有簇的簇内平方和相加,就得到了整体平方和。总体平方和越小,代表着每个簇内样本月相似,聚类的效果就越好。因此
KMeans追求的是,求解能够让Inertia最小化的质心。实际上,在质心不断变化不断迭代的过程中,总体平方和是越来越小的。我们可以使用数学来证明,当整体平方和最小的时候,质心就不再发生变化了。如此,K-Means的求解过程,就变成了一个最优化问题。
3.3 sklearn中的KMeans
class sklearn.cluster.KMeans(n_clusters=8, init=‘k-means++’, n_init=10, max_iter=300, tol=0.0001, precompute_distances=‘auto’, verbose=0, random_state=None, copy_x=True, n_jobs=1)
| 参数 | 含义 |
|---|---|
| n_clusters | 整数,可不填,默认为8 |
| init | 可输入“k-means++”,“random”或者一个n维数组 初始化质心的方法,默认为“k-means++”如果输入n维数组,数组的形状应该是(n_clusters,b_features)并给出初始质心 |
| n_init | 整数,默认10 使用不同的质心随机初始化的种子来运行kmeans算法的次数。最终结果会是基于inertia来计算的n_init次连续运行后的最佳输出 |
| max_iter | 整数,默认300 单次运行的k-means算法的最大迭代次数 |
| tol | 浮点数,默认1e-4 两次迭代间Inertia下降的量,如果两次迭代之间Inertia下降的值小于tol所设定的值,迭代就会停下 |
| precompute_distances | ‘auto’,True,False 默认为‘auto’ 预计算距离(更快但需要更多内存) ‘auto’:如果n_samples* n_clusters > 1200万,不要预先计算距离 True:始终预先计算距离 False:从不预先计算距离 |
| random_state | 整数,随机数或者None,默认None 确定质心初始化的随机数生成。使用int可以使随机性具有确定性 |
3.4 代码实现
引库
[1]:import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
创建数据集
[2]:X,y = make_blobs(n_samples=500,n_features=2,centers=4,random_state=1)
fig,ax1 = plt.subplots(1)
ax1.scatter(X[:,0],X[:,1]
,marker='o'
,s=8
)
plt.show()
训练模型
[3]:clusters=4
model_kmeans = KMeans(n_clusters=clusters,random_state=1)
cluster = model_kmeans.fit(X)
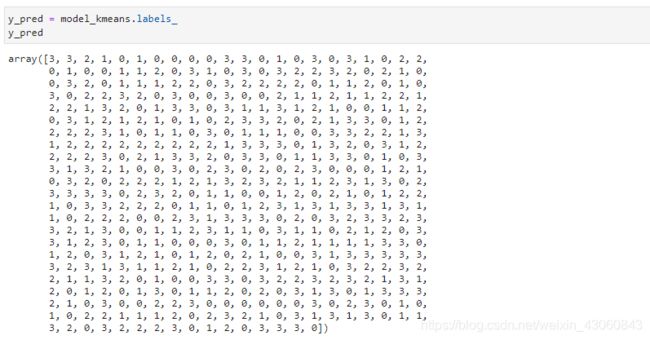
[4]:y_pred = model_kmeans.labels_
y_pred
[5]:centroid = cluster.cluster_centers_
centroid
array([[ -6.08459039, -3.17305983],
[ -1.54234022, 4.43517599],
[-10.00969056, -3.84944007],
[ -7.09306648, -8.10994454]])
可视化
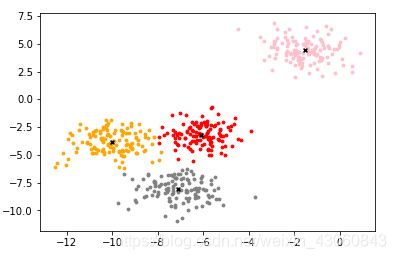
[6]:color = ["red","pink","orange","gray"]
fig, ax1 = plt.subplots(1)
for i in range(clusters):
ax1.scatter(X[y_pred==i, 0], X[y_pred==i, 1]
,marker='o'
,s=8
,c=color[i]
)
ax1.scatter(centroid[:,0],centroid[:,1]
,marker="x"
,s=15
,c="black")
plt.show()
3.5 聚类算法的模型评估指标
不同于分类模型和回归,聚类算法的模型评估不是一件简单的事。在分类中,有直接结果(标签)的输出,并且分类的结果有正误之分,所以我们使用预测的准确度,混淆矩阵,ROC曲线等等指标来进行评估,但无论如何评估,都是在”模型找到正确答案“的能力。而回归中,由于要拟合数据,我们有SSE均方误差,有损失函数来衡量模型的拟合程度。但这些衡量指标都不能够使用于聚类。
KMeans的目标是确保“簇内差异小,簇外差异大”,我们就可以通过衡量簇内差异来衡量聚类的效果。我们刚才说过,Inertia是用距离来衡量簇内差异的指标,因此,我们是否可以使用Inertia来作为聚类的衡量指标呢?
1. 为什么不用簇内距离平方和
KMeans的目标是确保“簇内差异小,簇外差异大”,我们就可以通过衡量簇内差异来衡量聚类的效果。我们刚才说过,Inertia是用距离来衡量簇内差异的指标,因此,我们是否可以使用Inertia来作为聚类的衡量指标呢?Inertia越小模型越好嘛。
可以,但是这个指标的缺点和极限太大
首先,它不是有界的。我们只知道,Inertia是越小越好,是0最好,但我们不知道,一个较小的Inertia究竟有没有达到模型的极限,能否继续提高。
[7]:n_clusters = 5
cluster_ = KMeans(n_clusters=n_clusters, random_state=0).fit(X)
inertia_ = cluster_.inertia_
inertia_
811.0952123653016
[8]:n_clusters = 6
cluster_ = KMeans(n_clusters=n_clusters, random_state=0).fit(X)
inertia_ = cluster_.inertia_
inertia_
728.2827697678249
[8]:n_clusters = 10
cluster_ = KMeans(n_clusters=n_clusters, random_state=0).fit(X)
inertia_ = cluster_.inertia_
inertia_
475.04707738309605
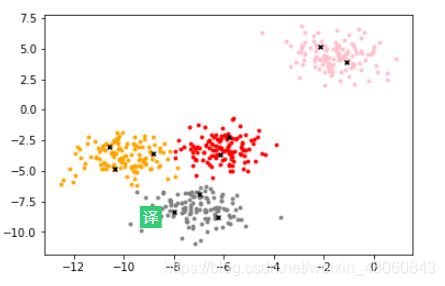
[9]:centroid = cluster_.cluster_centers_
color = ["red","pink","orange","gray","red","pink","orange","gray","red","pink"]
fig, ax1 = plt.subplots(1)
for i in range(n_clusters):
ax1.scatter(X[y_pred==i, 0], X[y_pred==i, 1]
,marker='o'
,s=8
,c=color[i]
)
ax1.scatter(centroid[:,0],centroid[:,1]
,marker="x"
,s=15
,c="black")
plt.show()
第二,它的计算太容易受到特征数目的影响,数据维度很大的时候,Inertia的计算量会陷入维度诅咒之中,计算量会爆炸,不适合用来一次次评估模型。
第三,它会受到超参数K的影响,在我们之前的常识中其实我们已经发现,随着K越大,Inertia注定会越来越小,但这并不代表模型的效果越来越好了
第四,Inertia对数据的分布有假设,它假设数据满足凸分布(即数据在二维平面图像上看起来是一个凸函数的样子),并且它假设数据是各向同性的(isotropic),即是说数据的属性在不同方向上代表着相同的含义。但是现实中的数据往往不是这样。所以使用Inertia作为评估指标,会让聚类算法在一些细长簇,环形簇,或者不规则形状的流形时表现不佳
2. 轮廓系数
在99%的情况下,我们是对没有真实标签的数据进行探索,也就是对不知道真正答案的数据进行聚类。这样的聚类,是完全依赖于评价簇内的稠密程度(簇内差异小)和簇间的离散程度(簇外差异大)来评估聚类的效果。其中轮廓系数是最常用的聚类算法的评价指标。它是对每个样本来定义的,它能够同时衡量:
-
样本与其自身所在的簇中的其他样本的相似度a,等于样本与同一簇中所有其他点之间的平均距离
-
样本与其他簇中的样本的相似度b,等于样本与下一个最近的簇中的所有点之间的平均距离
根据聚类的要求”簇内差异小,簇外差异大“,我们希望b永远大于a,并且大得越多越好。
单个样本的轮廓系数计算为:
s = b − a m a x ( a , b ) s = {b-a \over max(a,b)} s=max(a,b)b−a
很容易理解轮廓系数范围是(-1,1),其中值越接近1表示样本与自己所在的簇中的样本很相似,并且与其他簇中的样本不相似,当样本点与簇外的样本更相似的时候,轮廓系数就为负。当轮廓系数为0时,则代表两个簇中的样本相似度一致,两个簇本应该是一个簇。可以总结为轮廓系数越接近于1越好,负数则表示聚类效果非常差。
如果一个簇中的大多数样本具有比较高的轮廓系数,则簇会有较高的总轮廓系数,则整个数据集的平均轮廓系数越高,则聚类是合适的。如果许多样本点具有低轮廓系数甚至负值,则聚类是不合适的,聚类的超参数K可能设定得太大或者太小。
在sklearn中,我们使用模块metrics中的类silhouette_score来计算轮廓系数,它返回的是一个数据集中,所有样本的轮廓系数的均值。但我们还有同在metrics模块中的silhouette_sample,它的参数与轮廓系数一致,但返回的
是数据集中每个样本自己的轮廓系数。
我们来看看轮廓系数在我们自建的数据集上表现如何:
[10]:from sklearn.metrics import silhouette_score
from sklearn.metrics import silhouette_samples
[11]:silhouette_score(X,y_pred)
0.6505186632729437
[12]:silhouette_score(X,cluster_.labels_)
0.3356887273833359
轮廓系数有很多优点,它在有限空间中取值,使得我们对模型的聚类效果有一个“参考”。并且,轮廓系数对数据的 分布没有假设,因此在很多数据集上都表现良好。但它在每个簇的分割比较清洗时表现好。但轮廓系数也有缺 陷,它在凸型的类上表现会虚高,比如基于密度进行的聚类,或通过DBSCAN获得的聚类结果,如果使用轮廓系数 来衡量,则会表现出比真实聚类效果更高的分数。
参考资料:机器学习实战
菜菜的sklearn课堂