图像处理基本库的学习笔记5--公共数据集,PASCAL VOC数据集,NYUD V2数据集的简介与提取,COCO2017,医学影像数据集汇总
目录
- 公共数据集
- 计算机视觉标准数据集整理—PASCAL VOC数据集
- 数据集文件结构
- Annotation
- JPEGImages
- SegmentationClass
- SegmentationObject
- NYUD V2数据集的简介与提取
- COCO2017
- 医学影像数据集汇总
- 左心房图像分割集
公共数据集
《基于全卷积神经网络的图像分割算法的研究及应用》
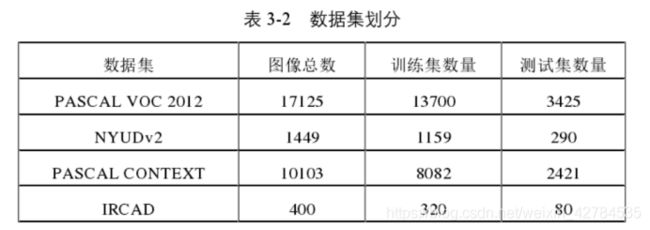
实验数据:本章节的公共数据集来源自计算机视觉领域的 PASCAL VOC 2012、NYUDv2、PASCAL CONTEXT,医学数据集来自法国 IRCAD 国际医疗中心数据库中的肝脏 CT 扫描图像。
本节按照 4:1 的比例将两个数据集分别划分成训练和测试两部分。在算法中,就会使用训练那部分完成算法网络模型的构建,在训练的过程中,为参数赋予不同值从而获得不同的结果,之后利用测试集对网络模型的结果进行记录和分析。
计算机视觉标准数据集整理—PASCAL VOC数据集
参考文章:https://blog.csdn.net/xingwei_09/article/details/79142558
PASCAL VOC为图像识别和分类提供了一整套标准化的优秀的数据集,从2005年到2012年每年都会举行一场图像识别challenge。此数据集可以用于图像分类、目标检测、图像分割。
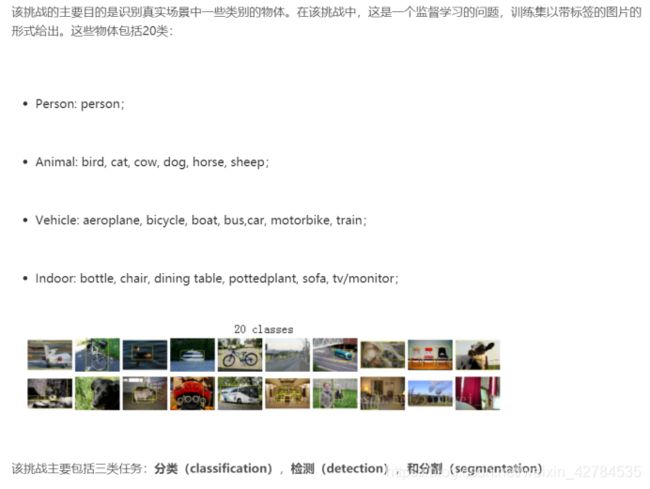
训练集由一套图像组成:每个图像拥有一个对应的标注文件,给出了图像中出现的物体的bounding box和class label,该物体属于上述20类中的某一类。同一张图像中,可能出现属于多个类别的多个物体。
所有的标注图片都有Detection需要的label,但只有部分数据有Segmentation Label。
VOC2007中包含9963张标注过的图片, 由train/val/test三部分组成, 共标注出24,640个物体。VOC2007的test数据label已经公布, 之后的没有公布(只有图片,没有label)。
对于检测任务,VOC2012的trainval/test包含08-11年的所有对应图片。 trainval有11540张图片共27450个物体。
对于分割任务, VOC2012的trainval包含07-11年的所有对应图片, test只包含08-11。trainval有 2913张图片共6929个物体。
这些图像中的一部分图像还拥有像素级的标注,用于segmentationcompetition.
用于action classification的图片集与用于classification/detection/segmentation的图片集不相交。它们被部分地标注上了图像中人的bounding box,参考点和动作。
用于person layout taster的图像,被额外的标注上了人的身体部位(头、手、脚),其测试集与主任务(classification/detection)的测试集不相交。
数据集按1:1的比例被分为训练(验证)集和测试集。这两部分的图像中类别的分布也大致相等。
数据集文件结构
Annotation
Annotations文件夹中存放的是xml格式的标签文件,每一个xml文件都对应于JPEGImages文件夹中的一张图片。
 Action是下存放的是人的动作(running、jumping等等,这也是VOC challenge的一部分)
Action是下存放的是人的动作(running、jumping等等,这也是VOC challenge的一部分)
Layout下存放的是人体部位(head、hand、feet等等,这也是VOC challenge的一部分)
Main下存放的是图像物体识别的数据,总共分为20类
Segmentation下存放的是可用于分割的图片的编号
JPEGImages
JPEGImages文件夹中包含了PASCAL VOC所提供的所有的图片,包含训练图片和测试图片,共有17125张。图片均以“年份_编号.jpg”格式命名。图片的尺寸大小不一,所以在后面训练的时候需要对图片进行resize操作。
图片的像素尺寸大小不一,但是横向图的尺寸大约在500375左右,纵向图的尺寸大约在375500左右,基本不会偏差超过100。(在之后的训练中,第一步就是将这些图片都resize到300300或是500500,所有原始图片不能离这个标准过远,具体resize方式可以看最下面github链接里data.py。)
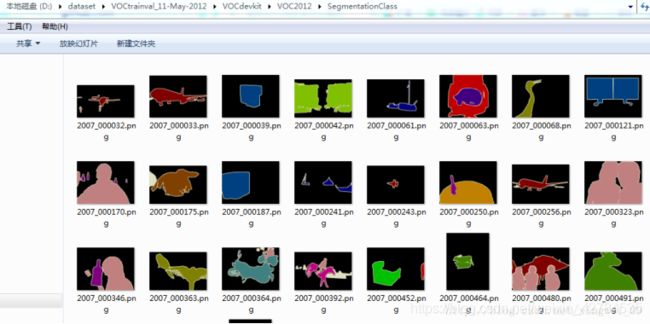
SegmentationClass
这里面包含了2913张图片,每一张图片都对应JPEGImages里面的相应编号的图片。图片的像素颜色共有20种,对应20类物体。
SegmentationObject
这里面同样包含了2913张图片,图片编号都与Class里面的图片编号相同。这里面的图片和Class里面图片的区别在于,这是针对Object的。
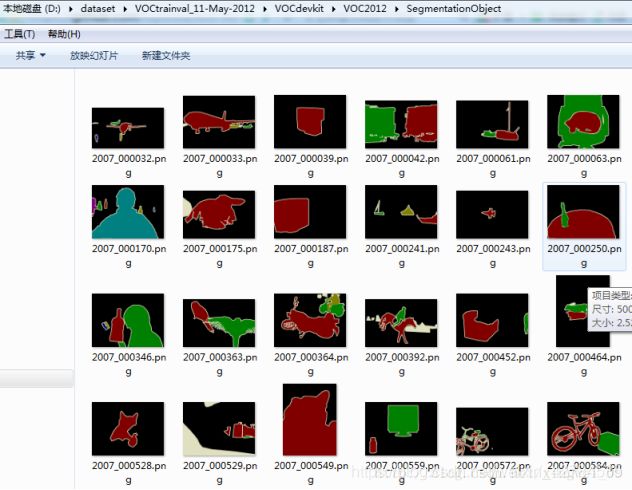
在Class里面,一张图片里如果有多架飞机,那么会全部标注为红色。而在Object里面,同一张图片里面的飞机会被不同颜色标注出来。
NYUD V2数据集的简介与提取
参考文章:https://blog.csdn.net/weixin_43915709/article/details/88774325
数据集:https://github.com/ankurhanda/nyuv2-meta-data
使用NYUDv2数据集进行语义分割的时候会发现,从官网直接下载的数据集有894类,而发现在许多论文中描述的是40类,有的也称作nyudv2-40;一些研究中也出现了13类的标签。
这里给出了40分类的label数据集:
labels40.mat,可直接进行提取# 从mat文件提取labels# 需要注意这个文件里面的格式和官方有所不同,长宽需要互换,也就是进行转置
import cv2
import scipy.io as scio
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
import os
dataFile = './labels40.mat'
data =
scio.loadmat(dataFile)
labels=np.array(data["labels40"])
path_converted='./nyu_labels40'
if not os.path.isdir(path_converted): os.makedirs(path_converted)
labels_number=[]
for i in range(1449):
labels_number.append(labels[:,:,i].transpose((1, 0))) # 转置
labels_0=np.array(labels_number[i])
#print labels_0.shape
print (type(labels_0))
label_img=Image.fromarray(np.uint8(labels_number[i]))
#label_img = label_img.rotate(270)
label_img = label_img.transpose(Image.ROTATE_270)
iconpath='./nyu_labels40/'+str('%06d'%(i+1))+'.png'
label_img.save(iconpath, optimize=True)
COCO2017
参考文章:https://blog.csdn.net/qq_41847324/article/details/86224628
COCO数据集可以说是语义分割等计算机视觉任务中应用较为广泛的一个数据集,具体可以应用到物体识别、语义分割及目标检测等方面。
国内下载地址:http://fun001.f3322.net:666/share/coco2017/
COCO数据集介绍
网上关于COCO数据集的介绍多如牛毛,本文就不过多的加以介绍了,简要的介绍以下。
以COCO2014为例:
下载完COCO2014后进行解压后,目录如下:
images
train2014
val2014
test2014
annotations
其中,images中的文件夹各自放置了训练、验证和测试的数据集图片。annotations文件夹中放置了标签文件,可以理解为Label,简要的来说,就是包含了某一类在图片中的具体位置的信息,
详细可见以下链接:https://blog.csdn.net/happyhorizion/article/details/77894205#semantic-scene-labeling图像分割
三、COCO数据集使用(数据载入)
所需环境为:
numpy
torch
tqdm(可视化数据载入)
os
pycocotools(coco数据集的应用API)
torchvision
PIL
医学影像数据集汇总
本文转自:https://blog.csdn.net/sinat_37842336/article/details/80582948
1、肺结节数据库LIDC-IDRI:
CSDN数据库介绍:http://blog.csdn.net/dcxhun3/article/details/54289598
数据库网址:https://wiki.cancerimagingarchive.net/display/Public/LIDC-IDRI
2、乳腺图像数据库DDSM MIAS
数据库介绍(DDSM SQL Data Base):http://deckard.mc.duke.edu/ddsm_sql/book1.html
图片格式为LJPEG需要使用对应的压缩方法对其进行解压,目前找到了xMedcon,但是不太会用,打不开相应的文件,也可能是不是使用这个软件压缩的。不过这个软件可以用来转换大部分的医学图像。matlab社区上有how to open lossless jpeg file,但是其中有一些答案提供的网址不能打开,可能是没有科学上网??
数据库网址:http://figment.csee.usf.edu/Mammography/Database.html
MIAS MiniMammographic Database(来自researchgate的一个问答):322例,尺寸:1024*1024pixel,图像数据是PGM格式,找到一个介绍和读取的博客代码使用的c,matlab问答相关
小型乳房X光数据库:http://peipa.essex.ac.uk/pix/mias/all-mias.tar.gz
这也是一个乳腺的图像数据库,但是现在还没有搞清楚下载、格式之类的:https://www.repository.cam.ac.uk/handle/1810/250394?show=full
3、医学图像问答(目前还没搞清楚干嘛的,好像是一个网站的问答。暂存)
网址:http://www.dclunie.com/medical-image-faq/html/index.html
4、左心室MRI图像Cardiac MRI Dataset: http://www.cse.yorku.ca/~mridataset/
右心室MRI数据RVSC
右心室分割挑战赛(2012):http://pagesperso.litislab.fr/cpetitjean/mr-images-and-contour-data/
5、Kaggle比赛网址:https://www.kaggle.com/
CT Medical Image Analysis Turorial这个比赛好像是分析CT纹理与患者年龄的关系。
肺癌分类比赛:https://www.kaggle.com/c/data-science-bowl-2017/data
分割肺癌(Kaggle):https://www.kaggle.com/kmader/finding-lungs-in-ct-dataDICOM文件打开使用Sante
DICOM Free,paraview也可以打开,Mango网站:https://idoimaging.com/programs/124;anteDicom
官方下载网址:http://www.santesoft.com/win/sante-dicom-viewer-free/download.html
6、Cancer Imaging Archive这个网站可以获得一些癌症的数据库,下载下来是jnpl文件需要使用jre环境进行下载:http://www.cancerimagingarchive.net/
7、OsiriX数据库:各种医学数据,好像得注册收费的样子,还没搞清楚http://www.osirix-viewer.com/resources/dicom-image-library/
8.Github上哈佛 beamandrew机器学习和医学影像研究者-贡献的数据集https://github.com/beamandrew/medical-data
9.ISBI(生物医学成像国际研讨会)https://grand-challenge.org/All_Challenges/
10.NITRC的IBSR数据集
左心房图像分割集
心脏MRI数据集该网页包含短轴心脏MR图像数据集及其左心室心内膜和心外膜分割的基本事实。
该数据集首先被编译并用作以下论文的一部分:Alexander Andreopoulos,John K. Tsotsos,用于分析心脏MRI的形状和外观的高效和一般化统计模型,医学图像分析,第12卷,第3期,2008年6月,第335-357页。
PDF使用是免费的; 我们恭敬地问,如果您使用此数据集,则引用上述文章作为其来源。作者要感谢多伦多儿童医院心脏放射科医师Paul Babyn博士和心脏放射学家Shi-Joon Yoo博士的数据集以及他们对该研究项目的帮助。
免责声明:数据集仅供研究之用,约克大学和参与数据集生成的研究人员均未提供担保或担保。
从33个科目获得的心脏MR图像。每个主题的序列由沿长轴的20帧和8-15个切片组成,**总共7980个图像。对应于每个主题x的序列位于名为sol_yxzt_patx.mat的不同的.mat(MATLAB)文件中。**这些是未经处理的原始图像,最初存储为16位DICOM图像。下载上述序列的分段。
我们手动分割了7980个图像中的每一个,其中左心室的心内膜和心外膜都是可见的,总共5011个分割的MR图像和10022个轮廓。对应于每个主题x的分段位于名为manual_seg_32points_patx.mat的不同的.mat(MATLAB)文件中。每个轮廓由像素坐标中给出的32个点描述。
两个小的MATLAB函数,用于可视化相应图像上的分割。
请参阅随附的README文件以获取其使用示例。下载包含像素间距(每像素mm)的元数据,每个受试者序列的长轴(每片的mm)之间的间距,每个受试者的年龄和诊断。