Dicom 医学图像与 nii 标签数据处理
Dicom 医学图像与 nii 标签数据处理
- Ⅰ. Dicom医学图像处理
- Ⅱ. nii标记数据处理
- Ⅲ. 综合数据处理
-
- 1. code1
- 2. 需要说明
- 3. code2
Ⅰ. Dicom医学图像处理
reference: https://blog.csdn.net/u011764992/article/details/84501300
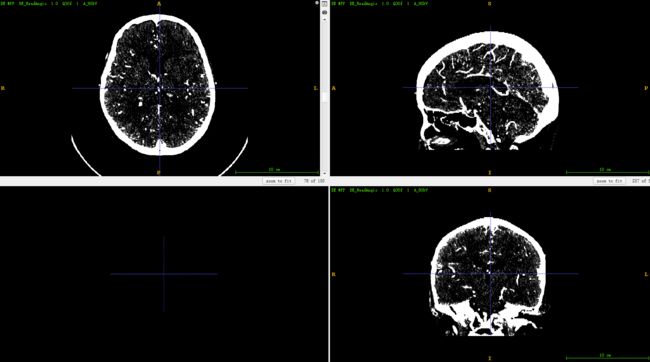
dicom 图像就长这样
dicom图像的源数据范围相当大,而转化为图像形式保存的数据则为uint8类型,直接转化会有损失,本文将其转为.npy形式。
Ⅱ. nii标记数据处理
reference: https://blog.csdn.net/weixin_43330946/article/details/89576759
这里有以前写的ITK-SNAP使用技巧: https://www.jianshu.com/p/79ac3ce55a93
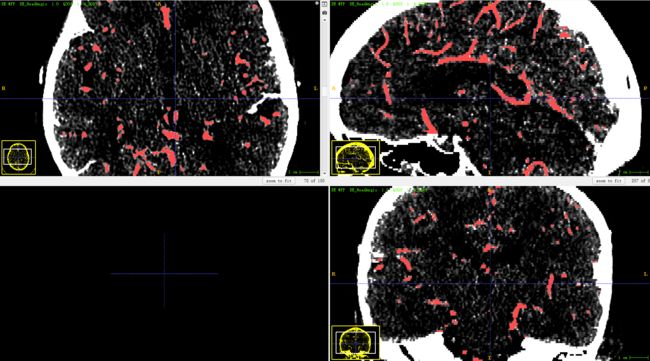
使用ITK-SNAP对dicom 图像进行标记
同样转化为.npy格式
Ⅲ. 综合数据处理
1. code1
使用本代码是在dicom数据转化为.npy格式,而.nii的标记数据还未进行转化的情况下
import numpy as np
import os #遍历文件夹
import nibabel as nib #nii格式一般都会用到这个包
import imageio #转换成图像
from matplotlib import pyplot as plt
import cv2
def nii_to_image(niifile):
filenames = os.listdir(filepath) #读取nii文件夹
slice_trans = []
for f in filenames:
#开始读取nii文件
img_path = os.path.join(filepath, f)
img = nib.load(img_path) #读取nii
img_fdata = img.get_fdata()
fname = f.replace('.nii','') #去掉nii的后缀名
#开始转换为图像
(x,y,z) = img.shape
for i in range(0,z):
silce = img_fdata[:, :, i]
slice_trans.append(silce)
return slice_trans
def get_image_file(file_dir):
image_data = []
files = os.listdir(file_dir)
files.sort(key= lambda x:int(x[:-4]))
for file in files:
if os.path.splitext(file)[1] == '.npy':
img_item = np.load(file_dir + file)
image_data.append(img_item)
return image_data
if __name__ == '__main__':
filepath = './imgData/seg12190000'
label = nii_to_image(filepath)
file_dir = './Documents/dicom2npy_75989854/'
image_data = get_image_file(file_dir)
imgfile = './imgData/label/'
for i in range(len(image_data)):
filename_item = imgfile + str(i + 1)
img_item = image_data[i]
# 简单对 img 进行处理
img_item[img_item < 0] = 0
img_item[img_item > 512] = 512
# img_item.dtype = np.uint8
img_item = img_item.astype(np.uint8)
# 不知道为什么只有这样才能整出来
label_item = label[149-i]
label_item = np.rot90(label_item, -1)
label_item = np.flip(label_item, 1)
# 图片做成三通道查看一下效果
img_item = cv2.merge([img_item, img_item, img_item])
img_item[label_item > 0] = img_item[label_item > 0] * 0.6 + (80, 0, 0)
# imageio.imwrite(os.path.join(imgfile, '{}.png'.format(i)), img_item)
# imageio.imwrite(os.path.join(imgfile, '{}.png'.format(i)), label_item)
# 保存调试好的label, 反序 + 左右翻转 + 旋转-1*90
np.save(os.path.join(imgfile, '{}.npy'.format(i)), label_item)
print(i)
2. 需要说明
不知道为什么,采用上述博客的做法即对提取出的标记数据以silce = img_fdata[:, :, i]直接进行切片效果对不上。在几番尝试后摸索出了这样的逻辑:
label_item = label[149-i]
label_item = np.rot90(label_item, -1)
label_item = np.flip(label_item, 1)
真是惊呆了,倒序+旋转+左右翻转?

不过总而言之这样就匹配上了
3. code2
针对前面已经提取好的.npy格式的源数据,编写了读入训练数据的类如下:
import os
import numpy as np
import matplotlib.pyplot as plt
import random
import cv2
class trainData():
def __init__(self):
''' 图像数据与标记数据路径 s1: 各为 150张 '''
self.image_dir = './imgData/train_data/s1/image/'
self.label_dir = './imgData/train_data/s1/label/'
self.image_data = None
self.label_data = None
def get_image_file(self):
''' 图像数据为 512 * 512, 为源数据值, 未作改动(范围在: -500左右 ~ 1000+ ) '''
file_dir = self.image_dir
image_data = []
files = os.listdir(file_dir)
files.sort(key= lambda x:int(x[:-4]))
for file in files:
if os.path.splitext(file)[1] == '.npy':
img_item = np.load(file_dir + file)
image_data.append(img_item)
self.image_data = image_data
return image_data
def get_label_file(self):
''' 标签数据为 512 * 512, 为0/1 '''
file_dir = self.label_dir
label_data = []
files = os.listdir(file_dir)
files.sort(key= lambda x:int(x[:-4]))
for file in files:
if os.path.splitext(file)[1] == '.npy':
img_item = np.load(file_dir + file)
label_data.append(img_item)
self.label_data = label_data
return label_data
def test_show(self):
''' 随机取九张展示 '''
if self.image_data == None or self.label_data == None:
self.get_image_file()
self.get_label_file()
_, figs = plt.subplots(3, 3)
for i in range(3):
for j in range(3):
index = random.randint(0,len(self.image_data)-1)
img_item = self.image_data[index]
label_item = self.label_data[index]
# 标签处简单处理,显示浅红色
img_item[img_item < 0] = 0
img_item[img_item > 255] = 255
img_item = img_item.astype(np.uint8)
img_item = cv2.merge([img_item, img_item, img_item])
img_item[label_item > 0] = img_item[label_item > 0] * 0.6 + (80, 0, 0)
figs[i][j].imshow(img_item)
plt.show()
def get_train_data(self):
'''
TODO:先试试使用未经处理的图像数据, 返回对象为包含np矩阵的列表
'''
self.get_image_file()
self.get_label_file()
return self.image_data, self.label_data
if __name__ == "__main__":
# 处理源图像与标记数据
trainDataLoader = trainData()
trainDataLoader.test_show()
x,y = trainDataLoader.get_train_data()
看看随机展示九张图的效果:
